Консервативные сигнатурные вставки и делеции ( CSI ) в белковых последовательностях представляют собой важную категорию молекулярных маркеров для понимания филогенетических взаимоотношений. [1] [2] CSI, вызванные редкими генетическими изменениями, предоставляют полезные филогенетические маркеры, которые обычно имеют определенный размер и с обеих сторон окружены консервативными областями для обеспечения их надежности. Хотя инделы могут представлять собой произвольные вставки или делеции, CSI определяются только как те белковые инделы, которые присутствуют в консервативных областях белка. [2] [3] [4] [5]
CSI, которые ограничены определенной кладой или группой видов, обычно обеспечивают хорошие филогенетические маркеры общего эволюционного происхождения. [2] Из-за редкости и весьма специфической природы таких изменений маловероятно, что они могут возникнуть независимо в результате конвергентной или параллельной эволюции (т.е. гомоплазии) и, следовательно, скорее всего, представляют собой синапоморфию . Другие мешающие факторы, такие как различия в темпах эволюции на разных участках или среди разных видов, также обычно не влияют на интерпретацию CSI. [2] [3] Определяя наличие или отсутствие CSI у видов, не входящих в группу, можно сделать вывод, была ли предковая форма CSI вставкой или делецией, и это можно использовать для установления укоренившихся филогенетических отношений между организмами. [1] [2]
CSI обнаруживаются путем поиска общих изменений в филогенетическом дереве, построенном на основе белковых последовательностей. Было обнаружено, что большинство идентифицированных CSI имеют высокую прогностическую ценность при добавлении новых последовательностей, сохраняя специфичность для первоначально идентифицированных клад видов. С их помощью можно идентифицировать как известные, так и даже ранее неизвестные виды, принадлежащие к этим группам, в различных средах обитания. [3] По сравнению с порядками ветвления деревьев, которые могут различаться в зависимости от метода, конкретные CSI создают более конкретные ограничения , которые дешевле применять с вычислительной точки зрения. [6]

Группоспецифичные CSI обычно являются общими для разных видов, принадлежащих к определенному таксону (например, роду, семейству, классу, отряду, типу), но они не присутствуют в других группах. Эти CSI, скорее всего, были введены у предка группы видов до того, как члены таксонов разошлись. Они предоставляют молекулярные средства для отличия представителей определенного таксона от всех других организмов. [2] [5]
На рис. 1 показан пример 5аа CSI, обнаруженного у всех видов, принадлежащих к таксону X. Это отличительная характеристика этого таксона, поскольку он не встречается ни у одного другого вида. Эта подпись, вероятно, была передана общему предку видов из этого таксона. Аналогично, другие сигнатуры, специфичные для группы (не показаны), могут быть общими либо для A1 и A2, либо для B1 и B2 и т. д., либо даже для X1 и X2, либо для X3 и X4 и т. д. Группы A, B, C, D и X на этой диаграмме может соответствовать различным типам бактерий или эукариот . [7]
Группоспецифические CSI использовались в прошлом для определения филогенетических взаимоотношений ряда типов бактерий и подгрупп внутри них. Например, вставка из 3 аминокислот была уникально распространена среди представителей типа Thermotogota (ранее Thermotogae) в незаменимом 50S рибосомальном белке L7/L12 в высококонсервативной области (82-124 аминокислоты). Он отсутствует ни у каких других видов бактерий и может быть использован для характеристики представителей Thermotogota от всех других бактерий. Групповые CSI также использовались для характеристики подгрупп внутри Thermotogota. [8]

CSI основной линии — это те, в которых консервативная вставка или делеция являются общими для нескольких основных типов, но отсутствуют в других типах. [2]
На рисунке 2 показан пример 5aa CSI, обнаруженного в консервативной области, которая обычно присутствует у видов, принадлежащих к типам X, Y и Z, но отсутствует у других типов (A, B и C). Эта сигнатура указывает на специфическое родство таксонов X, Y и Z, а также A, B и C. На основании наличия или отсутствия такой индели у видов чужой группы (а именно архей) можно сделать вывод, является ли индел является вставкой или делецией, и какая из этих двух групп A, B, C или X, Y, Z является предковой. [7]
В прошлом основные CSI использовались для определения филогенетического родства ряда типов бактерий. Большой CSI, состоящий примерно из 150–180 аминокислот в консервативной области гиразы B (между аминокислотами 529–751), обычно является общим для различных видов Pseudomonadota , Chlamydiota , Planctomycetota и Aquiificota . Этот CSI отсутствует у других предковых типов бактерий, а также у архей . [9] Аналогичным образом, большой CSI из примерно 100 аминокислот в гомологах RpoB (между аминокислотами 919-1058) присутствует у различных видов, принадлежащих к Pseudomonadota, Bacteroidota , Chlorobiota , Chlamydiota , Planctomycetota и Aquiificota. Этот CSI отсутствует у других предковых типов бактерий, а также у архей. [10] [11] В обоих случаях можно сделать вывод, что группы, лишенные CSI, являются предковыми.


Ключевой проблемой бактериальной филогении является понимание того, как разные виды бактерий связаны друг с другом и порядок их ветвления от общего предка. В настоящее время большинство филогенетических деревьев основаны на 16S рРНК или других генах/белках. Эти деревья не всегда способны с высокой степенью достоверности решить ключевые филогенетические вопросы. [12] [13] [14] [15] [16] Однако в последние годы открытие и анализ консервативных инделей (CSI) во многих повсеместно распространенных белках помогли в этом поиске. Предполагается, что генетические события, приводящие к ним, произошли в важных точках эволюционного ветвления, а модели их видового распределения предоставляют ценную информацию о порядке ветвления и взаимосвязях между различными бактериальными типами. [1] [2] [8]
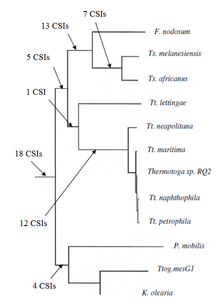
Недавно филогенетическое родство группы Thermotogota было охарактеризовано на основе подхода CSI. Ранее не было известно ни биохимических, ни молекулярных маркеров , позволяющих четко отличить виды этого типа от всех других бактерий. Было обнаружено более 60 CSI, специфичных для всего типа Thermotogota или его различных подгрупп. 18 CSI уникально присутствуют у различных видов Thermotogota и служат молекулярными маркерами этого типа. Кроме того, было много CSI, специфичных для различных подгрупп Thermotogota. 12 CSI были специфичны для клады, состоящей из различных видов Thermotogota, за исключением Tt. Леттинге. 14 CSI были специфичны для клады, состоящей из родов Fervidobacterium и Thermosipho , и 18 CSI были специфичны для рода Thermosiphon . [ нужна цитата ]
Наконец, было зарегистрировано 16 CSI, которые были общими для некоторых или всех видов Thermotogota или некоторых видов из других таксонов, таких как Archaea , Aquiificota , Bacillota , Pseudomonadota , Deinococcota , Fusobacteriota , Dictyoglomota , Chloroflexota и эукариоты . Совместное присутствие некоторых из этих CSI может быть связано с латеральным переносом генов (LGT) между этими группами. Однако количество CSI, которые обычно являются общими с другими таксонами, намного меньше, чем тех, которые специфичны для Thermotogota, и они не демонстрируют какой-либо конкретной закономерности. Следовательно, они не оказывают существенного влияния на различие Thermotogota. [8]
Мезофильные термопротеоты недавно были помещены в новый тип архей , названный Nitrososphaerota (ранее Thaumarchaeota). Однако существует очень мало молекулярных маркеров, которые могут отличить эту группу архей от типа Thermoproteota (ранее Crenarchaeota). Детальное филогенетическое исследование с использованием подхода CSI было проведено для различения этих типов в молекулярном отношении. 6 CSI были обнаружены уникальным образом у различных Nitrososphaerota, а именно у Cenarchaeum symbiosum , Nitrosopumilus maritimus и ряда некультивируемых морских Thermoproteota. Было обнаружено 3 CSI, которые обычно были общими для видов, принадлежащих к Nitrososphaerota и Thermoproteota. Кроме того, был обнаружен ряд CSI, специфичных для разных отрядов Thermoproteota: 3 CSI для Sulfolobales , 5 CSI для Thermoproteales , наконец, 2 CSI, общие для Sulfolobales и Desulfurococcales . Описанные сигнатуры предоставляют новые средства для различения Thermoproteota и Nitrososphaerota, а также могут использоваться в качестве инструмента для классификации и идентификации родственных видов. [17]
Членов отряда Pasteurellales в настоящее время различают главным образом по их положению в ветвлении дерева 16срРНК. В настоящее время известно очень мало молекулярных маркеров, позволяющих отличить представителей этого отряда от других бактерий. Недавно подход CSI был использован для выяснения филогенетических отношений между видами в этом порядке; было обнаружено более 40 CSI, которые уникально присущи всем или большинству видов. Внутри этой Pasteurellales формируются две основные клады: клада I, включающая Aggregatibacter , Pasteurella , Actinobacillus succinogenes , Mannheimia succiniciproducens , Haemophilus influenzae и Haemophilus somnus , поддерживается 13 CSI. Клада II, включающая Actinobacillus pleuropneumoniae , Actinobacillusminor , Haemophilus ducreyi , Mannheimia haemolytica и Haemophilus parasuis , была поддержана 9 CSI. На основании этих результатов было предложено разделить Pasteurellales из нынешнего одного семейства на два разных. Кроме того, описанные сигнатуры предоставят новые средства идентификации неоткрытых видов Pasteurellales. [18]
Класс Gammaproteobacteria образует одну из крупнейших групп бактерий. В настоящее время его отличают от других бактерий исключительно филогенетические деревья, основанные на 16s рРНК . Никаких молекулярных характеристик, уникальных для этого класса или его различных подгрупп, неизвестно. Чтобы лучше понять филогению этого класса, было проведено подробное исследование на основе CSI. Во-первых, было создано филогенетическое дерево, основанное на конкатенированных последовательностях ряда повсеместно распространенных белков. Порядок ветвления различных порядков класса Gammaproteobacteria (от самых последних до самых ранних) был следующим: Enterobacteriales > Pasteurellales > Vibrionales , Aeromonadales > Alteromonadales > Oceanospirillales , Pseudomonadales > Chromatiales, Legionellales , Mmethylococcales , Xanthomonadales , Cardiobacteriales , Thiotrichales . Кроме того, были обнаружены 4 CSI, уникальные для большинства видов класса Gammaproteobacteria. Делеция 2 аа в трансформилазе AICAR была уникальной для всех гаммапротеобактерий, за исключением Francisco tularensis . Делеция 4 ак в b-субъединице РНК-полимеразы и делеция 1 ак в рибосомном белке L16 были обнаружены уникально у различных видов, принадлежащих к порядкам Enterobacteriales, Pasteurellales, Vibrionales, Aeromonadales и Alteromonadales, но не были обнаружены у других гаммапротеобактерий. Наконец, делеция 2 ак в лейцил-тРНК-синтетазе обычно присутствовала у вышеупомянутых отрядов класса Gammaproteobacteria и у некоторых представителей отряда Oceanospirillales. [19] Другое исследование, основанное на CSI, также выявило 4 CSI, которые являются эксклюзивными для отряда Xanthomonadales. В совокупности эти два факта показывают, что Xanthomonadales являются монофилетической группой , являющейся предком других Gammaproteobacteria, что еще раз показывает, что Xanthomonadales является независимым подразделением и представляет собой одну из самых глубоко разветвленных линий внутри клады Gammaproteobacteria. [4] [19]
{{cite web}}: CS1 maint: bot: исходный статус URL неизвестен ( ссылка )