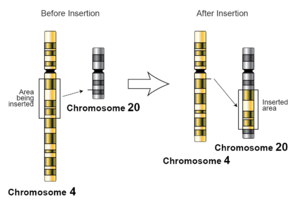
В генетике инсерция (также называемая инсерционной мутацией ) — это добавление одной или нескольких пар нуклеотидных оснований в последовательность ДНК . Это часто может происходить в микросателлитных областях из-за проскальзывания ДНК-полимеразы . Вставки могут быть любого размера: от одной пары оснований, неправильно вставленной в последовательность ДНК, до участка одной хромосомы, вставленного в другую. Считается, что механизм вставочных мутаций наименьшего одного основания заключается в разделении пар оснований между нитями матрицы и праймера с последующим укладкой несмежных оснований, которое может происходить локально в активном сайте ДНК-полимеразы. [1] На уровне хромосомы вставка означает вставку более крупной последовательности в хромосому. Это может произойти из-за неравного кроссинговера во время мейоза .
Добавление N-области представляет собой добавление некодируемых нуклеотидов во время рекомбинации терминальной дезоксинуклеотидилтрансферазой .
Инсерция P-нуклеотида представляет собой вставку палиндромных последовательностей , кодируемых концами рекомбинирующих генных сегментов.
Тринуклеотидные повторы классифицируют как инсерционные мутации [2] [3] , а иногда и как отдельный класс мутаций. [4]
Нуклеаза цинковых пальцев (ZFN) , эффекторные нуклеазы, подобные активатору транскрипции (TALEN) , и редактирование генов CRISPR — три основных метода, использованных в предыдущих исследованиях для внедрения генов. А инструменты CRISPR/Cas уже стали одним из наиболее часто используемых методов представления исследований.
На основе инструментов CRISPR/Cas уже разработаны различные системы для реализации конкретных функций. Например, одной из стратегий является система разрезания двухцепочечных нуклеаз с использованием обычного белка Cas9 с одной направляющей РНК (sgRNA) и последующим внедрением гена посредством соединения концов или деления клеток с помощью системы репарации ДНК . [5] Другим примером является система первичного редактирования , в которой используется никаза Cas9 и направляющая РНК первичного редактирования (пегРНК), несущая целевые гены. [5]
Одним из ограничений современной технологии является то, что размер для точной вставки ДНК недостаточно велик [6] для удовлетворения потребностей в исследованиях генома. Транспозиция ДНК под контролем РНК является новой областью решения этой проблемы. [7] Ожидается, что более эффективные методы будут разработаны и применены в области генной инженерии.
Вставки могут быть особенно опасными, если они происходят в экзоне , кодирующей аминокислоту области гена . Мутация сдвига рамки считывания , изменение нормальной рамки считывания гена, возникает, если количество вставленных нуклеотидов не делится на три, то есть количество нуклеотидов на кодон . Мутации сдвига рамки считывания изменят все аминокислоты, кодируемые геном после мутации. Обычно вставки и последующая мутация сдвига рамки считывания приводят к тому, что активная трансляция гена сталкивается с преждевременным стоп-кодоном , что приводит к прекращению трансляции и образованию усеченного белка. Транскрипты, несущие мутацию сдвига рамки считывания, также могут разрушаться в результате нонсенс-опосредованного распада во время трансляции, что не приводит к образованию какого-либо белкового продукта. При трансляции усеченные белки часто не могут функционировать должным образом или вообще не могут функционировать и могут привести к любому количеству генетических нарушений в зависимости от гена, в котором происходит вставка. [8]
Вставки внутри кадра происходят, когда рамка считывания не изменяется в результате вставки; количество вставленных нуклеотидов делится на три. Рамка считывания остается неповрежденной после вставки, и трансляция, скорее всего, завершится до завершения, если вставленные нуклеотиды не кодируют стоп-кодон. Однако из-за вставленных нуклеотидов готовый белок будет содержать, в зависимости от размера вставки, множество новых аминокислот, которые могут повлиять на функцию белка.