
Комплекс пируватдегидрогеназы ( PDC ) представляет собой комплекс из трех ферментов , которые превращают пируват в ацетил-КоА посредством процесса, называемого декарбоксилированием пирувата . [1] Ацетил-КоА затем может использоваться в цикле лимонной кислоты для осуществления клеточного дыхания , и этот комплекс связывает метаболический путь гликолиза с циклом лимонной кислоты . Декарбоксилирование пирувата также известно как «реакция пируватдегидрогеназы», поскольку оно также включает окисление пирувата. [2]
Этот мультиферментный комплекс структурно и функционально связан с мультиферментными комплексами оксоглутаратдегидрогеназы и оксокислотной дегидрогеназы с разветвленной цепью .
Реакция, катализируемая пируватдегидрогеназным комплексом:

Субъединица E1, называемая субъединицей пируватдегидрогеназы , представляет собой либо гомодимер (содержащий две цепи «ɑ», например, у Escherichia coli ), либо гетеротетрамер из двух разных цепей (две цепи «ɑ» и две «ꞵ»). Ион магния образует 4-координационный комплекс с тремя полярными аминокислотными остатками (Asp, Asn и Tyr), расположенными в альфа-цепи, и кофактором тиаминдифосфата (TPP) , непосредственно участвующим в декарбоксилировании пирувата . [3] [4]
Субъединица E2, или дигидролипоилацетилтрансфераза, как у прокариот, так и у эукариот, обычно состоит из трех доменов. N-концевой домен (липоильный домен) состоит из 1–3 липоильных групп примерно по 80 аминокислот каждая. Домен, связывающий периферическую субъединицу (PSBD), служит местом селективного связывания для других доменов субъединиц E1 и E3. Наконец, С-концевой (каталитический) домен катализирует перенос ацетильных групп и синтез ацетил-КоА. [5]

Субъединица Е3, называемая ферментом дигидролипоилдегидрогеназой , характеризуется как гомодимерный белок, в котором два остатка цистеина , участвующие в дисульфидной связи , и кофактор FAD в активном центре облегчают его основную функцию в качестве окислительного катализатора. Один из примеров структуры E3, обнаруженной у Pseudomonas putida , сформирован таким образом, что каждая отдельная субъединица гомодимера содержит два связывающих домена, ответственных за связывание FAD и связывание NAD, а также центральный домен и интерфейсный домен. [6] [7]
Вспомогательным белком, уникальным для большинства эукариот, является белок, связывающий Е3 (E3BP), который служит для связывания субъединицы E3 с комплексом PDC. В случае человеческого E3BP гидрофобные остатки пролина и лейцина в BP взаимодействуют с поверхностным сайтом узнавания, образованным при связывании двух идентичных мономеров E3. [8]
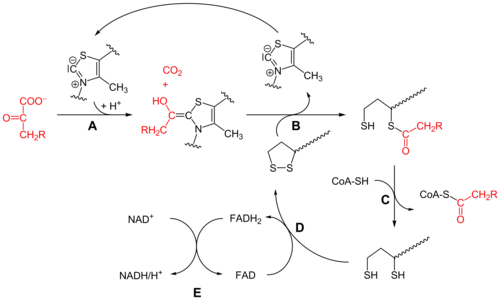
Первоначально пируват и тиаминпирофосфат (TPP или витамин B 1 ) связываются субъединицами пируватдегидрогеназы . [1] Тиазолиевое кольцо TPP находится в цвиттер-ионной форме , а анионный углерод C2 осуществляет нуклеофильную атаку на карбонил C2 (кетон) пирувата. Полученный гемитиоацеталь подвергается декарбоксилированию с образованием эквивалента ацил-аниона (см. химию циангидрина или альдегид-дитиана умполунга , а также бензоиновую конденсацию ). Этот анион атакует S1 окисленного липоата, присоединенного к остатку лизина . В механизме, подобном раскрытию кольца S N 2 , S2 замещается в виде сульфидного или сульфгидрильного фрагмента. Последующий коллапс тетраэдрического гемитиоацеталя выбрасывает тиазол, высвобождая кофактор TPP и образуя тиоацетат на S1 липоата. Процесс, катализируемый E1, является стадией, лимитирующей скорость всего пируватдегидрогеназного комплекса.
В этот момент липоат-тиоэфирная функциональность перемещается в активный центр дигидролипоилтрансацетилазы (E2), [1] где реакция трансацилирования переносит ацетил с «качающегося плеча» липоила на тиол кофермента А. При этом образуется ацетил-КоА , который высвобождается из ферментного комплекса и впоследствии входит в цикл лимонной кислоты . E2 также может быть известен как липоамидредуктаза-трансацетилаза.
Дигидролипоат , все еще связанный с остатком лизина комплекса, затем мигрирует к активному центру дигидролипоилдегидрогеназы (Е3), [1] где он подвергается флавин -опосредованному окислению, идентичному по химическому составу дисульфид-изомеразе . Во-первых, FAD окисляет дигидролипоат обратно в липоатное состояние покоя, образуя FADH 2 . Затем кофактор НАД + окисляет ФАДН 2 обратно в состояние покоя ФАД, образуя НАДН и H.
PDC представляет собой большой комплекс, состоящий из множества копий 3 или 4 субъединиц в зависимости от вида.
У грамотрицательных бактерий, например Escherichia coli , PDC состоит из центрального кубического ядра, состоящего из 24 молекул дигидролипоилтрансацетилазы ( Е2). До 32 копий пируватдегидрогеназы (Е1) и 16 молекул дигидролипоилдегидрогеназы (Е3) связываются с PSBD E2 вокруг ядра E2. У гаммапротеобактерий специфичность PSBD к связыванию E1 или E3 определяется его олигомерным состоянием.[9]
Напротив, у грамположительных бактерий (например, Bacillus stearothermophilus ) и эукариотов центральное ядро PDC содержит 60 молекул E2, организованных в икосаэдр. Это «ядро» субъединицы E2 координирует 30 субъединиц E1 и 12 копий E3.
Эукариоты также содержат 12 копий дополнительного корового белка, E3-связывающего белка (E3BP), который связывает субъединицы E3 с ядром E2. [10] Точное расположение E3BP не совсем ясно. Криоэлектронная микроскопия установила, что E3BP связывается с каждой из икосаэдрических граней дрожжей. [11] Однако было высказано предположение, что он заменяет эквивалентное количество молекул E2 в ядре бычьего PDC.
До 60 молекул E1 или E3 могут связываться с ядром E2 грамположительных бактерий – связывание является взаимоисключающим. У эукариот E1 специфически связывается с E2, а E3 — с E3BP. Считается, что присутствует до 30 ферментов E1 и 6 E3, хотя точное количество молекул может варьироваться in vivo и часто отражает метаболические потребности рассматриваемой ткани.
Пируватдегидрогеназа ингибируется при увеличении одного или нескольких из трех следующих соотношений: АТФ / АДФ , НАДН /НАД + и ацетил-КоА / КоА . [ нужна цитата ]
У эукариот PDC жестко регулируется собственной специфической киназой пируватдегидрогеназы (PDK) и фосфатазой пируватдегидрогеназы (PDP), деактивируя и активируя ее соответственно. [12]
Продукты реакции действуют как аллостерические ингибиторы ПДК, поскольку активируют ПДК. Субстраты, в свою очередь, ингибируют PDK, реактивируя PDC.
Во время голодания количество PDK увеличивается в большинстве тканей, включая скелетные мышцы , за счет увеличения транскрипции генов . При этих же условиях количество ПДП уменьшается. В результате ингибирование PDC предотвращает катаболизм глюкозы и предшественников глюконеогенеза в мышцах и других тканях . Метаболизм смещается в сторону утилизации жира , в то время как распад мышечного белка для обеспечения предшественников глюконеогенеза сводится к минимуму, а доступная глюкоза сохраняется для использования мозгом . [ нужна цитата ]
Ионы кальция играют роль в регуляции PDC в мышечной ткани, поскольку они активируют PDP, стимулируя гликолиз при его высвобождении в цитозоль – во время мышечного сокращения . Некоторые продукты этой транскрипции выделяют H2 в мышцы. Это может привести к распаду ионов кальция с течением времени. [ нужна цитата ]
В эукариотических клетках декарбоксилирование пирувата происходит внутри митохондриального матрикса после транспорта субстрата пирувата из цитозоля . Транспорт пирувата в митохондрии осуществляется посредством транспортного белка пируват-транслоказы. Пируваттранслоказа переносит пируват симпортным способом с протоном (через внутреннюю митохондриальную мембрану), что можно рассматривать как форму вторичного активного транспорта, но для использования дескриптора «вторичный активный транспорт» может потребоваться дальнейшее подтверждение/подтверждение. здесь (Примечание: метод транспортировки пирувата посредством пируваттранслоказы, по-видимому, связан с протонным градиентом согласно S. Papa et al., 1971, что, по-видимому, соответствует по определению вторичному активному транспорту). [13]
Альтернативные источники говорят, что «транспорт пирувата через внешнюю митохондриальную мембрану, по-видимому, легко осуществляется через крупные неселективные каналы, такие как потенциал-зависимые анионные каналы , которые обеспечивают пассивную диффузию», а транспорт через внутреннюю митохондриальную мембрану опосредуется митохондриальным переносчиком пирувата 1 ( MPC1) и митохондриальный пируватный носитель 2 (MPC2). [14]
При попадании в митохондриальный матрикс пируват декарбоксилируется с образованием ацетил-КоА (а также диоксида углерода и НАДН). Эта необратимая реакция захватывает ацетил-КоА внутри митохондрий (ацетил-КоА может транспортироваться из митохондриального матрикса только в условиях высокого содержания оксалоацетата через цитратный челнок, промежуточный продукт ТСА, которого обычно мало). Диоксид углерода, образующийся в результате этой реакции, неполярен и мал по размеру и может диффундировать из митохондрий и из клетки. [ нужна цитата ]
У прокариот , не имеющих митохондрий, эта реакция либо осуществляется в цитозоле, либо не осуществляется вообще. [ нужна цитата ]
Было обнаружено, что фермент пируватдегидрогеназа, обнаруженный в митохондриях эукариотических клеток, очень похож на фермент Geobacillus stearothermophilus , который является разновидностью грамположительных бактерий . Несмотря на сходство комплекса пируватдегидрогеназы с грамположительными бактериями, сходства с таковым у грамотрицательных бактерий мало . Сходство четвертичных структур пируватдегидрогеназы и ферментов грамположительных бактерий указывает на общую эволюционную историю, которая отличается от истории эволюции соответствующих ферментов, обнаруженных у грамотрицательных бактерий. Пируватдегидрогеназа, обнаруженная в митохондриях эукариот, в результате эндосимбиоза указывает на наследственные связи, восходящие к грамположительным бактериям. [15]
Комплексы пируватдегидрогеназы имеют много общего с дегидрогеназой 2-оксокислот с разветвленной цепью (BCOADH), особенно в их субстратной специфичности в отношении альфа-кетокислот. В частности, BCOADH катализирует распад аминокислот, и эти ферменты должны были преобладать в те периоды, когда на доисторической Земле доминировала среда, богатая аминокислотами. Субъединица E2 пируватдегидрогеназы произошла от гена E2, обнаруженного в BCOADH, в то время как оба фермента содержат идентичные субъединицы E3 из-за присутствия только одного гена E3. Поскольку субъединицы E1 обладают отличительной специфичностью к конкретным субстратам, субъединицы E1 пируватдегидрогеназы и BCOADH различаются, но имеют генетическое сходство. Грамположительные бактерии и цианобактерии, которые позже дали начало митохондриям и хлоропластам, обнаруженным в эукариотических клетках, сохранили субъединицы E1, которые генетически связаны с субъединицами, обнаруженными в ферментах BCOADH. [16] [17]
Дефицит пируватдегидрогеназы (PDCD) может быть результатом мутаций любого из ферментов или кофакторов, используемых для построения комплекса. Его основным клиническим признаком является лактоацидоз . [18] Такие мутации PDCD, приводящие к последующему дефициту продукции NAD и FAD, препятствуют процессам окислительного фосфорилирования, которые являются ключевыми в аэробном дыхании. Таким образом, вместо этого ацетил-КоА восстанавливается посредством анаэробных механизмов до других молекул, таких как лактат, что приводит к избытку лактата в организме и связанным с ним неврологическим патологиям. [19]
Хотя дефицит пируватдегидрогеназы встречается редко, существует множество различных генов, мутировавших или нефункциональных, которые могут вызвать этот дефицит. Во-первых, субъединица Е1 пируватдегидрогеназы содержит четыре различных субъединицы: две альфа-субъединицы, обозначаемые как Е1-альфа, и две бета-субъединицы, обозначаемые как Е1-бета. Ген PDHA1, обнаруженный в субъединицах E1-альфа, при мутации вызывает 80% случаев дефицита пируватдегидрогеназы, поскольку эта мутация сокращает белок E1-альфа. Снижение функционального уровня Е1-альфа препятствует достаточному связыванию пируватдегидрогеназы с пируватом, тем самым снижая активность всего комплекса. [20] Когда ген PDHB, обнаруженный в бета-субъединице E1 комплекса, мутирует, это также приводит к дефициту пируватдегидрогеназы. [21] Аналогичным образом, мутации, обнаруженные в других субъединицах комплекса, таких как ген DLAT, обнаруженный в субъединице E2, ген PDHX, обнаруженный в субъединице E3, а также мутация в гене фосфатазы пируватдегидрогеназы, известном как PDP1, имеют все они связаны с дефицитом пируватдегидрогеназы, тогда как их конкретный вклад в болезненное состояние неизвестен. [22] [23] [24]