Фаговый дисплей — это лабораторный метод изучения взаимодействий белок-белок , белок - пептид и белок- ДНК , который использует бактериофаги ( вирусы , которые заражают бактерии ) для соединения белков с генетической информацией , которая их кодирует . [1] В этом методе ген, кодирующий интересующий белок, вставляется в ген белка оболочки фага , заставляя фаг «отображать» белок снаружи, в то время как ген белка находится внутри, что приводит к связи между генотипом и фенотипом . Белки, которые отображают фаги, затем можно скринировать против других белков, пептидов или последовательностей ДНК, чтобы обнаружить взаимодействие между отображаемым белком и белком других молекул. Таким образом, большие библиотеки белков могут быть скринированы и амплифицированы в процессе, называемом селекцией in vitro , которая аналогична естественному отбору .
Наиболее распространенными бактериофагами, используемыми в фаговом дисплее, являются нитчатые фаги M13 и fd , [2] [3], хотя также использовались фаги T4 , [4] T7 и λ .
Фаговый дисплей был впервые описан Джорджем П. Смитом в 1985 году, когда он продемонстрировал отображение пептидов на нитчатом фаге (длинные тонкие вирусы, которые заражают бактерии) путем слияния капсидного белка вируса с одним пептидом из набора пептидных последовательностей. [1] Это отображало различные пептиды на внешних поверхностях набора вирусных клонов, где этап скрининга процесса изолировал пептиды с самой высокой аффинностью связывания. В 1988 году Стивен Пармли и Джордж Смит описали биопэннинг для аффинного отбора и продемонстрировали, что рекурсивные раунды отбора могут обогащать клоны, присутствующие в количестве 1 на миллиард или меньше. [5] В 1990 году Джейми Скотт и Джордж Смит описали создание больших случайных пептидных библиотек, отображаемых на нитчатом фаге. [6] Технология фагового дисплея была дополнительно разработана и улучшена группами в Лаборатории молекулярной биологии с Грегом Винтером и Джоном Маккафферти , Научно- исследовательском институте Скриппса с Ричардом Лернером и Карлосом Барбасом и Немецком центре исследований рака с Фрэнком Брайтлингом и Стефаном Дюбелем для отображения белков, таких как антитела , для терапевтической белковой инженерии . Смит и Винтер были награждены половиной доли Нобелевской премии по химии 2018 года за их вклад в разработку фагового дисплея. [7] Патент Джорджа Печеника, претендующий на приоритет с 1985 года, также описывает создание пептидных библиотек. [8]
Как и двухгибридная система , фаговый дисплей используется для высокопроизводительного скрининга белковых взаимодействий. В случае нитевидного фагового дисплея M13 ДНК, кодирующая интересующий белок или пептид, лигируется в ген pIII или pVIII, кодирующий либо минорный, либо мажорный белок оболочки соответственно. Иногда используются множественные сайты клонирования , чтобы гарантировать, что фрагменты вставлены во все три возможные рамки считывания, так что фрагмент кДНК транслируется в правильной рамке. Затем гибрид гена фага и вставки ДНК вставляется (процесс, известный как « трансдукция ») в бактериальные клетки E. coli, такие как TG1, SS320, ER2738 или XL1-Blue E. coli . Если используется вектор « фагемида » (упрощенный дисплейный конструктный вектор), фаговые частицы не будут высвобождаться из клеток E. coli , пока они не будут инфицированы фагом-помощником , что позволяет упаковывать фаговую ДНК и собирать зрелые вирионы с соответствующим фрагментом белка как часть их внешнего покрытия на малом (pIII) или большом (pVIII) белке покрытия. Иммобилизуя соответствующую ДНК или белковую цель(и) на поверхности лунки микротитровального планшета , фаг, который отображает белок, связывающийся с одной из этих целей на своей поверхности, останется, в то время как другие будут удалены промывкой. Те, что остались, могут быть элюированы , использованы для получения большего количества фага (путем бактериального заражения фагом-помощником) и для получения смеси фагов, которая обогащена соответствующим (т. е. связывающим) фагом. Повторный цикл этих шагов называется «пэннингом» в отношении обогащения образца золота путем удаления нежелательных материалов. Фаг, элюированный на последнем этапе, можно использовать для заражения подходящего бактериального хозяина, из которого можно собрать фагмиды, а затем вырезать и секвенировать соответствующую последовательность ДНК для идентификации соответствующих взаимодействующих белков или фрагментов белков. [ необходима ссылка ]
Использование вспомогательного фага можно исключить, используя технологию «клеточной линии бактериальной упаковки». [9]
Элюирование можно осуществить, комбинируя буфер элюирования с низким pH с сонификацией, которая, в дополнение к ослаблению взаимодействия пептид-мишень, также служит для отсоединения целевой молекулы от поверхности иммобилизации. Этот основанный на ультразвуке метод позволяет проводить одноэтапный отбор высокоаффинного пептида. [10]
Применение технологии фагового дисплея включает определение партнеров взаимодействия белка (который будет использоваться в качестве иммобилизованной фаговой «приманки» с библиотекой ДНК, состоящей из всех кодирующих последовательностей клетки, ткани или организма), так что функция или механизм функции этого белка могут быть определены. [11] Фаговый дисплей также является широко используемым методом для эволюции белка in vitro (также называемой белковой инженерией ). Таким образом, фаговый дисплей является полезным инструментом в открытии лекарств . Он используется для поиска новых лигандов (ингибиторов ферментов, агонистов и антагонистов рецепторов) для целевых белков. [12] [13] [14] Эта техника также используется для определения опухолевых антигенов (для использования в диагностике и терапевтическом нацеливании) [15] и для поиска взаимодействий белок-ДНК [16] с использованием специально сконструированных библиотек ДНК с рандомизированными сегментами. В последнее время фаговый дисплей также использовался в контексте лечения рака, например, в подходе адоптивного переноса клеток. [17] В этих случаях фаговый дисплей используется для создания и отбора синтетических антител, нацеленных на белки поверхности опухоли. [17] Они превращаются в синтетические рецепторы для Т-клеток, полученных от пациента, которые используются для борьбы с заболеванием. [18]
Конкурирующие методы эволюции белков in vitro включают дрожжевой дисплей , бактериальный дисплей , рибосомный дисплей и дисплей мРНК . [ необходима ссылка ]
Изобретение фагового дисплея антител произвело революцию в обнаружении лекарств на основе антител. Первоначальная работа была проделана лабораториями Лаборатории молекулярной биологии MRC ( Грег Винтер и Джон Маккафферти ), Исследовательского института Скриппса (Ричард Лернер и Карлос Ф. Барбас) и Немецкого центра исследований рака (Фрэнк Брейтлинг и Стефан Дюбель). [19] [20] [21] В 1991 году группа Скриппса сообщила о первом отображении и отборе человеческих антител на фаге. [22] Это первоначальное исследование описывало быстрое выделение Fab-фрагмента человеческого антитела , который связывал столбнячный токсин , и метод затем был расширен для быстрого клонирования человеческих антител против ВИЧ-1 для разработки вакцин и терапии. [23] [24] [25] [26] [27]
Фаговый дисплей библиотек антител стал мощным методом как для изучения иммунного ответа, так и для быстрого отбора и развития человеческих антител для терапии. Антитело-фаговый дисплей был позже использован Карлосом Ф. Барбасом в Научно-исследовательском институте Скриппса для создания синтетических библиотек человеческих антител, принцип, впервые запатентованный в 1990 году Брейтлингом и его коллегами (Патент CA 2035384), что позволило создавать человеческие антитела in vitro из синтетических элементов разнообразия. [28] [29] [30] [31]
Библиотеки антител, отображающие миллионы различных антител на фаге, часто используются в фармацевтической промышленности для выделения высокоспецифичных терапевтических антител для разработки лекарств на основе антител, в первую очередь, как противораковых или противовоспалительных терапевтических средств. Одним из самых успешных был адалимумаб , открытый Cambridge Antibody Technology как D2E7 и разработанный и продаваемый Abbott Laboratories . Адалимумаб, антитело к TNF-альфа , был первым в мире полностью человеческим антителом [32], годовой объем продаж которого превысил 1 млрд долларов. [33]
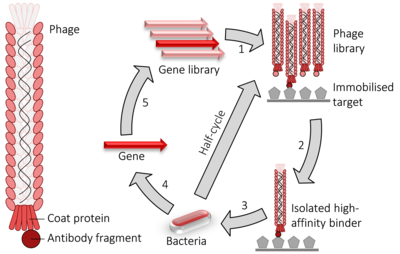
Ниже представлена последовательность событий, которые выполняются при скрининге фагового дисплея для идентификации полипептидов, которые связываются с высокой аффинностью с желаемым целевым белком или последовательностью ДНК: [ необходима ссылка ]
pIII — это белок, определяющий инфекционность вириона. pIII состоит из трех доменов (N1, N2 и CT), соединенных линкерами, богатыми глицином. [34] Домен N2 связывается с F-пилусом во время инфицирования вириона, освобождая домен N1, который затем взаимодействует с белком TolA на поверхности бактерии. [34] Вставки в этот белок обычно добавляются в положение 249 (внутри линкерной области между CT и N2), положение 198 (внутри домена N2) и на N-конце (вставлены между N-концевой секреционной последовательностью и N-концом pIII). [34] Однако при использовании сайта BamHI, расположенного в положении 198, необходимо соблюдать осторожность с неспаренным остатком цистеина (C201), который может вызвать проблемы во время фагового отображения, если используется неусеченная версия pIII. [34]
Преимущество использования pIII вместо pVIII заключается в том, что pIII допускает моновалентный дисплей при использовании фагмиды (плазмиды, полученной из фагов Ff ) в сочетании с фагом-помощником. Более того, pIII допускает вставку более крупных последовательностей белков (>100 аминокислот) [35] и более устойчив к этому, чем pVIII. Однако использование pIII в качестве партнера по слиянию может привести к снижению инфекционности фага, что приведет к таким проблемам, как смещение отбора, вызванное разницей в скорости роста фага [36] или, что еще хуже, неспособность фага заражать своего хозяина. [34] Потери инфекционности фага можно избежать, используя плазмиду фагмиды и фаг-помощник, так что полученный фаг будет содержать как дикий тип, так и слитый pIII. [34]
cDNA также анализировалась с использованием pIII через систему двух комплементарных лейциновых молний [37] Direct Interaction Rescue [38] или путем добавления линкера из 8-10 аминокислот между cDNA и pIII на C-конце. [39]
pVIII является основным белком оболочки фагов Ff. Пептиды обычно сливаются с N-концом pVIII. [34] Обычно пептиды, которые могут быть слиты с pVIII, имеют длину 6-8 аминокислот. [34] Ограничение размера, по-видимому, в меньшей степени связано со структурным препятствием, вызванным добавленной секцией [40] , а в большей степени с исключением размера, вызванным pIV во время экспорта белка оболочки. [40] Поскольку на типичном фаге имеется около 2700 копий белка, более вероятно, что интересующий белок будет экспрессироваться поливалентно, даже если используется фагмида. [34] Это делает использование этого белка неблагоприятным для обнаружения партнеров по связыванию с высокой аффинностью. [34]
Для преодоления проблемы размера pVIII были разработаны искусственные белки оболочки. [41] Примером является инвертированный искусственный белок оболочки (ACP) Вайса и Сидху, который позволяет отображать большие белки на C-конце. [41] ACP могли отображать белок 20 кДа, однако только на низких уровнях (в основном только моновалентно). [41]
pVI широко использовался для отображения библиотек кДНК. [34] Отображение библиотек кДНК с помощью фагового дисплея является привлекательной альтернативой методу дрожжевого-2-гибрида для обнаружения взаимодействующих белков и пептидов из-за его высокой пропускной способности. [34] pVI использовался предпочтительно по сравнению с pVIII и pIII для экспрессии библиотек кДНК, поскольку можно добавить интересующий белок к C-концу pVI, не влияя существенно на роль pVI в сборке фага. Это означает, что стоп-кодон в кДНК больше не является проблемой. [42] Однако фаговое отображение кДНК всегда ограничено неспособностью большинства прокариот производить посттрансляционные модификации, присутствующие в эукариотических клетках, или неправильным сворачиванием многодоменных белков.
В то время как pVI был полезен для анализа библиотек кДНК, pIII и pVIII остаются наиболее используемыми белками оболочки для фагового дисплея. [34]
В эксперименте 1995 года была предпринята попытка отображения глутатион-S-трансферазы как на pVII, так и на pIX, но она не удалась. [43] Однако фаговое отображение этого белка было успешно завершено после добавления периплазматической сигнальной последовательности (pelB или ompA) на N-конце. [44] В недавнем исследовании было показано, что AviTag, FLAG и His могут быть отображены на pVII без необходимости в сигнальной последовательности. Затем экспрессия одноцепочечных Fv (scFv) и одноцепочечных Т-клеточных рецепторов (scTCR) была выражена как с сигнальной последовательностью, так и без нее. [45]
PelB (аминокислотная сигнальная последовательность, которая направляет белок в периплазму, где сигнальная пептидаза затем отщепляет PelB) улучшила уровень фагового дисплея по сравнению со слияниями pVII и pIX без сигнальной последовательности. Однако это привело к включению большего количества геномов фагов-помощников, а не геномов фагмид. Во всех случаях уровни фагового дисплея были ниже, чем при использовании слияния pIII. Однако более низкий дисплей может быть более благоприятным для выбора связующих веществ, поскольку более низкий дисплей ближе к истинному моновалентному дисплею. В пяти из шести случаев слияния pVII и pIX без pelB были более эффективными, чем слияния pIII в анализах аффинного отбора. В статье даже утверждается, что платформы отображения pVII и pIX могут превзойти pIII в долгосрочной перспективе. [45]
Использование pVII и pIX вместо pIII также может быть преимуществом, поскольку спасение вириона может быть осуществлено без разрыва связи вирион-антиген, если используемый pIII является диким типом. Вместо этого можно было бы расщепить участок между шариком и антигеном для элюирования. Поскольку pIII не поврежден, неважно, остается ли антиген связанным с фагом. [45]
Проблема использования фагов Ff для фагового дисплея заключается в том, что они требуют, чтобы интересующий белок был перемещен через внутреннюю мембрану бактерий, прежде чем он будет собран в фаг. [46] Некоторые белки не могут пройти этот процесс и, следовательно, не могут быть отображены на поверхности фагов Ff. В этих случаях вместо этого используется фаговый дисплей T7. [46] При фаговом дисплее T7 отображаемый белок прикрепляется к C-концу капсидного белка гена 10 T7. [46]
Недостатком использования T7 является то, что размер белка, который может быть выражен на поверхности, ограничен более короткими пептидами, поскольку большие изменения в геноме T7 не могут быть размещены, как в M13, где фаг просто удлиняет свою оболочку, чтобы вместить более крупный геном внутри себя. Однако это может быть полезно для производства большой библиотеки белков для отбора scFV, где scFV экспрессируется на фаге M13, а антигены экспрессируются на поверхности фага T7. [47]
Базы данных и вычислительные инструменты для мимотопов стали важной частью изучения фагового дисплея. [48] Базы данных, [49] программы и веб-серверы [50] широко использовались для исключения пептидов, не связанных с мишенью, [51] характеризуют взаимодействия малых молекул с белками и картируют белок-белковые взаимодействия. Пользователи могут использовать трехмерную структуру белка и пептиды, выбранные из эксперимента по фаговому дисплею, для картирования конформационных эпитопов. Некоторые из быстрых и эффективных вычислительных методов доступны онлайн. [50]
Конкурирующие методы: