ДНК-штрихкодирование водорослей обычно используется для идентификации видов и филогенетических исследований. Водоросли образуют филогенетически гетерогенную группу, что означает, что применение единого универсального штрихкода/ маркера для разграничения видов нецелесообразно, поэтому для этой цели в разных группах водорослей применяются разные маркеры/штрихкоды.
Штрихкодирование ДНК диатомовых водорослей — это метод таксономической идентификации диатомовых водорослей вплоть до уровня видов . Он проводится с использованием ДНК или РНК с последующей амплификацией и секвенированием определенных консервативных участков в геноме диатомовых водорослей с последующим таксономическим назначением.
Одной из основных проблем идентификации диатомовых водорослей является то, что их часто собирают как смесь диатомовых водорослей из нескольких видов. Метабаркодирование ДНК — это процесс идентификации отдельных видов из смешанного образца ДНК окружающей среды (также называемой eDNA), которая представляет собой ДНК, извлеченную непосредственно из окружающей среды, например, из образцов почвы или воды.
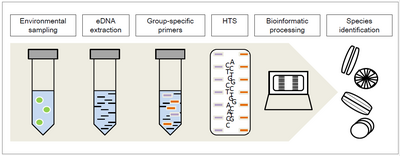
Новый применяемый метод — метабаркодирование ДНК диатомовых водорослей , которое используется для оценки экологического качества рек и ручьев из-за специфической реакции диатомовых водорослей на определенные экологические условия. Поскольку идентификация видов по морфологии является относительно сложной и требует много времени и опыта, [1] [2] метабаркодирование ДНК с высокой пропускной способностью (HTS) позволяет проводить таксономическое назначение и, следовательно, идентификацию для всего образца относительно группоспецифичных праймеров, выбранных для предыдущей амплификации ДНК .
До настоящего времени уже было разработано несколько ДНК- маркеров , в основном нацеленных на 18S рРНК . [3] При использовании гипервариабельной области V4 малой субъединицы рибосомальной ДНК (SSU рДНК) было обнаружено, что идентификация на основе ДНК более эффективна, чем классический подход, основанный на морфологии. [4] Другими консервативными областями в геномах, которые часто используются в качестве маркерных генов, являются рибулозо-1-5-бисфосфаткарбоксилаза (rbcL), цитохромоксидаза I (cox1, COI), [5] ITS [6] и 28S . [7] Было неоднократно показано, что молекулярные данные, полученные с помощью метабаркодирования диатомовой электронной ДНК, довольно точно отражают биотические индексы диатомовых водорослей, основанные на морфологии, и, следовательно, обеспечивают аналогичную оценку состояния экосистемы. [8] [9] В то же время диатомовые водоросли обычно используются для оценки экологического качества в других пресноводных экосистемах. [7] Вместе с водными беспозвоночными они считаются лучшими индикаторами нарушений, связанных с физическими, химическими или биологическими условиями водотоков. Многочисленные исследования используют бентосные диатомовые водоросли для биомониторинга. [10] [11] [12] [13] Поскольку не было найдено идеального ДНК-штрихкода диатомовых водорослей, было предложено использовать разные маркеры для разных целей. Действительно, высоковариабельные гены cox1, ITS и 28S считались более подходящими для таксономических исследований, в то время как более консервативные гены 18S и rbcL кажутся более подходящими для биомониторинга.
Применение концепции ДНК-штрихкодирования к диатомовым водорослям обещает большой потенциал для решения проблемы неточной идентификации видов и, таким образом, облегчения анализа биоразнообразия образцов окружающей среды. [14]
Молекулярные методы, основанные на технологии NGS, почти всегда приводят к большему числу идентифицированных таксонов, присутствие которых впоследствии может быть подтверждено световой микроскопией. [4] Результаты этого исследования свидетельствуют о том, что штрихкодирование eDNA диатомовых водорослей подходит для оценки качества воды и может дополнять или улучшать традиционные методы. Stoeck et al. [15] также показали, что штрихкодирование eDNA обеспечивает более глубокое понимание разнообразия диатомовых водорослей или других сообществ протистов и, следовательно, может использоваться для экологического прогнозирования глобального разнообразия. Другие исследования показали другие результаты. Например, инвентаризации, полученные с помощью молекулярного метода, были ближе к инвентаризациям, полученным с помощью морфологического метода, когда в фокусе находятся распространенные виды. [5]
ДНК-метабаркодирование также может повысить таксономическое разрешение и сопоставимость по географическим регионам, что часто бывает затруднительно при использовании только морфологических признаков. Более того, идентификация на основе ДНК позволяет расширить спектр потенциальных биоиндикаторов, включая незаметные таксономические группы, которые могут быть высокочувствительными или толерантными к определенным стрессорам. Косвенно молекулярные методы также могут помочь заполнить пробелы в знаниях об экологии видов за счет увеличения количества обработанных образцов в сочетании с уменьшением времени обработки (рентабельность), а также за счет повышения точности и достоверности корреляции между возникновением видов/MOTU и факторами окружающей среды. [16]
.jpg/440px-Naviculoid_diatom_(3074442495).jpg)
В настоящее время нет консенсуса относительно методов сохранения и выделения ДНК, выбора ДНК-штрихкодов и праймеров ПЦР, а также соглашения относительно параметров кластеризации MOTU и их таксономического назначения. [16] Отбор проб и молекулярные этапы должны быть стандартизированы посредством исследований по разработке. [5] Одним из основных ограничений является доступность справочных штрихкодов для видов диатомовых водорослей. Справочная база данных таксонов-биоиндикаторов далека от завершения, несмотря на постоянные усилия многочисленных национальных инициатив по штрихкодированию, для многих видов по-прежнему отсутствует информация о штрихкодах. Кроме того, большинство существующих данных меташтрихкодирования доступны только локально и географически разбросаны, что препятствует разработке глобально полезных инструментов. [16] Виско и др. [17] подсчитали, что в настоящее время в справочных базах данных представлены не более 30% видов европейских диатомовых водорослей. Например, наблюдается существенный дефицит ряда видов из сообществ Фенноскандии (особенно ацидофильных диатомовых водорослей, таких как Eunotia incisa ). Также было показано, что таксономическая идентификация с помощью ДНК-штрихкодирования не является точной выше уровня вида, например, для различения разновидностей (ссылка отсутствует).
Другим известным ограничением штрихкодирования для таксономической идентификации является метод кластеризации, используемый до таксономического присвоения: он часто приводит к огромной потере генетической информации, и единственным надежным способом оценки эффектов различных кластеризации и различных процессов таксономического присвоения было бы сравнение списка видов, созданного различными конвейерами при использовании одной и той же справочной базы данных. Это еще предстоит сделать для различных конвейеров, используемых при молекулярной оценке диатомовых сообществ в Европе. [16] Таксономически проверенные базы данных, которые включают доступные ваучеры, также имеют решающее значение для надежной идентификации таксонов с помощью NGS. [18]
Кроме того, часто обнаруживается, что смещение праймера является основным источником вариаций в штрихкодировании, а эффективность праймеров ПЦР может различаться у разных видов диатомовых водорослей, то есть некоторые праймеры приводят к предпочтительной амплификации одного таксона по сравнению с другим. [16]
Вывод обилия из данных метабаркодирования считается одной из самых сложных проблем в использовании окружающей среды. [19] [20] Количество сгенерированных последовательностей с помощью HTS напрямую не соответствует количеству образцов или биомассы, и разные виды могут производить разное количество прочтений (например, из-за различий в размере хлоропласта с маркером rbcL). Васселон и др. [21] недавно создали поправочный коэффициент биообъема при использовании маркера rbcL. Например, Achnanthidium minutissimum имеет небольшой биообъем и, таким образом, будет генерировать меньше копий фрагмента rbcL (расположенного в хлоропласте), чем более крупные виды. Однако этот поправочный коэффициент требует обширной калибровки с собственным биообъемом каждого вида и был протестирован только на нескольких видах. Колебания числа копий генов для других маркеров, таких как маркер 18S, по-видимому, не являются видоспецифичными, но пока не были протестированы.
Маркеры штрихкодирования обычно объединяют гипервариабельные области генома (чтобы обеспечить различие между видами) с очень консервативной областью (чтобы обеспечить специфичность к целевому организму). Несколько ДНК-маркеров, принадлежащих ядерному, митохондриальному и хлоропластному геномам ( rbcL , COI , ITS + 5.8S , SSU , 18S ...), были разработаны и успешно используются для идентификации диатомовых водорослей с помощью NGS. [22] [23] [6]
Область гена 18S широко использовалась в качестве маркера в других группах протистов [24] [25], и Ян и др. [26] были первыми, кто протестировал область гена 18S для штрихкодирования диатомовых водорослей. Циммерман и др. [7] предложили фрагмент длиной 390–410 п.н. из локуса гена рРНК 18S длиной 1800 п.н. в качестве маркера штрихкода для анализа образцов окружающей среды с помощью HTS и обсуждают его использование и ограничения для идентификации диатомовых водорослей. Этот фрагмент включает субъединицу V4, которая является самой большой и сложной из высоковариабельных областей в локусе 18S. [27] Они подчеркнули, что эта гипервариабельная область гена 18S имеет большой потенциал для изучения разнообразия протистов в больших масштабах, но имеет ограниченную эффективность для идентификации ниже уровня вида или криптических видов.
Ген rbcl используется для таксономических исследований (Trobajo et al. 2009), преимущества которых включают в себя то, что редко встречаются внутригеномные вариации, и их очень легко выравнивать и сравнивать. Справочная библиотека открытого доступа, называемая R-Syst::diatom, включает данные для двух штрихкодов (18S и rbcL). Она свободно доступна через веб-сайт. [28] Керммарек и др. [5] также успешно использовали ген rbcL для экологической оценки диатомовых водорослей. Маркер rbcL также легко выравнивается и сравнивается.
Мониц и Качмарска [23] исследовали успешность амплификации маркеров SSU, COI и ITS2 и обнаружили, что фрагмент ITS-2 + 5.8S размером 300–400 п.н. обеспечивал наивысшую частоту успешности амплификации и хорошее разрешение видов. Этот маркер впоследствии использовался для разделения морфологически определенных видов с частотой успешности 99,5%. Несмотря на эту успешность амплификации, Циммерман и др. [7] критиковали использование ITS-2 из-за внутрииндивидуальной гетерогенности. Было высказано предположение, что маркеры SSU [7] или rbcL (Mann et al., 2010) менее гетерогенны между особями и, следовательно, более полезны при различении видов.
Диатомовые водоросли обычно используются как часть набора инструментов биомониторинга, которые должны контролироваться в рамках Европейской водной рамочной директивы. [29] Диатомовые водоросли используются в качестве индикатора здоровья экосистемы в пресных водах, поскольку они повсеместно распространены, напрямую зависят от изменений физико-химических параметров и показывают лучшую связь с экологическими переменными, чем другие таксоны, например, беспозвоночные, что дает лучшую общую картину качества воды. [30]

За последние годы исследователи разработали и стандартизировали инструменты для метабаркодирования и секвенирования диатомовых водорослей, чтобы дополнить традиционную оценку с использованием микроскопии, открыв новое направление биомониторинга водных систем. [31] Использование бентосных диатомовых водорослей с помощью метода секвенирования нового поколения для речного биомониторинга выявило в нем хороший потенциал. [5] Многие исследования показали, что метабаркодирование и HTS (высокопроизводительное секвенирование) могут использоваться для оценки состояния качества и разнообразия пресных вод. В рамках Агентства по охране окружающей среды Келли и др. [32] разработали подход метабаркодирования на основе ДНК для оценки сообществ диатомовых водорослей в реках Великобритании. Васселон и др. [33] сравнили морфологический и HTS подходы для диатомовых водорослей и обнаружили, что HTS дает надежное представление о состоянии качества для большинства рек с точки зрения индекса специфической чувствительности к загрязнению (SPI). Васселон и др. [34] также применили ДНК-метабаркодирование сообществ диатомовых водорослей к сети мониторинга рек на тропическом острове Майотта (французский DOM-TOM).
Риме и др. [35] также исследовали возможность использования HTS для оценки разнообразия диатомовых водорослей и показали, что индексы разнообразия, полученные как с помощью HTS, так и с помощью микроскопического анализа, хорошо коррелируют, хотя и не идеально.
ДНК-штрихкодирование и меташтрихкодирование можно использовать для установления молекулярных показателей и индексов, которые потенциально дают выводы, в целом схожие с выводами традиционных подходов об экологическом и экологическом статусе водных экосистем. [36]
Диатомовые водоросли используются в качестве диагностического инструмента для утопления в судебно-медицинской практике. Тест на диатомовые водоросли основан на принципе вдыхания диатомовых водорослей из воды в легкие и их распределения и отложения по всему телу. Методы ДНК могут использоваться для подтверждения того, действительно ли причиной смерти было утопление, и определения причины утопления. [37] Метабаркодирование ДНК диатомовых водорослей дает возможность быстро проанализировать сообщество диатомовых водорослей, присутствующее в теле, и определить причину утопления, а также исследовать, могло ли тело быть перемещено из одного места в другое.
Метабаркодирование диатомовых водорослей может помочь в определении скрытых видов, которые трудно идентифицировать с помощью микроскопии, а также помочь в пополнении справочных баз данных путем сравнения морфологических комплексов с данными метабаркодирования. [35]
Хлорофиты обладают древней и таксономически очень разнообразной родословной (Fang et al. 2014), включая также наземные растения. Несмотря на то, что более 14 000 видов были описаны на основе структурных и ультраструктурных критериев (Hall et al. 2010), их морфологическая идентификация часто ограничена.
Для идентификации на основе ДНК было предложено несколько штрихкодов для хлорофитов, чтобы обойти проблематику морфологического. Хотя ген, кодирующий цитохромоксидазу I (COI, COX) (ссылка), является стандартным штрихкодом для животных, он оказался неудовлетворительным для хлорофитов, поскольку ген содержит несколько интронов в этой группе водорослей (Turmel et al. 2002). Для хлорофитов использовались ядерные маркерные гены: SSU rDNA, LSU rDNA, rDNA ITS (Leliaert et al. 2014). [38]
Макроводоросли — морфологическая, а не таксономическая группа — могут быть очень сложными для идентификации из-за их простой морфологии, фенотипической пластичности и альтернативных стадий жизненного цикла. Таким образом, систематика и идентификация водорослей стали в значительной степени полагаться на генетические/молекулярные инструменты, такие как ДНК-штрихкодирование . [39] [40] Ген SSU rDNA является широко используемым штрихкодом для филогенетических исследований макроводорослей. [41] Однако SSU rDNA является высококонсервативной областью и, как правило, не имеет разрешения для идентификации видов.
За последние 2 десятилетия были разработаны определенные стандарты для ДНК-штрихкодирования с целью идентификации видов для каждой из основных групп макроводорослей. [42] [39] [43] [44] [45] Ген субъединицы I цитохром -с-оксидазы (COI) обычно используется в качестве штрихкода для красных и бурых водорослей, в то время как tufA (фактор удлинения пластид), rbcL (большая субъединица рубиско) и ITS ( внутренний транскрибируемый спейсер ) обычно используются для зеленых водорослей. [41] [45] Эти штрихкоды обычно имеют длину 600-700 п.н.
Штрихкоды обычно различаются между 3 основными группами макроводорослей (красными, зелеными и бурыми), поскольку их эволюционное наследие очень разнообразно. [46] Макроводоросли являются полифилетической группой, что означает, что внутри группы они не все имеют недавнего общего предка, что затрудняет поиск гена, который сохраняется среди всех, но достаточно изменчив для идентификации вида.
Адаптировано из [40]
Подробную информацию о ДНК-штрихкодировании различных организмов можно найти здесь: