Неполная сортировка по генеалогии [1] [2] [3] , также называемая гемиплазией, глубоким слиянием , сохранением предкового полиморфизма или трансвидовым полиморфизмом, описывает явление в популяционной генетике , когда копии предковых генов не могут объединиться (оглядываясь назад во времени) в общую предковую копию до тех пор, пока не окажутся глубже предыдущих событий видообразования. Это вызвано сортировкой по генеалогии генетических полиморфизмов, которые были сохранены в последовательных узлах в видовом дереве. [4] Другими словами, дерево , созданное одним геном, отличается от дерева на уровне популяции или вида, создавая несогласованное дерево. Каким бы ни был механизм, результатом является то, что созданное дерево на уровне вида может отличаться в зависимости от выбранных генов, используемых для оценки. [5] [6] Это контрастирует с полной сортировкой по генеалогии , где дерево, созданное геном, совпадает с деревом на уровне популяции или вида. Оба являются общими результатами в филогенетическом анализе, хотя это зависит от гена, организма и метода отбора проб.

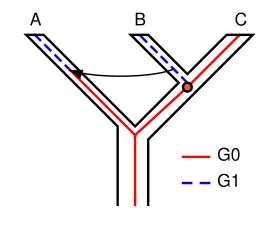
Концепция неполной сортировки по генеалогии имеет некоторые важные последствия для филогенетических методов. Сохранение полиморфизмов в различных событиях видообразования может привести к неполной сортировке по генеалогии. Предположим, что происходят два последовательных события видообразования, когда вид-предок дает начало сначала виду A, а затем видам B и C. При изучении одного гена он может иметь несколько версий ( аллелей ), что приводит к появлению разных признаков (полиморфизмов). В примере, показанном на рисунке 1, ген G имеет две версии (аллели), G0 и G1. Предок A, B и C изначально имел только одну версию гена G, G0. В какой-то момент произошла мутация, и предковая популяция стала полиморфной, причем некоторые особи имели G0, а другие G1. Когда вид A отделился, он сохранил только G1, в то время как предок B и C остался полиморфным. Когда B и C разошлись, B сохранил только G1, а C только G0; ни один из них не был теперь полиморфным в G. Дерево для гена G показывает A и B как сестер, тогда как дерево видов показывает B и C как сестер. Если филогения этих видов основана на гене G, она не будет представлять фактические отношения между видами. Другими словами, наиболее родственные виды не обязательно унаследуют наиболее родственные гены. Это, конечно, упрощенный пример неполной сортировки по линиям, и в реальных исследованиях она обычно более сложная, содержащая больше генов и видов. [7] [8]
Однако другие механизмы могут приводить к такому же кажущемуся несоответствию, например, аллели могут перемещаться через границы видов посредством гибридизации, а ДНК может передаваться между видами вирусами. [9] Это проиллюстрировано на рисунке 2. Здесь предок A, B и C, и предок B и C, имели только версию G0 гена G. Мутация произошла при расхождении B и C, и B приобрел мутировавшую версию, G1. Некоторое время спустя стрелка показывает, что G1 был передан от B к A каким-то образом (например, гибридизацией или горизонтальным переносом генов). Изучение только конечных состояний G у трех видов заставляет предположить, что A и B являются сестрами, а не B и C, как на рисунке 1, но на рисунке 2 это не вызвано неполной сортировкой линий.
Неполная сортировка по линиям имеет важные последствия для филогенетических исследований. Существует вероятность, что при создании филогенетического дерева оно может не походить на фактические отношения из-за этой неполной сортировки по линиям. Однако поток генов между линиями путем гибридизации или горизонтального переноса генов может привести к тому же конфликтующему филогенетическому дереву. Различение этих различных процессов может показаться сложным, но разрабатывается много исследований и различных статистических подходов, чтобы получить более глубокое представление об этой эволюционной динамике. [10] Одним из решений по уменьшению последствий неполной сортировки по линиям является использование нескольких генов для создания филогений видов или популяций. Чем больше генов используется, тем надежнее становится филогения. [8]
Неполная сортировка по генеалогии обычно происходит при половом размножении, поскольку вид невозможно проследить до одного человека или пары. Когда популяции племени организмов велики (т. е. тысячи), каждый ген имеет некоторое разнообразие, и генное дерево состоит из других ранее существовавших линий. Если популяция больше, эти предковые линии будут сохраняться дольше. Когда вы получаете большие предковые популяции вместе с близко расположенными по времени событиями видообразования, эти различные части ДНК сохраняют конфликтующие связи. Это затрудняет определение общего предка или точек ветвления. [5]
При изучении приматов шимпанзе и бонобо более связаны друг с другом, чем с любыми другими таксонами, и, таким образом, являются сестринскими таксонами . Тем не менее, для 1,6% генома бонобо последовательности более тесно связаны с гомологами людей, чем с шимпанзе, что, вероятно, является результатом неполной сортировки по линиям. [5] Исследование более 23 000 выравниваний последовательностей ДНК в семействе гоминидов (высшие обезьяны, включая людей) показало, что около 23% не подтверждают известную сестринскую связь шимпанзе и людей. [9]
В эволюции человека неполная сортировка по линиям используется для построения диаграммы линий гомининов, которые могли не быть отсортированы в то же время, когда видообразование произошло в доисторические времена. [11] Благодаря появлению генетического тестирования и секвенирования генома исследователи обнаружили, что генетические связи между линиями гомининов могут не соответствовать предыдущим представлениям об их родстве, основанным на физических характеристиках. [11] Более того, расхождение последнего общего предка (LCA) не обязательно может происходить в то же время, что и видообразование. [12] Сортировка по линиям — это метод, который позволяет палеоантропологам исследовать генетические связи и расхождения, которые могут не соответствовать их предыдущим моделям видообразования, основанным только на филогении . [11]
Неполная сортировка генеалогического древа человека является областью большого интереса. Существует ряд неизвестных при рассмотрении как перехода от архаичных людей к современным людям, так и расхождения других человекообразных обезьян с линией гомининов. [13]
Неполная сортировка по родословной означает, что среднее время расхождения между генами может отличаться от времени расхождения между видами. Модели предполагают, что среднее время расхождения между генами в геноме человека и шимпанзе старше, чем раскол между людьми и гориллами. Это означает, что общий предок людей и шимпанзе оставил следы генетического материала, который присутствовал у общего предка людей, шимпанзе и горилл. [12] Однако генетическое дерево несколько отличается от дерева видов или филогении. [14] В филогенетическом дереве, когда мы смотрим на эволюционные отношения между человеком, шимпанзе бонобо и гориллой, результаты показывают, что разделение бонобо и шимпанзе произошло в непосредственной близости по времени от общего предка предка бонобо-шимпанзе и людей, [12] указывая на то, что люди и шимпанзе имели общего предка в течение нескольких миллионов лет после отделения от горилл. Это создает явление, которое называется неполной сортировкой по родословной. Сегодня исследователи полагаются на фрагменты ДНК, чтобы изучить эволюционные отношения между людьми и их аналогами в надежде, что это предоставит информацию о видообразовании и предковых процессах из геномов разных типов людей. [15]

Неполная сортировка по генеалогии является общей чертой в вирусной филодинамике , где филогения, представленная передачей заболевания от одного человека к другому, то есть дерево на уровне популяции, часто не соответствует дереву, созданному в результате генетического анализа из-за узких мест популяции , которые являются неотъемлемой чертой вирусной передачи заболевания. Рисунок 3 иллюстрирует, как это может происходить. Это имеет отношение к криминальной передаче ВИЧ , где в некоторых уголовных делах филогенетический анализ одного или двух генов из штаммов обвиняемого и жертвы использовался для вывода о передаче; однако общность неполной сортировки по генеалогии означает, что передача не может быть выведена исключительно на основе такого базового анализа. [16]
Жак и Лист (2019) [17] показывают, что концепция неполной сортировки по родословной может быть применена для учета недревовидных явлений в эволюции языка. Калян и Франсуа (2019), сторонники метода исторической глоттометрии , модели, бросающей вызов применимости модели дерева в исторической лингвистике, сходятся во мнении, что «историческая глоттометрия не бросает вызов модели генеалогического древа, если принять во внимание неполную сортировку по родословной». [18]
{{cite book}}: CS1 maint: отсутствует местоположение издателя ( ссылка )