Используемая в настоящее время нотация нуклеиновых кислот была впервые формализована Международным союзом теоретической и прикладной химии (IUPAC) в 1970 году. [1] В этой общепринятой нотации используются римские символы G, C, A и T для обозначения четырех нуклеотидов. обычно встречается в дезоксирибонуклеиновых кислотах (ДНК).
Учитывая быстро растущую роль генетического секвенирования, синтеза и анализа в биологии, некоторые исследователи разработали альтернативные обозначения для дальнейшей поддержки анализа и манипулирования генетическими данными. Эти обозначения обычно используют размер, форму и симметрию для достижения этих целей.
Символы вырожденных оснований в биохимии представляют собой представление IUPAC [2] [3] положения в последовательности ДНК , которое может иметь несколько возможных альтернатив. Их не следует путать с неканоническими основаниями, поскольку каждая конкретная последовательность фактически будет иметь одно из правильных оснований. Они используются для кодирования консенсусной последовательности популяции выровненных последовательностей и используются, например, в филогенетическом анализе для суммирования в одну множественную последовательность или для поиска BLAST , даже если вырожденные символы IUPAC замаскированы (поскольку они не закодированы).
В общепринятой системе ИЮПАК азотистые основания обозначаются первыми буквами их химических названий: гуанин, цитозин, аденин и тимин. [1] Это сокращение также включает одиннадцать «неоднозначных» символов, связанных со всеми возможными комбинациями четырех оснований ДНК. [4] Символы неоднозначности были разработаны для кодирования позиционных изменений, чтобы сообщать об ошибках секвенирования ДНК , консенсусных последовательностях или однонуклеотидных полиморфизмах . Обозначения ИЮПАК, включая символы неоднозначности и предлагаемые мнемоники, показаны в таблице 1.
Несмотря на широкое и почти универсальное признание, система ИЮПАК имеет ряд ограничений, связанных с ее использованием латинского алфавита. Плохая разборчивость латинских символов в верхнем регистре, которые обычно используются при отображении генетических данных, может быть главным среди этих ограничений. Значение внешних проекций для различения букв хорошо документировано. [5] Однако эти выступы отсутствуют в заглавных буквах, которые в некоторых случаях различимы только по тонким внутренним подсказкам. Возьмем, к примеру, заглавные буквы C и G, используемые для обозначения цитозина и гуанина. Эти символы обычно составляют половину символов генетической последовательности, но отличаются небольшой внутренней галочкой (в зависимости от гарнитуры). Тем не менее, эти латинские символы доступны в наборе символов ASCII , наиболее часто используемом в текстовых сообщениях, что усиливает повсеместное распространение этой системы.
Другой недостаток нотации ИЮПАК связан с тем, что ее одиннадцать символов неоднозначности были выбраны из остальных символов латинского алфавита. Авторы обозначений постарались выделить символы неоднозначности с логической мнемоникой. Например, S используется для обозначения возможности обнаружения цитозина или гуанина в генетических локусах, оба из которых образуют сильные межцепочечные связывающие взаимодействия. И наоборот, более слабые взаимодействия тимина и аденина обозначаются буквой W. Однако удобная мнемоника не так легко доступна для других символов неоднозначности, представленных в таблице 1. Это затрудняет использование символов неоднозначности и может объяснить их ограниченное применение.
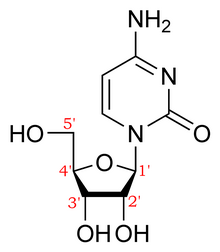
Положения атомов углерода в рибозном сахаре, который образует основу цепи нуклеиновой кислоты, пронумерованы и используются для обозначения направления нуклеиновых кислот (5'->3' против 3'->5'). Это называется направленностью . [3]
Проблемы разборчивости, связанные с генетическими данными, закодированными в ИЮПАК, побудили биологов рассмотреть альтернативные стратегии отображения генетических данных. Эти творческие подходы к визуализации последовательностей ДНК обычно основывались на использовании пространственно распределенных символов и/или визуально различных форм для кодирования длинных последовательностей нуклеиновых кислот. Были предприняты попытки использовать альтернативные обозначения нуклеотидных последовательностей, однако общее распространение было низким. Некоторые из этих подходов кратко изложены ниже.

В 1986 году Ковин и др. описал новый метод визуализации последовательности ДНК, известный как Stave Projection. [6] Их стратегия заключалась в том, чтобы закодировать нуклеотиды в виде кружков на серии горизонтальных полос, похожих на ноты на музыкальном нотном стане. Как показано на рисунке 1, каждый пробел на пятилинейном посохе соответствовал одному из четырех оснований ДНК. Пространственное распределение кругов значительно облегчило различение отдельных оснований и сравнение генетических последовательностей, чем данные, закодированные в ИЮПАК.
Порядок оснований (сверху вниз, G, A, T, C) выбирается таким, чтобы комплементарную цепь можно было прочитать, перевернув проекцию вверх ногами.
Циммерман и др. применили другой подход к визуализации генетических данных. [7] Вместо того, чтобы полагаться на пространственно распределенные круги для выделения генетических особенностей, они использовали четыре геометрически разнообразных символа, найденных в стандартном компьютерном шрифте, чтобы различать четыре основы. Авторы разработали простой макрос WordPerfect для перевода символов IUPAC в более визуально различимые символы.
С ростом доступности редакторов шрифтов Джарвиус и Ландегрен разработали новый набор генетических символов, известный как шрифт DNA Skyline, в котором для представления различных оснований ДНК используются все более высокие блоки. [8] Несмотря на то, что шрифт DNA Skyline напоминает пространственно распределенную проекцию Stave Projection Ковина и др ., он легко загружается и позволяет осуществлять перевод в нотацию IUPAC и обратно путем простого изменения шрифта в большинстве стандартных текстовых редакторов.

Амбиграммы (символы, которые передают разное значение, если смотреть в разной ориентации) были разработаны для отражения структурной симметрии, обнаруженной в двойной спирали ДНК. [9] Присвоив амбиграфические символы комплементарным основаниям (например, гуанин: b, цитозин: q, аденин: n и тимин: u), можно дополнить последовательности ДНК, просто повернув текст на 180 градусов. [10] Амбиграфическая нотация нуклеиновой кислоты также позволяет легко идентифицировать генетические палиндромы, такие как сайты рестрикции эндонуклеаз, как фрагменты текста, которые можно поворачивать на 180 градусов без изменения последовательности.
Одним из примеров амбиграфической нотации нуклеиновых кислот является AmbiScript, рационально разработанная нотация нуклеиновых кислот, сочетающая в себе многие визуальные и функциональные особенности своих предшественников. [11] В его обозначениях также используются пространственно смещенные символы для облегчения визуального просмотра и анализа генетических данных. AmbiScript также был разработан для обозначения неоднозначных положений нуклеотидов с помощью составных символов. Целью этой стратегии было предложить более интуитивное решение проблемы использования символов неоднозначности, впервые предложенное ИЮПАК. [4] Как и шрифты DNA Skyline Джарвиуса и Ландегрена, шрифты AmbiScript можно загрузить и применить к данным последовательностей, закодированных в формате IUPAC.
Пары оснований Уотсона и Крика обозначаются знаком «•», «-» или «.» (пример: A•T или поли(rC)•2поли(rC)).
Пары оснований тройной спирали Хугстина обозначаются знаком «*» или «:» (пример: C•G*G+, или T•A*T, или C•G*G, или T•A*A).