Полимеразная цепная реакция в реальном времени ( ПЦР в реальном времени или кПЦР при количественном использовании) — это лабораторный метод молекулярной биологии, основанный на полимеразной цепной реакции (ПЦР). Он контролирует амплификацию целевой молекулы ДНК во время ПЦР (т.е. в реальном времени), а не в ее конце, как в обычной ПЦР. ПЦР в реальном времени может использоваться количественно и полуколичественно (т.е. выше/ниже определенного количества молекул ДНК).
Двумя распространенными методами обнаружения продуктов ПЦР при ПЦР в реальном времени являются (1) неспецифические флуоресцентные красители , которые интеркалируются с любой двухцепочечной ДНК, и (2) специфичные для последовательности ДНК-зонды , состоящие из олигонуклеотидов , меченных флуоресцентным репортером. , что позволяет обнаружить только после гибридизации зонда с комплементарной ему последовательностью.
В рекомендациях «Минимальная информация для публикации экспериментов по количественной ПЦР в реальном времени» ( MIQE ) предлагается использовать аббревиатуру qPCR для количественной ПЦР в реальном времени и использовать RT-qPCR для обратной транскрипции – qPCR. [1] Аббревиатура «ОТ-ПЦР» обычно обозначает полимеразную цепную реакцию с обратной транскрипцией , а не ПЦР в реальном времени, но не все авторы придерживаются этого соглашения. [2]

Клетки всех организмов регулируют экспрессию генов путем обновления транскриптов гена (одноцепочечная РНК ): количество экспрессируемого гена в клетке можно измерить по количеству копий транскрипта РНК этого гена, присутствующего в образце. Чтобы надежно обнаружить и количественно оценить экспрессию генов по небольшим количествам РНК, необходима амплификация транскрипта гена. Полимеразная цепная реакция (ПЦР) — распространенный метод амплификации ДНК; Для ПЦР на основе РНК образец РНК сначала подвергается обратной транскрипции в комплементарную ДНК (кДНК) с помощью обратной транскриптазы .
Для амплификации небольших количеств ДНК используется та же методология, что и в обычной ПЦР, с использованием ДНК-матрицы, по крайней мере, одной пары специфических праймеров , дезоксирибонуклеотидтрифосфатов , подходящего буферного раствора и термостабильной ДНК-полимеразы . Вещество, маркированное флуорофором, добавляется к этой смеси в термоциклере , содержащем датчики для измерения флуоресценции флуорофора после его возбуждения на необходимой длине волны, что позволяет измерить скорость генерации одного или нескольких конкретных продуктов. Это позволяет измерять скорость образования амплифицированного продукта в каждом цикле ПЦР. Полученные таким образом данные можно проанализировать с помощью компьютерного программного обеспечения для расчета относительной экспрессии генов (или числа копий мРНК ) в нескольких образцах. Количественная ПЦР также может применяться для обнаружения и количественного определения ДНК в образцах для определения присутствия и распространенности определенной последовательности ДНК в этих образцах. [3] Это измерение проводится после каждого цикла амплификации, и именно поэтому этот метод называется ПЦР в реальном времени (то есть немедленной или одновременной ПЦР).
Количественная ПЦР и микрочипы ДНК — современные методы изучения экспрессии генов . Для измерения количества мРНК использовались более старые методы: дифференциальный дисплей , анализ защиты от РНКазы и нозерн-блоттинг . Нозерн-блоттинг часто используется для оценки уровня экспрессии гена путем визуализации количества его транскрипта мРНК в образце. В этом методе очищенную РНК отделяют электрофорезом в агарозном геле , переносят на твердую матрицу (например, нейлоновую мембрану) и исследуют с помощью специфического зонда ДНК или РНК , который комплементарен интересующему гену. Хотя этот метод все еще используется для оценки экспрессии генов, он требует относительно больших количеств РНК и дает только качественную или полуколичественную информацию об уровнях мРНК. [4] Ошибки оценки, возникающие из-за различий в методе количественного определения, могут быть результатом целостности ДНК, эффективности ферментов и многих других факторов. По этой причине был разработан ряд систем стандартизации (часто называемых методами нормализации ). Некоторые из них были разработаны для количественной оценки общей экспрессии генов, но наиболее распространенные направлены на количественную оценку конкретного изучаемого гена по отношению к другому гену, называемому нормализующим геном, который выбран из-за его почти постоянного уровня экспрессии. Эти гены часто выбирают из генов домашнего хозяйства, поскольку их функции, связанные с основным клеточным выживанием, обычно подразумевают конститутивную экспрессию генов . [5] [6] Это позволяет исследователям сообщать о соотношении экспрессии интересующих генов, разделенном на экспрессию выбранного нормализатора, тем самым позволяя сравнивать первый, не зная его абсолютного уровня экспрессии.
Наиболее часто используемые нормализующие гены — это те, которые кодируют следующие молекулы: тубулин , глицеральдегид-3-фосфатдегидрогеназа , альбумин , циклофилин и рибосомальные РНК . [4]
ПЦР в реальном времени проводится в термоциклере , способном освещать каждый образец лучом света по крайней мере одной заданной длины волны и обнаруживать флуоресценцию, испускаемую возбужденным флуорофором . Термоциклер также способен быстро нагревать и охлаждать образцы, тем самым используя преимущества физико-химических свойств нуклеиновых кислот и ДНК-полимеразы .
Процесс ПЦР обычно состоит из серии изменений температуры, которые повторяются 25–50 раз. Эти циклы обычно состоят из трех стадий: первая, при температуре около 95 ° C, позволяет разделить двойную цепь нуклеиновой кислоты; второй, при температуре около 50–60 °C, позволяет связывать праймеры с матрицей ДНК; [7] третий, при температуре от 68 до 72 ° C, облегчает полимеризацию , осуществляемую ДНК-полимеразой. Из-за небольшого размера фрагментов последний этап в этом типе ПЦР обычно опускается, поскольку фермент способен реплицировать ампликон ДНК во время перехода между стадией выравнивания и стадией денатурации. Кроме того, при четырехступенчатой ПЦР флуоресценция измеряется во время коротких температурных фаз продолжительностью всего несколько секунд в каждом цикле, с температурой, например, 80 °C, чтобы уменьшить сигнал, вызванный присутствием димеров праймера. при использовании неспецифического красителя. [8] Температуры и время, используемые для каждого цикла, зависят от широкого спектра параметров, таких как: фермент, используемый для синтеза ДНК, концентрация двухвалентных ионов и дезоксирибонуклеотидтрифосфатов (dNTP) в реакции и температура связывания ДНК. праймеры. [9]
Методику ПЦР в реальном времени можно классифицировать по химическому составу, используемому для обнаружения продукта ПЦР, специфическим или неспецифическим флуорохромам.
ДНК-связывающий краситель связывается со всей двухцепочечной (ds) ДНК в ПЦР, увеличивая квантовый выход флуоресценции красителя. Таким образом, увеличение количества продукта ДНК во время ПЦР приводит к увеличению интенсивности флуоресценции, измеряемой в каждом цикле. Однако красители дцДНК, такие как SYBR Green, будут связываться со всеми продуктами ПЦР дцДНК, включая неспецифические продукты ПЦР (такие как димер праймера ). Это потенциально может помешать или помешать точному мониторингу намеченной целевой последовательности.
При ПЦР в реальном времени с красителями дцДНК реакцию готовят как обычно, с добавлением флуоресцентного красителя дцДНК. Затем реакцию запускают в приборе для ПЦР в реальном времени , и после каждого цикла интенсивность флуоресценции измеряют с помощью детектора; краситель флуоресцирует только при связывании с дцДНК (т. е. с продуктом ПЦР). Преимущество этого метода состоит в том, что для проведения амплификации требуется только пара праймеров, что снижает затраты; несколько целевых последовательностей можно отслеживать в пробирке с использованием различных типов красителей.

Флуоресцентные репортерные зонды обнаруживают только ДНК, содержащую последовательность, комплементарную зонду; следовательно, использование репортерного зонда значительно повышает специфичность и позволяет выполнять метод даже в присутствии другой дцДНК. Используя метки разного цвета, флуоресцентные зонды можно использовать в мультиплексных анализах для мониторинга нескольких целевых последовательностей в одной пробирке. Специфичность флуоресцентных репортерных зондов также предотвращает помехи измерений, вызванные димерами праймеров , которые являются нежелательными потенциальными побочными продуктами в ПЦР. Однако флуоресцентные репортерные зонды не предотвращают ингибирующее действие димеров праймеров, что может подавлять накопление желаемых продуктов реакции.
В основе метода лежит зонд на основе ДНК с флуоресцентным репортером на одном конце и тушителем флуоресценции на противоположном конце зонда. Непосредственная близость репортера к тушителю препятствует обнаружению его флуоресценции; Разрушение зонда под действием 5'-3'- экзонуклеазной активности Taq- полимеразы нарушает близость репортера-тушителя и, таким образом, обеспечивает негашеное излучение флуоресценции, которое можно обнаружить после возбуждения лазером. Таким образом, увеличение количества продукта, на который нацелен репортерный зонд, в каждом цикле ПЦР вызывает пропорциональное увеличение флуоресценции из-за разрушения зонда и высвобождения репортера.

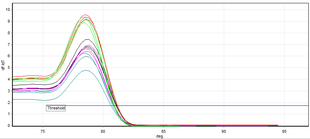
ПЦР в реальном времени позволяет идентифицировать специфические амплифицированные фрагменты ДНК, используя анализ их температуры плавления (также называемой значением T m от температуры плавления ). Используемый метод обычно представляет собой ПЦР с использованием двухцепочечных ДНК-связывающих красителей в качестве репортеров, а используемый краситель обычно представляет собой SYBR Green. Температура плавления ДНК специфична для амплифицированного фрагмента. Результаты этого метода получены путем сравнения кривых диссоциации анализируемых образцов ДНК. [11]
В отличие от обычной ПЦР, этот метод позволяет избежать предварительного использования методов электрофореза для демонстрации результатов всех образцов. Это связано с тем, что, несмотря на то, что количественная ПЦР является кинетическим методом, она обычно оценивается в определенной конечной точке. Поэтому этот метод обычно обеспечивает более быстрые результаты и/или использует меньше реагентов, чем электрофорез. Если требуется последующий электрофорез, необходимо протестировать только те образцы, которые ПЦР в реальном времени показала сомнительными, и/или подтвердить результаты для образцов, которые дали положительный результат на конкретную детерминанту.
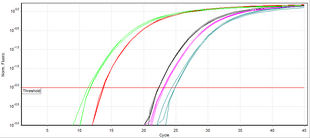
В отличие от ПЦР по конечной точке (обычной ПЦР), ПЦР в реальном времени позволяет отслеживать желаемый продукт в любой точке процесса амплификации путем измерения флуоресценции (в реальном времени производится измерение его уровня выше заданного порога). Обычно используемый метод количественного определения ДНК с помощью ПЦР в реальном времени основан на построении графика зависимости флуоресценции от количества циклов в логарифмическом масштабе . Порог обнаружения флуоресценции ДНК устанавливается в 3–5 раз больше стандартного отклонения сигнала-шума от фона. Количество циклов, при которых флуоресценция превышает порог, называется пороговым циклом (C t ) или, согласно рекомендациям MIQE, циклом количественного определения (C q ) . [1]
Во время фазы экспоненциальной амплификации количество матрицы целевой ДНК (ампликона) удваивается каждый цикл. Например, образец ДНК, C q которого предшествует C q другого образца на 3 цикла, содержал в 2 3 = 8 раз больше матрицы. Однако эффективность амплификации часто варьируется в зависимости от праймеров и матриц. Поэтому эффективность комбинации праймер-матрица оценивается в эксперименте по титрованию с серийными разведениями ДНК-матрицы для создания стандартной кривой изменения (C q ) при каждом разведении. Наклон линейной регрессии затем используется для определения эффективности амплификации, которая составляет 100% , если разбавление 1:2 приводит к разнице (C q ) , равной 1. Метод порогового цикла делает несколько предположений о механизме реакции и имеет зависимость от данных из областей с низким соотношением сигнал/шум профиля усиления, которые могут привести к существенной дисперсии во время анализа данных. [12]
Для количественной оценки экспрессии гена (C q ) РНК или ДНК интересующего гена вычитают из (C q ) РНК/ДНК гена домашнего хозяйства в том же образце, чтобы нормализовать вариации количества и качества РНК между разными образцами. Эту процедуру нормализации обычно называют ΔCt - методом [13] и она позволяет сравнивать экспрессию интересующего гена в различных образцах. Однако для такого сравнения экспрессия нормализующего эталонного гена должна быть очень схожей во всех образцах. Поэтому выбор эталонного гена, отвечающего этому критерию, имеет большое значение и часто является сложной задачей, поскольку только очень немногие гены демонстрируют одинаковые уровни экспрессии в различных состояниях или тканях. [14] [15] Хотя анализ порога цикла интегрирован во многие коммерческие программные системы, существуют более точные и надежные методы анализа данных профиля амплификации, которые следует учитывать в случаях, когда воспроизводимость является проблемой. [12]
Также были предложены методы количественной оценки qPCR, основанные на механизме, и они имеют то преимущество, что не требуют стандартной кривой для количественной оценки. Было показано, что такие методы, как MAK2 [16], имеют равную или лучшую количественную эффективность, чем методы стандартной кривой. Эти методы, основанные на механизме, используют знания о процессе амплификации полимеразы для оценки исходной концентрации образца. Расширение этого подхода включает точную модель всего профиля реакции ПЦР, что позволяет использовать данные с высоким соотношением сигнал/шум и возможность проверять качество данных перед анализом. [12]
По данным исследования Ruijter et al. [17] MAK2 предполагает постоянную эффективность амплификации во время реакции ПЦР. Однако теоретический анализ полимеразной цепной реакции, в результате которой был получен MAK2, показал, что эффективность амплификации не является постоянной на протяжении всей ПЦР. Хотя количественное определение MAK2 обеспечивает надежную оценку целевой концентрации ДНК в образце при нормальных условиях кПЦР, MAK2 не дает надежного количественного определения целевой концентрации для анализов кПЦР с использованием конкурентиметров.
Существует множество применений количественной полимеразной цепной реакции в лаборатории . Его обычно используют как для диагностики , так и для фундаментальных исследований . Использование этого метода в промышленности включает количественную оценку микробной нагрузки в пищевых продуктах или растительных материалах, обнаружение ГМО ( генетически модифицированных организмов ), а также количественную оценку и генотипирование вирусных патогенов человека.
Количественная оценка экспрессии генов традиционными методами обнаружения ДНК ненадежна. Обнаружение мРНК с помощью нозерн-блоттинга или продуктов ПЦР на геле или Саузерн-блоттинге не позволяет провести точную количественную оценку. [18] Например, за 20–40 циклов типичной ПЦР количество продукта ДНК достигает плато , которое напрямую не коррелирует с количеством целевой ДНК в исходной ПЦР. [19]
ПЦР в реальном времени можно использовать для количественного определения нуклеиновых кислот двумя распространенными методами: относительным количественным определением и абсолютным количественным определением. [20] Абсолютное количественное определение дает точное количество целевых молекул ДНК путем сравнения со стандартами ДНК с использованием калибровочной кривой . Поэтому очень важно, чтобы ПЦР образца и стандарта имели одинаковую эффективность амплификации . [21] Относительная количественная оценка основана на внутренних эталонных генах для определения кратных различий в экспрессии целевого гена. Количественная оценка выражается как изменение уровней экспрессии мРНК, интерпретируемой как комплементарная ДНК (кДНК, полученная путем обратной транскрипции мРНК). Относительную количественную оценку легче проводить, поскольку она не требует калибровочной кривой, поскольку количество исследуемого гена сравнивается с количеством контрольного эталонного гена.
Поскольку единицы, используемые для выражения результатов относительного количественного определения, не имеют значения, результаты можно сравнить с несколькими различными RTqPCR. Причиной использования одного или нескольких генов домашнего хозяйства является коррекция неспецифических вариаций, таких как различия в количестве и качестве используемой РНК, которые могут повлиять на эффективность обратной транскрипции и, следовательно, на эффективность всего процесса ПЦР. Однако наиболее важным аспектом процесса является то, что эталонный ген должен быть стабильным. [22]
Выбор этих эталонных генов традиционно проводился в молекулярной биологии с использованием качественных или полуколичественных исследований, таких как визуальный осмотр гелей РНК, нозерн-блоттинг- денситометрия или полуколичественная ПЦР (имитация ПЦР). Сейчас, в эпоху генома , можно провести более детальную оценку многих организмов с помощью транскриптомных технологий . [23] Однако исследования показали, что амплификация большинства эталонных генов, используемых для количественной оценки экспрессии мРНК, варьируется в зависимости от условий эксперимента. [24] [25] [26] Поэтому необходимо провести первоначальное статистически обоснованное методологическое исследование, чтобы выбрать наиболее подходящий эталонный ген.
Был разработан ряд статистических алгоритмов, которые позволяют определить, какой ген или гены наиболее подходят для использования в данных условиях. Такие программы, как geNORM или BestKeeper, могут сравнивать пары или средние геометрические значения матрицы различных эталонных генов и тканей . [4] [6]
Диагностическая качественная ПЦР применяется для быстрого обнаружения нуклеиновых кислот , которые являются диагностическими, например, для инфекционных заболеваний , [27] [28] рака и генетических аномалий. Внедрение качественных ПЦР-анализов в клиническую микробиологическую лабораторию значительно улучшило диагностику инфекционных заболеваний [29] и используется в качестве инструмента для выявления вновь возникающих заболеваний, таких как новые штаммы гриппа и коронавируса , [30] в диагностических тестах. . [31] [32]
Количественная ПЦР также используется микробиологами, работающими в области безопасности пищевых продуктов, порчи пищевых продуктов и ферментации, а также для оценки микробного риска качества воды (питьевых и рекреационных вод), а также в сфере охраны здоровья населения. [33]
qPCR также может использоваться для амплификации таксономических или функциональных маркеров генов в ДНК, взятых из образцов окружающей среды. [34] Маркеры представлены генетическими фрагментами ДНК или комплементарной ДНК. [34] Амплифицируя определенный генетический элемент, можно количественно определить количество элемента в образце до амплификации. [34] Использование таксономических маркеров (рибосомальных генов) и кПЦР может помочь определить количество микроорганизмов в образце и идентифицировать различные семейства, роды или виды на основе специфичности маркера. [34] Использование функциональных маркеров (генов, кодирующих белки) может показать экспрессию генов внутри сообщества, что может раскрыть информацию об окружающей среде. [34]
Сельскохозяйственная промышленность постоянно стремится производить размножение или саженцы растений, свободные от патогенов, чтобы предотвратить экономические потери и защитить здоровье. Были разработаны системы, которые позволяют обнаруживать небольшие количества ДНК Phytophthora ramorum , оомицета, убивающего дубы и другие виды, смешанного с ДНК растения-хозяина. Различение ДНК возбудителя и растения основано на амплификации последовательностей ITS, спейсеров, расположенных в кодирующей области гена рибосомальной РНК , характерных для каждого таксона. [35] Для идентификации того же патогена также были разработаны полевые версии этого метода. [36]
qPCR с использованием обратной транскрипции (RT-qPCR) может использоваться для обнаружения ГМО, учитывая его чувствительность и динамический диапазон обнаружения ДНК. Альтернативы, такие как анализ ДНК или белка, обычно менее чувствительны. Используются специфические праймеры, которые амплифицируют не трансген, а промотор , терминатор или даже промежуточные последовательности, используемые в процессе конструирования вектора. Поскольку процесс создания трансгенного растения обычно приводит к вставке более чем одной копии трансгена, его количество также обычно оценивается. Это часто осуществляется путем относительного количественного определения с использованием контрольного гена обработанного вида, который присутствует только в виде единственной копии. [37] [38]
Вирусы могут присутствовать в организме человека вследствие прямой инфекции или сопутствующих инфекций, что затрудняет диагностику с использованием классических методов и может привести к неправильному прогнозу и лечению. Использование кПЦР позволяет как количественно, так и генотипировать (характеристика штамма, проводимая с использованием кривых плавления) вируса, такого как вирус гепатита В. [39] Во многих случаях важна степень инфицирования, измеряемая количеством копий вирусного генома на единицу ткани пациента; например, вероятность реактивации вируса простого герпеса 1 типа связана с количеством инфицированных нейронов в ганглиях . [40] Эта количественная оценка проводится либо с обратной транскрипцией, либо без нее, как это происходит, если вирус интегрируется в геном человека в любой точке своего цикла, как это происходит в случае ВПЧ (вирус папилломы человека), где некоторые из его варианты связаны с появлением рака шейки матки . [41] ПЦР в реальном времени также позволила количественно определить цитомегаловирус человека (ЦМВ), который наблюдается у пациентов с иммуносупрессией после трансплантации паренхиматозных органов или костного мозга. [42]
{{cite journal}}: CS1 maint: несколько имен: список авторов ( ссылка )