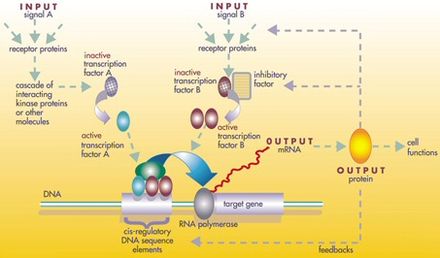
В генетике ген -регулятор , регулятор или регуляторный ген — это ген, участвующий в контроле экспрессии одного или нескольких других генов. Регуляторные последовательности , которые кодируют регуляторные гены, часто находятся на конце пяти основных пар (5') к началу транскрипции гена , который они регулируют. Кроме того, эти последовательности также могут быть обнаружены на конце трех основных пар (3') к началу транскрипции. В обоих случаях, независимо от того, находится ли регуляторная последовательность до (5') или после (3') гена, который она регулирует, последовательность часто находится на расстоянии многих килобаз от начала транскрипции . Ген-регулятор может кодировать белок или работать на уровне РНК , как в случае генов, кодирующих микроРНК . Примером гена-регулятора является ген, который кодирует белок- репрессор , который подавляет активность оператора ( ген, который связывает белки-репрессоры, таким образом подавляя трансляцию РНК в белок через РНК-полимеразу ). [1]
У прокариот гены-регуляторы часто кодируют белки-репрессоры . Белки-репрессоры связываются с операторами или промоторами , не давая РНК-полимеразе транскрибировать РНК. Обычно они постоянно экспрессируются, поэтому клетка всегда имеет запас молекул-репрессоров. [2] Индукторы заставляют белки-репрессоры менять форму или иным образом становиться неспособными связывать ДНК, позволяя РНК-полимеразе продолжать транскрипцию. Гены-регуляторы могут располагаться внутри оперона , рядом с ним или далеко от него. [3]
Другие регуляторные гены кодируют активаторные белки . Активатор связывается с сайтом на молекуле ДНК и вызывает увеличение транскрипции близлежащего гена. У прокариот хорошо известным активаторным белком является катаболитный активаторный белок (CAP), участвующий в положительном контроле lac -оперона .
В регуляции экспрессии генов , изучаемой в эволюционной биологии развития (evo-devo), важную роль играют как активаторы, так и репрессоры. [4]
Регуляторные гены также можно описать как положительные или отрицательные регуляторы, в зависимости от условий окружающей среды, окружающей клетку. Положительные регуляторы — это регуляторные элементы, которые позволяют РНК-полимеразе связываться с промоторной областью, тем самым позволяя транскрипции происходить. В терминах lac-оперона положительным регулятором будет комплекс CRP-cAMP, который должен быть связан близко к месту начала транскрипции lac-генов. Связывание этого положительного регулятора позволяет РНК-полимеразе успешно связываться с промотором последовательности гена lac, что ускоряет транскрипцию lac- генов ; lac Z, lac Y и lac A. Отрицательные регуляторы — это регуляторные элементы, которые препятствуют связыванию РНК-полимеразы с промоторной областью, тем самым подавляя транскрипцию. В терминах lac-оперона отрицательным регулятором будет lac-репрессор, который связывается с промотором в том же месте, с которым обычно связывается РНК-полимераза. Связывание lac-репрессора с сайтом связывания РНК-полимеразы ингибирует транскрипцию lac-генов. Только когда индуктор связан с lac-репрессором, сайт связывания будет свободен для РНК-полимеразы, чтобы осуществить транскрипцию lac-генов. [5] [6] [7]
Промоторы находятся в начале гена и служат местом, где собирается транскрипционный аппарат и начинается транскрипция гена. Усилители включают промоторы в определенных местах, времени и уровнях и могут быть просто определены как «промоторы промотора». Считается, что сайленсеры отключают экспрессию гена в определенные моменты времени и местах. Инсуляторы, также называемые граничными элементами, представляют собой последовательности ДНК, которые создают цис-регуляторные границы, которые не позволяют регуляторным элементам одного гена влиять на соседние гены. Общая догма заключается в том, что эти регуляторные элементы активируются путем связывания факторов транскрипции , белков, которые связываются с определенными последовательностями ДНК и контролируют транскрипцию мРНК . Может быть несколько факторов транскрипции, которые должны связываться с одним регуляторным элементом, чтобы активировать его. Кроме того, несколько других белков, называемых кофакторами транскрипции, связываются с самими факторами транскрипции, чтобы контролировать транскрипцию. [8] [9]
Отрицательные регуляторы действуют, чтобы предотвратить транскрипцию или трансляцию. Такие примеры, как cFLIP, подавляют механизмы гибели клеток, приводящие к патологическим расстройствам, таким как рак , и, таким образом, играют решающую роль в устойчивости к лекарствам . Обход таких агентов является проблемой в терапии рака . [10] Отрицательные регуляторы гибели клеток при раке включают cFLIP , семейство Bcl 2 , Survivin , HSP , IAP , NF-κB , Akt , mTOR и FADD . [10]
Существует несколько различных методов обнаружения регуляторных генов, но из многих есть несколько определенных, которые используются чаще других. Один из этих избранных называется ChIP-chip. ChIP-chip — это метод in vivo, используемый для определения геномных сайтов связывания для факторов транскрипции в двухкомпонентных регуляторах ответа системы. Анализ на основе микрочипов in vitro (DAP-chip) может использоваться для определения генных целей и функций двухкомпонентных систем передачи сигнала . Этот анализ использует тот факт, что регуляторы ответа могут быть фосфорилированы и, таким образом, активированы in vitro с использованием доноров малых молекул, таких как ацетилфосфат . [11] [12]
Филогенетический футпринтинг — это метод, который использует множественные выравнивания последовательностей для определения местоположений консервативных последовательностей, таких как регуляторные элементы. Наряду с множественными выравниваниями последовательностей, филогенетический футпринтинг также требует статистических показателей консервативных и неконсервативных последовательностей. Используя информацию, предоставленную множественными выравниваниями последовательностей и статистическими показателями, можно определить наиболее консервативные мотивы в ортологичных областях интереса. [13] [14]
{{cite book}}: |journal=проигнорировано ( помощь )