Интрон — это любая нуклеотидная последовательность внутри гена , которая не экспрессируется или не действует в конечном продукте РНК. Слово интрон происходит от термина intragenic regi on , т. е. область внутри гена. [1] Термин интрон относится как к последовательности ДНК внутри гена, так и к соответствующей последовательности РНК в транскриптах РНК . [2] Неинтронные последовательности, которые объединяются в результате этой обработки РНК для формирования зрелой РНК, называются экзонами . [3]
Интроны обнаружены в генах большинства эукариот и многих эукариотических вирусов, и они могут быть расположены как в генах, кодирующих белок, так и в генах, которые функционируют как РНК ( некодирующие гены ). Существует четыре основных типа интронов: интроны тРНК, интроны группы I, интроны группы II и сплайсосомные интроны (см. ниже). Интроны редко встречаются у бактерий и архей (прокариот).
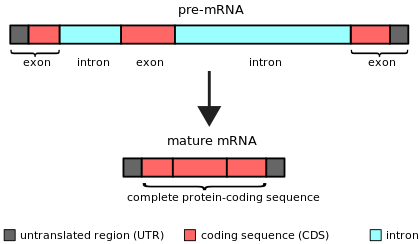
Интроны были впервые обнаружены в генах, кодирующих белок аденовируса , [4] [5] и впоследствии были идентифицированы в генах, кодирующих гены транспортной РНК и рибосомальной РНК. Сейчас известно, что интроны встречаются в самых разных генах организмов, бактерий, [6] и вирусов во всех биологических царствах.
Тот факт, что гены были разделены или прерваны интронами, был открыт независимо в 1977 году Филиппом Алленом Шарпом и Ричардом Дж. Робертсом , за что они разделили Нобелевскую премию по физиологии и медицине в 1993 году [7], хотя заслуга исследователей и сотрудников в их лабораториях, которые проводили эксперименты, приведшие к открытию, Сьюзан Бергет и Луизы Чоу , не была учтена . [8] [9] Термин «интрон» был введен американским биохимиком Уолтером Гилбертом : [1]
«Понятие цистрона [ т. е. гена] ... должно быть заменено понятием транскрипционной единицы, содержащей области, которые будут утрачены из зрелого мессенджера, – которые я предлагаю называть интронами (для внутригенных областей), – чередующиеся с областями, которые будут экспрессироваться – экзонами» (Гилберт, 1978).
Термин интрон также относится к интрацистрону , то есть дополнительному фрагменту ДНК, который возникает внутри цистрона . [10]
Хотя интроны иногда называют промежуточными последовательностями , [11] термин «промежуточная последовательность» может относиться к любому из нескольких семейств внутренних последовательностей нуклеиновых кислот, которые не присутствуют в конечном продукте гена, включая интеины , нетранслируемые области (UTR) и нуклеотиды, удаленные при редактировании РНК , в дополнение к интронам.
Частота интронов в различных геномах, как было замечено, сильно различается в спектре биологических организмов. Например, интроны чрезвычайно распространены в ядерном геноме челюстных позвоночных (например, людей, мышей и рыб-собак (фугу)), где гены, кодирующие белок, почти всегда содержат несколько интронов, в то время как интроны редки в ядерных генах некоторых эукариотических микроорганизмов, [12] например, пекарских/пивных дрожжей ( Saccharomyces cerevisiae ). Напротив, митохондриальные геномы позвоночных полностью лишены интронов, в то время как геномы эукариотических микроорганизмов могут содержать много интронов. [13]
Особенно экстремальным случаем является ген dhc7 Drosophila , содержащий интрон размером ≥3,6 мегабаз (Мб), транскрипция которого занимает примерно три дня. [14] [15] С другой стороны, исследование 2015 года показывает, что самая короткая известная длина интрона у метазоа составляет 30 пар оснований (пн), принадлежащих гену человека MST1L . [16] Самые короткие известные интроны принадлежат гетеротрихным инфузориям, таким как Stentor coeruleus , у которых большинство (> 95%) интронов имеют длину 15 или 16 пн. [17]
Сплайсинг всех интронсодержащих молекул РНК внешне схож, как описано выше. Однако, различные типы интронов были идентифицированы путем изучения структуры интрона с помощью анализа последовательности ДНК, а также генетического и биохимического анализа реакций сплайсинга РНК. Было идентифицировано по крайней мере четыре различных класса интронов:
Группа III интронов предлагается отнести к пятому семейству, но мало что известно о биохимическом аппарате, который опосредует их сплайсинг. Они, по-видимому, связаны с группой II интронов и, возможно, со сплайсосомными интронами. [18]
Ядерные пре-мРНК интроны (сплайсосомные интроны) характеризуются специфическими интронными последовательностями, расположенными на границах между интронами и экзонами. [19] Эти последовательности распознаются молекулами сплайсосомной РНК, когда инициируются реакции сплайсинга. [20] Кроме того, они содержат точку ветвления, определенную нуклеотидную последовательность вблизи 3'-конца интрона, которая становится ковалентно связанной с 5'-концом интрона во время процесса сплайсинга, образуя разветвленный ( лариат ) [ необходимо разъяснение (сложный жаргон) ] интрон. Помимо этих трех коротких консервативных элементов, ядерные пре-мРНК интронные последовательности сильно изменчивы. Ядерные пре-мРНК интроны часто намного длиннее окружающих их экзонов.
Интроны транспортной РНК, которые зависят от белков для удаления, находятся в определенном месте в антикодоновой петле несплайсированных предшественников тРНК и удаляются эндонуклеазой сплайсинга тРНК. Затем экзоны связываются вместе вторым белком, лигазой сплайсинга тРНК. [21] Обратите внимание, что самосплайсирующиеся интроны также иногда встречаются в генах тРНК. [22]
Интроны группы I и группы II обнаружены в генах, кодирующих белки ( информационная РНК ), транспортную РНК и рибосомальную РНК в очень широком диапазоне живых организмов. [23] [24] После транскрипции в РНК интроны группы I и группы II также вступают в обширные внутренние взаимодействия, которые позволяют им складываться в специфическую сложную трехмерную архитектуру . Эти сложные архитектуры позволяют некоторым интронам группы I и группы II быть самосплайсирующимися , то есть молекула РНК, содержащая интрон, может перестраивать свою собственную ковалентную структуру таким образом, чтобы точно удалить интрон и связать экзоны вместе в правильном порядке. В некоторых случаях в сплайсинге участвуют определенные интрон-связывающие белки, действуя таким образом, что они помогают интрону складываться в трехмерную структуру, необходимую для активности самосплайсинга. Интроны группы I и группы II отличаются различными наборами внутренних консервативных последовательностей и складчатых структур, а также тем фактом, что сплайсинг молекул РНК, содержащих интроны группы II, генерирует разветвленные интроны (подобно интронам сплайсосомных РНК), в то время как интроны группы I используют некодируемый гуанозиновый нуклеотид (обычно ГТФ) для инициирования сплайсинга, добавляя его к 5'-концу вырезанного интрона.
Сплайсосома — очень сложная структура, содержащая до ста белков и пять различных РНК. Субстратом реакции является длинная молекула РНК, а реакции переэтерификации, катализируемые сплайсосомой, требуют объединения участков, которые могут находиться на расстоянии тысяч нуклеотидов друг от друга. [25] [26] Все биохимические реакции связаны с известными частотами ошибок, и чем сложнее реакция, тем выше частота ошибок. Поэтому неудивительно, что реакция сплайсинга, катализируемая сплайсосомой, имеет значительную частоту ошибок, даже несмотря на то, что существуют вспомогательные факторы сплайсосомы, которые подавляют случайное расщепление скрытых участков сплайсинга. [27]
В идеальных условиях реакция сплайсинга, вероятно, будет точной на 99,999% (частота ошибок 10−5 ) , и правильные экзоны будут соединены, а правильный интрон будет удален. [28] Однако эти идеальные условия требуют очень близких совпадений с лучшими последовательностями сайтов сплайсинга и отсутствия каких-либо конкурирующих криптических последовательностей сайтов сплайсинга внутри интронов, и эти условия редко выполняются в крупных эукариотических генах, которые могут охватывать более 40 килобаз. Недавние исследования показали, что фактическая частота ошибок может быть значительно выше 10−5 и может достигать 2% или 3% ошибок (частота ошибок 2 или 3 x 10−2 ) на ген. [29] [30] [31] Дополнительные исследования показывают, что частота ошибок составляет не менее 0,1% на интрон. [32] [33] Этот относительно высокий уровень ошибок сплайсинга объясняет, почему большинство вариантов сплайсинга быстро деградируют из-за бессмысленного распада. [34] [35]
Наличие неаккуратных участков связывания в генах приводит к ошибкам сплайсинга, и может показаться странным, что эти участки не были устранены естественным отбором. Аргумент в пользу их устойчивости аналогичен аргументу в пользу мусорной ДНК. [32] [36]
Хотя мутации, которые создают или разрушают сайты связывания, могут быть немного вредными, большое количество возможных таких мутаций делает неизбежным, что некоторые из них достигнут фиксации в популяции. Это особенно актуально для видов, таких как люди, с относительно небольшими долгосрочными эффективными размерами популяции. Таким образом, вполне вероятно, что геном человека несет существенную нагрузку неоптимальных последовательностей, которые вызывают генерацию аберрантных изоформ транскриптов. В этом исследовании мы представляем прямые доказательства того, что это действительно так. [32]
Хотя каталитическая реакция может быть достаточно точной для эффективной обработки большую часть времени, общая частота ошибок может быть частично ограничена точностью транскрипции, поскольку ошибки транскрипции будут вносить мутации, которые создают криптические сайты сплайсинга. Кроме того, частота ошибок транскрипции 10−5 – 10−6 достаточно высока, чтобы один из каждых 25000 транскрибированных экзонов имел ошибку включения в одном из сайтов сплайсинга, что приведет к пропущенному интрону или пропущенному экзону. Почти все многоэкзонные гены будут производить неправильно сплайсированные транскрипты, но частота этого фонового шума будет зависеть от размера генов, количества интронов и качества последовательностей сайтов сплайсинга. [30] [33]
В некоторых случаях варианты сплайсинга будут производиться мутациями в гене (ДНК). Это могут быть полиморфизмы SNP, которые создают скрытый сайт сплайсинга или мутируют функциональный сайт. Это также могут быть мутации соматических клеток, которые влияют на сплайсинг в определенной ткани или клеточной линии. [37] [38] [39] Когда мутантный аллель находится в гетерозиготном состоянии, это приведет к образованию двух обильных вариантов сплайсинга: одного функционального и одного нефункционального. В гомозиготном состоянии мутантные аллели могут вызывать генетическое заболевание, такое как гемофилия, обнаруженная у потомков королевы Виктории, когда мутация в одном из интронов в гене фактора свертывания крови создает скрытый 3'-сайт сплайсинга, что приводит к аберрантному сплайсингу. [40] Значительная часть человеческих смертей от болезней может быть вызвана мутациями, которые мешают нормальному сплайсингу; в основном путем создания скрытых сайтов сплайсинга. [41] [38]
Неправильно сплайсированные транскрипты можно легко обнаружить, а их последовательности ввести в онлайн-базы данных. Обычно их называют «альтернативно сплайсированные» транскрипты, что может сбивать с толку, поскольку этот термин не различает реальный, биологически релевантный альтернативный сплайсинг и шум обработки из-за ошибок сплайсинга. Одним из центральных вопросов в области альтернативного сплайсинга является выявление различий между этими двумя возможностями. Многие ученые утверждали, что нулевой гипотезой должен быть шум сплайсинга, возлагая бремя доказательства на тех, кто утверждает биологически релевантный альтернативный сплайсинг. По мнению этих ученых, утверждение о функции должно сопровождаться убедительными доказательствами того, что несколько функциональных продуктов производятся из одного и того же гена. [42] [43]
Хотя интроны не кодируют белковые продукты, они являются неотъемлемой частью регуляции экспрессии генов. Некоторые интроны сами кодируют функциональные РНК посредством дальнейшей обработки после сплайсинга для получения некодирующих молекул РНК. [44] Альтернативный сплайсинг широко используется для получения нескольких белков из одного гена. Кроме того, некоторые интроны играют существенную роль в широком спектре функций регуляции экспрессии генов, таких как бессмысленный распад [45] и экспорт мРНК. [46]
После первоначального открытия интронов в генах, кодирующих белки, в эукариотическом ядре возникли серьезные споры о том, были ли интроны в современных организмах унаследованы от общего древнего предка (так называемая гипотеза ранних интронов), или же они появились в генах сравнительно недавно в эволюционном процессе (так называемая гипотеза поздних интронов). Другая теория заключается в том, что сплайсосома и интронно-экзонная структура генов являются реликтом мира РНК (гипотеза первых интронов). [47] Все еще ведутся серьезные споры о том, какая из этих гипотез наиболее верна, но на данный момент общепринятым является тот факт, что после образования первой эукариотической клетки интроны группы II из бактериального эндосимбионта вторглись в геном хозяина. Вначале эти самосплайсирующиеся интроны вырезали себя из предшественника мРНК, но со временем некоторые из них утратили эту способность, и их вырезание должно было сопровождаться транс-помощью других интронов группы II. В конце концов, ряд специфических транс-действующих интронов эволюционировали, и они стали предшественниками snRNA сплайсосомы . Эффективность сплайсинга была улучшена за счет ассоциации со стабилизирующими белками для формирования примитивной сплайсосомы. [48] [49] [50] [51]
Ранние исследования геномных последовательностей ДНК из широкого спектра организмов показывают, что интронно-экзонная структура гомологичных генов у разных организмов может сильно различаться. [52] Более поздние исследования полных эукариотических геномов показали, что длина и плотность (интроны/ген) интронов значительно различаются между родственными видами. Например, в то время как человеческий геном содержит в среднем 8,4 интрона/ген (139 418 в геноме), одноклеточный гриб Encephalitozoon cuniculi содержит всего 0,0075 интрона/ген (15 интронов в геноме). [53] Поскольку эукариоты произошли от общего предка ( общее происхождение ), в ходе эволюции должно было происходить значительное увеличение или уменьшение количества интронов. [54] [55] Считается, что этот процесс подвержен отбору, с тенденцией к приобретению интронов у более крупных видов из-за их меньшей численности популяции и наоборот у более мелких (особенно одноклеточных) видов. [56] Биологические факторы также влияют на то, какие гены в геноме теряют или накапливают интроны. [57] [58] [59]
Альтернативный сплайсинг экзонов в гене после вырезания интрона приводит к большей изменчивости последовательностей белков, транслируемых с одного гена, что позволяет генерировать несколько родственных белков из одного гена и одного предшественника транскрипта мРНК. Контроль альтернативного сплайсинга РНК осуществляется сложной сетью сигнальных молекул, которые реагируют на широкий спектр внутриклеточных и внеклеточных сигналов.
Интроны содержат несколько коротких последовательностей, которые важны для эффективного сплайсинга, такие как акцепторные и донорные сайты на обоих концах интрона, а также сайт точки ветвления, которые требуются для правильного сплайсинга сплайсосомой . Известно, что некоторые интроны усиливают экспрессию гена, в котором они содержатся, с помощью процесса, известного как интрон-опосредованное усиление (IME).
Активно транскрибируемые области ДНК часто образуют R-петли , которые уязвимы для повреждения ДНК . В высокоэкспрессируемых генах дрожжей интроны подавляют образование R-петли и возникновение повреждений ДНК. [60] Анализ всего генома как у дрожжей, так и у людей показал, что гены, содержащие интроны, имеют пониженные уровни R-петли и пониженное повреждение ДНК по сравнению с генами без интронов с аналогичной экспрессией. [60] Вставка интрона в ген, склонный к R-петле, также может подавлять образование R-петли и рекомбинацию . Бонне и др. (2017) [60] предположили, что функция интронов в поддержании генетической стабильности может объяснить их эволюционное поддержание в определенных местах, особенно в высокоэкспрессируемых генах.
Физическое присутствие интронов способствует клеточной устойчивости к голоданию посредством усиленной интронами репрессии генов рибосомных белков путей восприятия питательных веществ. [61]
Интроны могут быть потеряны или приобретены в течение эволюционного времени, как показали многие сравнительные исследования ортологичных генов. Последующие анализы выявили тысячи примеров событий потери и приобретения интронов, и было высказано предположение, что возникновение эукариот или начальные стадии эволюции эукариот включали вторжение интронов. [62] Были выявлены два окончательных механизма потери интронов: потеря интронов, опосредованная обратной транскриптазой (RTMIL), и геномные делеции, и известно, что они происходят. [63] Однако окончательные механизмы приобретения интронов остаются неуловимыми и спорными. На данный момент было описано по меньшей мере семь механизмов приобретения интронов: транспозиция интронов, вставка транспозона, тандемная геномная дупликация, перенос интронов, приобретение интронов во время репарации двухцепочечных разрывов (DSBR), вставка интрона группы II и интронизация. Теоретически, должно быть проще всего вывести происхождение недавно приобретенных интронов из-за отсутствия мутаций, вызванных хозяином, однако даже недавно приобретенные интроны не возникли ни в одном из вышеупомянутых механизмов. Таким образом, эти результаты поднимают вопрос о том, не описывают ли предложенные механизмы приобретения интронов механистическое происхождение многих новых интронов, поскольку они не являются точными механизмами приобретения интронов, или существуют другие, еще не открытые, процессы, генерирующие новые интроны. [64]
При транспозиции интронов, наиболее часто предполагаемом механизме получения интронов, считается, что сплайсированный интрон делает обратный сплайсинг либо в свою собственную мРНК, либо в другую мРНК в ранее не содержащей интронов позиции. Затем эта содержащая интрон мРНК подвергается обратной транскрипции, и полученная в результате кДНК, содержащая интрон, может затем вызвать получение интронов посредством полной или частичной рекомбинации с ее исходным геномным локусом.
Было показано, что вставки транспозонов генерируют тысячи новых интронов у различных видов эукариот. [65] Вставки транспозонов иногда приводят к дублированию этой последовательности с каждой стороны транспозона. Такая вставка может интронизировать транспозон, не нарушая кодирующую последовательность, когда транспозон вставляется в последовательность AGGT или кодирует сайты сплайсинга в последовательности транспозона. Там, где транспозоны, генерирующие интроны, не создают дупликации целевых сайтов, элементы включают оба сайта сплайсинга GT (5') и AG (3'), тем самым точно сплайсируя, не затрагивая последовательность, кодирующую белок. [65] Пока не понятно, почему эти элементы сплайсируются, случайно или в результате какого-то предпочтительного действия транспозона.
При тандемной геномной дупликации, из-за сходства между консенсусными донорными и акцепторными сайтами сплайсинга, которые оба очень похожи на AGGT, тандемная геномная дупликация экзонного сегмента, содержащего последовательность AGGT, генерирует два потенциальных сайта сплайсинга. При распознавании сплайсосомой последовательность между исходным и дублированным AGGT будет сплайсингована, что приведет к созданию интрона без изменения кодирующей последовательности гена. Репарация двухцепочечных разрывов посредством негомологичного соединения концов была недавно идентифицирована как источник прироста интронов, когда исследователи идентифицировали короткие прямые повторы, фланкирующие 43% полученных интронов у дафнии. [64] Эти числа необходимо сравнить с числом консервативных интронов, фланкированных повторами, в других организмах, однако, для статистической значимости. Для вставки интрона группы II было предложено ретрохомирование интрона группы II в ядерный ген вызвать недавний сплайсосомный прирост интронов.
Перенос интрона, как предполагалось, приводит к приобретению интрона, когда паралог или псевдоген приобретает интрон, а затем переносит этот интрон посредством рекомбинации в место, где интрон отсутствует в его сестринском паралоге. Интронизация — это процесс, посредством которого мутации создают новые интроны из ранее экзонной последовательности. Таким образом, в отличие от других предложенных механизмов приобретения интрона, этот механизм не требует вставки или генерации ДНК для создания нового интрона. [64]
Единственный предполагаемый механизм недавнего увеличения интронов, не имеющий прямых доказательств, — это вставка интронов группы II, которая при демонстрации in vivo отменяет экспрессию генов. [66] Таким образом, интроны группы II, вероятно, являются предполагаемыми предками сплайсосомных интронов, действующих как сайт-специфические ретроэлементы, и больше не отвечают за увеличение интронов. [67] [68] Тандемная геномная дупликация — единственный предложенный механизм с подтверждающими экспериментальными доказательствами in vivo: короткая внутригенная тандемная дупликация может вставить новый интрон в ген, кодирующий белок, оставляя соответствующую пептидную последовательность неизменной. [69] Этот механизм также имеет обширные косвенные доказательства, подтверждающие идею о том, что тандемная геномная дупликация является распространенным механизмом увеличения интронов. Тестирование других предложенных механизмов in vivo, в частности увеличения интронов во время DSBR, переноса интронов и интронизации, возможно, хотя эти механизмы должны быть продемонстрированы in vivo, чтобы подтвердить их как фактические механизмы увеличения интронов. Дальнейшие геномные анализы, особенно проводимые на уровне популяции, могут затем количественно оценить относительный вклад каждого механизма, возможно, выявляя видоспецифичные отклонения, которые могут пролить свет на различные скорости приобретения интронов среди разных видов. [64]
Структура:
Сращивание:
Функция
Другие: