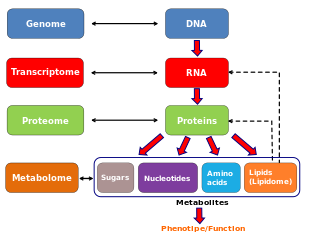
Метаболом относится к полному набору низкомолекулярных химических веществ , обнаруженных в биологическом образце. [1] Биологический образец может представлять собой клетку , клеточную органеллу , орган , ткань , экстракт ткани, биологическую жидкость или целый организм . Низкомолекулярные химические вещества, обнаруженные в данном метаболоме, могут включать как эндогенные метаболиты , которые естественным образом вырабатываются организмом (такие как аминокислоты , органические кислоты , нуклеиновые кислоты , жирные кислоты , амины , сахара , витамины , кофакторы , пигменты , антибиотики , и т. д.), а также экзогенные химические вещества (такие как лекарства, загрязнители окружающей среды , пищевые добавки , токсины и другие ксенобиотики ), которые не производятся организмом естественным путем. [2] [3]
Другими словами, существует как эндогенный метаболом, так и экзогенный метаболом. Эндогенный метаболом можно дополнительно подразделить на «первичный» и «вторичный» метаболом (особенно, когда речь идет о растительных или микробных метаболомах). Первичный метаболит принимает непосредственное участие в нормальном росте, развитии и размножении. Вторичный метаболит не принимает непосредственного участия в этих процессах, но обычно выполняет важную экологическую функцию. Вторичные метаболиты могут включать пигменты, антибиотики или отходы, полученные из частично метаболизированных ксенобиотиков. Изучение метаболома называется метаболомикой .
Слово «метаболом», по-видимому, представляет собой смесь слов «метаболит» и « хромосома ». Он был построен таким образом, чтобы подразумевать, что метаболиты косвенно кодируются генами или действуют на гены и генные продукты. Термин «метаболом» впервые был использован в 1998 году [1] [4] и, вероятно, был придуман для соответствия существующим биологическим терминам, относящимся к полному набору генов ( геному ), полному набору белков ( протеому ) и полному набору генов (геному). совокупность транскриптов ( транскриптом ). Первая книга по метаболомике была опубликована в 2003 году. [5] Первый журнал, посвященный метаболомике (названный просто «Метаболомика»), был выпущен в 2005 году, и в настоящее время его редактирует профессор Рой Гудакр . Некоторые из наиболее важных ранних работ по метаболомному анализу перечислены в ссылках ниже. [6] [7] [8] [9]
Метаболом отражает взаимодействие между геномом организма и окружающей средой. В результате метаболом организма может служить отличным зондом его фенотипа (т.е. продукта его генотипа и окружающей среды). Метаболиты можно измерить (идентифицировать, количественно или классифицировать) с использованием ряда различных технологий, включая ЯМР-спектроскопию и масс-спектрометрию . Большинство методов масс-спектрометрии (МС) необходимо сочетать с различными формами жидкостной хроматографии (ЖХ), газовой хроматографии (ГХ) или капиллярного электрофореза (КЭ), чтобы облегчить разделение соединений. Каждый метод обычно способен идентифицировать или охарактеризовать 50–5000 различных метаболитов или «особенностей» метаболитов одновременно, в зависимости от используемого инструмента или протокола. В настоящее время невозможно проанализировать весь спектр метаболитов одним аналитическим методом.
Спектроскопия ядерного магнитного резонанса (ЯМР) — это метод аналитической химии, который измеряет поглощение радиочастотного излучения определенных ядер, когда молекулы, содержащие эти ядра, помещаются в сильные магнитные поля . Частота (т.е. химический сдвиг ), с которой поглощает данный атом или ядро, сильно зависит от химического окружения (связь, ближайшие соседи по химической структуре, растворитель) этого атома в данной молекуле. Картины поглощения ЯМР создают «резонансные» пики на разных частотах или с разными химическими сдвигами – эта совокупность пиков называется спектром ЯМР . Поскольку каждое химическое соединение имеет различную химическую структуру, каждое соединение будет иметь уникальный (или почти уникальный) спектр ЯМР. В результате ЯМР особенно полезен для характеристики, идентификации и количественного определения небольших молекул, таких как метаболиты. Широкое использование ЯМР для «классических» метаболических исследований, а также его исключительная способность обрабатывать сложные смеси метаболитов, вероятно, являются причиной того, что ЯМР стал одной из первых технологий, получивших широкое распространение для рутинных измерений метаболома. Как аналитический метод ЯМР является неразрушающим, несмещенным, легко поддающимся количественному определению, практически не требует разделения, позволяет идентифицировать новые соединения и не требует химической дериватизации. ЯМР особенно пригоден для обнаружения соединений, которые менее поддаются анализу ЖХ-МС , например сахара, амины или летучие жидкости, или анализу ГХ-МС , например, большие молекулы (>500 Да) или относительно нереакционноспособные соединения. ЯМР не очень чувствительный метод с нижним пределом обнаружения около 5 мкМ. Обычно с помощью метаболомных исследований на основе ЯМР можно идентифицировать 50-150 соединений.
Масс-спектрометрия — это аналитический метод, позволяющий измерить отношение массы к заряду молекул. Молекулы или молекулярные фрагменты обычно заряжаются или ионизируются путем распыления их через заряженное поле ( ионизация электрораспылением ), бомбардировки их электронами от горячей нити накала ( электронная ионизация ) или обработки их лазером, когда они помещаются на пластины со специальным покрытием (ионизация с помощью матрицы). лазерная десорбция ионизация). Затем заряженные молекулы перемещаются в пространстве с помощью электродов или магнитов, и измеряются их скорость, степень кривизны или другие физические характеристики, чтобы определить соотношение их массы к заряду. По этим данным можно определить массу исходной молекулы. Дальнейшая фрагментация молекулы посредством контролируемых столкновений с молекулами газа или электронами может помочь определить структуру молекул. Очень точные измерения массы также можно использовать для определения элементных формул или элементного состава соединений. Большинство форм масс-спектрометрии требуют той или иной формы разделения с использованием жидкостной или газовой хроматографии . Этот этап разделения необходим для упрощения получаемых масс-спектров и обеспечения более точной идентификации соединений. Некоторые методы масс-спектрометрии также требуют, чтобы молекулы были дериватизированы или химически модифицированы, чтобы они были более пригодны для хроматографического разделения (это особенно верно для ГХ-МС). Как аналитический метод, МС является очень чувствительным методом, который требует очень небольшого количества образца (<1 нг материала или <10 мкл биожидкости) и может генерировать сигналы для тысяч метаболитов из одного образца. Приборы MS также можно настроить для проведения метаболомного анализа с очень высокой пропускной способностью (от сотен до тысяч образцов в день). Количественная оценка метаболитов и характеристика структур новых соединений с помощью МС сложнее, чем с помощью ЯМР. ЖХ-МС особенно подходит для обнаружения гидрофобных молекул ( липидов , жирных кислот) и пептидов, тогда как ГХ-МС лучше всего подходит для обнаружения небольших молекул (<500 Да) и легколетучих соединений ( эфиров , аминов, кетонов , алканов , тиолов ).
В отличие от генома или даже протеома , метаболом — это очень динамичная структура, которая может резко меняться в течение всего нескольких секунд или минут. В результате растет интерес к измерению метаболитов в течение нескольких периодов времени или в течение коротких интервалов времени с использованием модифицированных версий метаболомики на основе ЯМР или МС.
Поскольку метаболом организма во многом определяется его геномом, разные виды будут иметь разные метаболомы. Действительно, тот факт, что метаболом помидора отличается от метаболома яблока, является причиной того, почему эти два фрукта имеют такой разный вкус. Более того, разные ткани, разные органы и биожидкости, связанные с этими органами и тканями, также могут иметь совершенно разные метаболомы. Тот факт, что разные организмы и разные ткани/биожидкости имеют такие разные метаболомы, привел к разработке ряда баз данных метаболомов, специфичных для организма и биожидкости. Некоторые из наиболее известных баз данных метаболомов включают Базу данных метаболомов человека или HMDB, [10] Базу данных метаболомов дрожжей или YMDB, [11] Базу данных метаболомов E. coli или ECMDB, [12] базу данных метаболомов Arabidopsis или AraCyc [13] как а также базу данных метаболомов мочи, [14] базу данных метаболомов спинномозговой жидкости (СМЖ) [ 15 ] и базу данных метаболомов сыворотки . [16] Последние три базы данных относятся к биологическим жидкостям человека. Также существует ряд очень популярных баз данных общих метаболитов, в том числе KEGG , [17] MetaboLights, [18] База данных метаболома Голма , [19] MetaCyc , [20] LipidMaps [21] и Metlin . [22] Базы данных метаболомов можно отличить от баз данных метаболитов тем, что базы данных метаболитов содержат слегка аннотированные или синоптические данные о метаболитах от множества организмов, в то время как базы данных метаболомов содержат богато подробные и часто упоминаемые данные о химических, путях, спектрах и концентрациях метаболитов для конкретных организмов.
База данных метаболомов человека (HMDB) — это свободно доступная база данных с открытым доступом, содержащая подробные данные о более чем 40 000 метаболитах, которые уже идентифицированы или могут быть обнаружены в организме человека. HMDB содержит три вида информации:
Химические данные включают более 40 000 структур метаболитов с подробными описаниями, обширными химическими классификациями, информацией о синтезе и наблюдаемыми/расчетными химическими свойствами. Он также содержит около 10 000 экспериментально измеренных спектров ЯМР , ГХ-МС и ЖХ/МС более чем 1100 различных метаболитов. Клиническая информация включает данные о более чем 10 000 концентрациях метаболитов в биожидкостях, информацию о концентрации метаболитов при более чем 600 различных заболеваниях человека и данные о путях более чем 200 различных врожденных нарушений метаболизма. Биохимическая информация включает около 6000 последовательностей белков (и ДНК) и более 5000 биохимических реакций, связанных с этими метаболитами. HMDB поддерживает широкий спектр онлайн-запросов, включая текстовый поиск, поиск химической структуры, поиск сходства последовательностей и поиск спектрального сходства. Это делает его особенно полезным для исследователей-метаболомиков, которые пытаются идентифицировать или понять метаболиты в клинических метаболомических исследованиях. Первая версия HMDB была выпущена 1 января 2007 года и составлена учеными Университета Альберты и Университета Калгари . Тогда они сообщили данные о 2500 метаболитах, 1200 лекарствах и 3500 пищевых компонентах. С тех пор эти ученые значительно расширили коллекцию. Версия 3.5 HMDB содержит >16 000 эндогенных метаболитов, >1500 лекарств и >22 000 пищевых компонентов или пищевых метаболитов. [23]
Ученые из Университета Альберты систематически характеризуют специфические метаболомы биожидкостей, включая метаболом сыворотки, [16] метаболом мочи, [14] метаболом спинномозговой жидкости (СМЖ) [15] и метаболом слюны. Эти усилия включали как экспериментальный метаболомный анализ (с использованием ЯМР, ГХ-МС, ИСП-МС , ЖХ-МС и ВЭЖХ ), так и обширный анализ литературы. По их данным, метаболом сыворотки человека содержит не менее 4200 различных соединений (включая множество липидов), метаболом мочи человека содержит не менее 3000 различных соединений (включая сотни летучих веществ и кишечных микробных метаболитов), метаболом спинномозговой жидкости человека содержит около 500 различных соединений. соединения, в то время как метаболом слюны человека содержит около 400 различных метаболитов, включая множество бактериальных продуктов.
База данных метаболомов дрожжей — это свободно доступная онлайн-база данных, содержащая более 2000 низкомолекулярных метаболитов, обнаруженных или продуцируемых Saccharomyces cerevisiae ( пекарские дрожжи ). YMDB содержит два вида информации:
Химическая информация в YMDB включает 2027 структур метаболитов с подробными описаниями метаболитов, обширными химическими классификациями, информацией о синтезе и наблюдаемыми/расчетными химическими свойствами. Он также содержит около 4000 спектров ЯМР, ГХ-МС и ЖХ/МС, полученных от более чем 500 различных метаболитов. Биохимическая информация в YMDB включает >1100 последовательностей белков (и ДНК) и >900 биохимических реакций. YMDB поддерживает широкий спектр запросов, включая текстовый поиск, поиск химической структуры, поиск сходства последовательностей и поиск спектрального сходства. Это делает его особенно полезным для исследователей-метаболомистов, изучающих дрожжи как модельный организм или стремящихся оптимизировать производство ферментированных напитков (вина, пива).
Вторичная ионизация электрораспылением – масс-спектрометрия высокого разрешения SESI-HRMS – это неинвазивный аналитический метод, который позволяет нам контролировать метаболическую активность дрожжей. SESI-HRMS обнаружил около 300 метаболитов в процессе дрожжевого брожения, это говорит о том, что о большом количестве метаболитов глюкозы в литературе не сообщается. [24]
База данных метаболомов E. Coli — это свободно доступная онлайн-база данных, содержащая более 2700 низкомолекулярных метаболитов, обнаруженных или продуцируемых Escherichia coli (штамм E. coli K12, MG1655). ECMDB содержит два вида информации:
Химическая информация включает более 2700 структур метаболитов с подробными описаниями метаболитов, обширными химическими классификациями, информацией о синтезе и наблюдаемыми/расчетными химическими свойствами. Он также содержит около 5000 спектров ЯМР, ГХ-МС и ЖХ-МС более чем 600 различных метаболитов. Биохимическая информация включает >1600 последовательностей белков (и ДНК) и >3100 биохимических реакций, связанных с этими метаболитами. ECMDB поддерживает множество различных типов онлайн-запросов, включая текстовый поиск, поиск химической структуры, поиск сходства последовательностей и поиск спектрального сходства. Это делает его особенно полезным для исследователей-метаболомиков, изучающих E. coli в качестве модельного организма.
Вторичная ионизация электрораспылением (SESI-MS) позволяет различать одиннадцать штаммов E. Coli благодаря профилированию летучих органических соединений. [25]
В 2021 году в Интернете был опубликован первый атлас метаболомов мозга мыши и животного (млекопитающего) на разных этапах жизни. Данные различаются по областям мозга, и метаболические изменения могут быть «сопоставлены с существующими атласами генов и белков мозга». [26] [27]
Микробиота кишечника человека способствует этиологии колоректального рака через свой метаболом. [28] В частности, превращение первичных желчных кислот во вторичные желчные кислоты в результате бактериального метаболизма в толстой кишке способствует канцерогенезу . [28]