Алгоритм Смита-Уотермана выполняет локальное выравнивание последовательностей , то есть для определения схожих областей между двумя строками последовательностей нуклеиновых кислот или последовательностей белков . Вместо того, чтобы рассматривать всю последовательность, алгоритм Смита-Уотермана сравнивает сегменты всех возможных длин и оптимизирует меру сходства .
Алгоритм был впервые предложен Темплом Ф. Смитом и Майклом С. Уотерманом в 1981 году. [1] Как и алгоритм Нидлмана–Вунша , вариацией которого он является, Смит–Уотерман является алгоритмом динамического программирования . Как таковой, он обладает желаемым свойством, заключающимся в том, что он гарантированно находит оптимальное локальное выравнивание относительно используемой системы подсчета очков (которая включает матрицу подстановки и схему подсчета пробелов ). Главное отличие от алгоритма Нидлмана–Вунша заключается в том, что ячейки матрицы с отрицательными подсчетами устанавливаются в ноль. Процедура трассировки начинается с ячейки матрицы с наивысшим подсчетом очков и продолжается до тех пор, пока не встретится ячейка с нулевым подсчетом очков, что даст локальное выравнивание с наивысшим подсчетом очков. Из-за своей кубической временной сложности он часто не может быть практически применен к крупномасштабным задачам и заменяется на более эффективные в вычислительном отношении альтернативы, такие как (Gotoh, 1982), [2] ( Altschul и Erickson, 1986), [3] и (Myers и Miller, 1988). [4]
В 1970 году Сол Б. Нидлман и Кристиан Д. Вунш предложили эвристический алгоритм гомологии для выравнивания последовательностей, также называемый алгоритмом Нидлмана–Вунша. [5] Это алгоритм глобального выравнивания, который требует шагов расчета ( и являются длинами двух выравниваемых последовательностей). Он использует итерационный расчет матрицы для демонстрации глобального выравнивания. В следующем десятилетии Санкофф, [6] Райхерт, [7] Бейер [8] и другие сформулировали альтернативные эвристические алгоритмы для анализа последовательностей генов. Селлерс представил систему для измерения расстояний последовательностей. [9] В 1976 году Уотерман и др. добавили концепцию пробелов в исходную систему измерения. [10] В 1981 году Смит и Уотерман опубликовали свой алгоритм Смита–Уотермана для расчета локального выравнивания.
Алгоритм Смита-Уотермана довольно требователен к времени: для выравнивания двух последовательностей длиной и требуется время. Гото [ 2] и Альтшул [3] оптимизировали алгоритм до шагов. Сложность пространства была оптимизирована Майерсом и Миллером [4] от до (линейно), где — длина более короткой последовательности, для случая, когда требуется только одно из многих возможных оптимальных выравниваний. Позже Чоудхури, Ле и Рамачандран [11] оптимизировали производительность кэша алгоритма, сохраняя линейное использование пространства по общей длине входных последовательностей.
В последние годы геномные проекты, проводимые на различных организмах, генерировали огромные объемы данных о последовательностях генов и белков, что требует вычислительного анализа. Выравнивание последовательностей показывает связи между генами или белками, что приводит к лучшему пониманию их гомологии и функциональности. Выравнивание последовательностей также может выявить консервативные домены и мотивы .
Одной из причин локального выравнивания является сложность получения правильных выравниваний в регионах с низким сходством между отдаленно связанными биологическими последовательностями, поскольку мутации добавили слишком много «шума» за эволюционное время, чтобы позволить провести осмысленное сравнение этих регионов. Локальное выравнивание полностью избегает таких регионов и фокусируется на тех, у которых положительный балл, т. е. на тех, у которых эволюционно сохранен сигнал сходства. Предпосылкой для локального выравнивания является отрицательный балл ожидания. Балл ожидания определяется как средний балл, который система подсчета ( матрица подстановки и штрафы за пропуски ) дала бы для случайной последовательности.
Другой мотивацией для использования локальных выравниваний является то, что существует надежная статистическая модель (разработанная Карлином и Альтшулем) для оптимальных локальных выравниваний. Выравнивание неродственных последовательностей имеет тенденцию давать оптимальные оценки локального выравнивания, которые следуют распределению экстремальных значений. Это свойство позволяет программам выдавать ожидаемое значение для оптимального локального выравнивания двух последовательностей, которое является мерой того, как часто две неродственные последовательности будут давать оптимальное локальное выравнивание, оценка которого больше или равна наблюдаемой оценке. Очень низкие ожидаемые значения указывают на то, что две рассматриваемые последовательности могут быть гомологичными , то есть они могут иметь общего предка.
Пусть и — последовательности, которые необходимо выровнять, где и — длины и соответственно.
Алгоритм Смита-Уотермана выравнивает две последовательности по совпадениям/несовпадениям (также известным как замены), вставкам и удалениям. Как вставки, так и удаления являются операциями, которые вводят пробелы, которые представлены тире. Алгоритм Смита-Уотермана состоит из нескольких шагов:
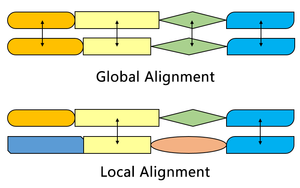
Алгоритм Смита-Уотермана находит сегменты в двух последовательностях, которые имеют сходства, в то время как алгоритм Нидлмана-Вунша выравнивает две полные последовательности. Таким образом, они служат разным целям. Оба алгоритма используют концепции матрицы подстановки, функции штрафа за пробел, матрицы подсчета очков и процесса трассировки. Три основных различия:
Одно из самых важных отличий заключается в том, что в системе оценок алгоритма Смита-Уотермана не присваивается отрицательная оценка, что позволяет проводить локальное выравнивание. Если какой-либо элемент имеет оценку ниже нуля, это означает, что последовательности до этой позиции не имеют сходства; этот элемент будет затем установлен на ноль, чтобы исключить влияние предыдущего выравнивания. Таким образом, вычисление может продолжаться для поиска выравнивания в любой позиции впоследствии.
Начальная матрица оценок алгоритма Смита–Уотермана позволяет выравнивать любой сегмент одной последовательности в произвольное положение в другой последовательности. Однако в алгоритме Нидлмана–Вунша штраф за конечный зазор также необходимо учитывать для выравнивания полных последовательностей.
Каждой замене основания или аминокислоты присваивается оценка. В общем случае совпадениям присваиваются положительные оценки, а несовпадениям присваиваются относительно более низкие оценки. Возьмем в качестве примера последовательность ДНК. Если совпадения получают +1, несовпадения получают -1, то матрица замен имеет вид:
Эту матрицу замещения можно описать следующим образом:
Различные замены оснований или замены аминокислот могут иметь разные оценки. Матрица замены аминокислот обычно сложнее, чем у оснований. См. PAM , BLOSUM .
Штраф за пробел обозначает баллы за вставку или удаление. Простая стратегия штрафа за пробел заключается в использовании фиксированного балла для каждого пробела. Однако в биологии баллы необходимо подсчитывать по-разному по практическим причинам. С одной стороны, частичное сходство между двумя последовательностями является обычным явлением; с другой стороны, одно событие мутации гена может привести к вставке одного длинного пробела. Поэтому соединенные пробелы, образующие длинный пробел, обычно более предпочтительны, чем несколько разбросанных коротких пробелов. Чтобы учесть это различие, в систему подсчета баллов были добавлены концепции открытия пробела и расширения пробела. Балл за открытие пробела обычно выше, чем балл за расширение пробела. Например, параметры по умолчанию в EMBOSS Water следующие: открытие пробела = 10, расширение пробела = 0,5.
Здесь мы обсудим две общие стратегии для штрафа за пробел. Смотрите штраф за пробел для дополнительных стратегий. Пусть будет функцией штрафа за пробел для пробела длиной :
Линейный штраф за разрыв имеет одинаковые баллы за открытие и расширение разрыва:
,
где - стоимость одного зазора.
Штраф за зазор прямо пропорционален длине зазора. При использовании линейного штрафа за зазор алгоритм Смита-Уотермана можно упростить до:
Упрощенный алгоритм использует шаги. Когда элемент оценивается, необходимо учитывать только штрафы за пробелы от элементов, которые непосредственно примыкают к этому элементу.
Аффинный штраф за разрыв учитывает открытие и расширение разрыва по отдельности:
,
где — штраф за открытие разрыва, а — штраф за расширение разрыва. Например, штраф за разрыв длиной 2 равен .
В оригинальной статье по алгоритму Смита-Уотермана использовался произвольный штраф за зазор. Он использует шаги, поэтому требует много времени. Гото оптимизировал шаги для аффинного штрафа за зазор до , [2] но оптимизированный алгоритм пытается найти только одно оптимальное выравнивание, и оптимальное выравнивание не гарантируется. [3] Альтшуль модифицировал алгоритм Гото, чтобы найти все оптимальные выравнивания, сохраняя при этом вычислительную сложность. [3] Позже Майерс и Миллер указали, что алгоритм Гото и Альтшуля можно дополнительно модифицировать на основе метода, опубликованного Хиршбергом в 1975 году, [12] и применили этот метод. [4] Алгоритм Майерса и Миллера может выровнять две последовательности, используя пространство, причем длина более короткой последовательности будет равна длине более короткой последовательности. Позже Чоудхури, Ле и Рамачандран [11] показали, как запустить алгоритм Гото с эффективностью кэширования в линейном пространстве, используя другую рекурсивную стратегию «разделяй и властвуй», чем та, которую использовал Хиршберг. Полученный алгоритм на практике работает быстрее, чем алгоритм Майерса и Миллера, благодаря более высокой производительности кэша. [11]
Возьмем в качестве примера выравнивание последовательностей TACGGGCCCGCTAC и TAGCCCTATCGGTCA . При использовании линейной функции штрафа за пробелы результат будет следующим (Выравнивания выполнены EMBOSS Water. Матрица подстановки DNAfull (оценка сходства: +5 для совпадающих символов, в противном случае -4). Открытие и расширение пробела равны 0,0 и 1,0 соответственно):
TACGGGCCCGCTA-CTA---G-CC-CTATC
При использовании аффинного штрафа за зазор результат будет следующим (открытие и расширение зазора равны 5,0 и 1,0 соответственно):
TACGGGCCCGCTATA---GCC--CTA
Этот пример показывает, что аффинный штраф за зазоры может помочь избежать разрозненных небольших зазоров.
Функция матрицы оценок заключается в проведении сравнений один к одному между всеми компонентами в двух последовательностях и записи результатов оптимального выравнивания. Процесс оценки отражает концепцию динамического программирования. Окончательное оптимальное выравнивание находится путем итеративного расширения растущего оптимального выравнивания. Другими словами, текущее оптимальное выравнивание генерируется путем решения того, какой путь (совпадение/несовпадение или вставка пробела) дает наивысшую оценку по сравнению с предыдущим оптимальным выравниванием. Размер матрицы равен длине одной последовательности плюс 1 на длину другой последовательности плюс 1. Дополнительная первая строка и первый столбец служат для выравнивания одной последовательности с любыми позициями в другой последовательности. И первая строка, и первый столбец устанавливаются на 0, чтобы конечный пробел не штрафовался. Первоначальная матрица оценок имеет вид:
Возьмем в качестве примера выравнивание последовательностей ДНК TGTTACGG и GGTTGACTA . Используйте следующую схему:
Инициализируйте и заполните матрицу подсчета очков, как показано ниже. На этом рисунке показан процесс подсчета очков первых трех элементов. Желтый цвет обозначает рассматриваемые основания. Красный цвет обозначает максимально возможный балл для оцениваемой ячейки.
Готовая матрица оценок показана ниже слева. Синий цвет показывает наивысшую оценку. Элемент может получить оценку от более чем одного элемента, каждый из которых сформирует другой путь, если этот элемент будет отслежен. В случае нескольких наивысших оценок отслежку следует выполнять, начиная с каждой наивысшей оценки. Процесс отслежки показан ниже справа. Наилучшее локальное выравнивание генерируется в обратном направлении.
Результат выравнивания:
GTT - AC GTTGAC
Реализация алгоритма Смита-Уотермана, SSEARCH, доступна в пакете анализа последовательностей FASTA из UVA FASTA Downloads. Эта реализация включает ускоренный код Altivec для процессоров PowerPC G4 и G5, который ускоряет сравнение в 10–20 раз, используя модификацию подхода Возняка, 1997, [13] и векторизацию SSE2, разработанную Фарраром [14], что делает поиск оптимальной базы данных последовательностей белков вполне практичным. Библиотека, SSW, расширяет реализацию Фаррара, чтобы возвращать информацию о выравнивании в дополнение к оптимальной оценке Смита-Уотермана. [15]
Cray продемонстрировал ускорение алгоритма Смита-Уотермана с использованием реконфигурируемой вычислительной платформы на основе чипов FPGA , с результатами, показывающими до 28-кратного ускорения по сравнению со стандартными решениями на основе микропроцессоров. Другая версия алгоритма Смита-Уотермана на основе FPGA показывает ускорение FPGA (Virtex-4) до 100 раз [16] по сравнению с процессором Opteron 2,2 ГГц. [17] Системы TimeLogic DeCypher и CodeQuest также ускоряют Smith-Waterman и Framesearch с использованием карт PCIe FPGA.
Магистерская диссертация 2011 года [18] включает анализ ускорения Смита-Уотермана на базе ПЛИС.
В публикации 2016 года OpenCL-код, скомпилированный с помощью Xilinx SDAccel, ускоряет геномное секвенирование, превосходит производительность CPU/GPU/Вт в 12-21 раз, была представлена очень эффективная реализация. При использовании одной карты PCIe FPGA, оснащенной Xilinx Virtex-7 2000T FPGA, уровень производительности на ватт был лучше, чем CPU/GPU в 12-21 раз.
Национальная лаборатория Лоуренса в Ливерморе и Объединенный институт генома Министерства энергетики США реализовали ускоренную версию локального поиска выравнивания последовательностей Смита-Уотермана с использованием графических процессоров (GPU) с предварительными результатами, показывающими 2-кратное ускорение по сравнению с программными реализациями. [19] Подобный метод уже был реализован в программном обеспечении Biofacet с 1997 года с тем же коэффициентом ускорения. [20]
Также доступны несколько реализаций алгоритма на GPU в платформе CUDA C от NVIDIA . [21] По сравнению с самой известной реализацией на CPU (использующей инструкции SIMD на архитектуре x86) от Farrar, тесты производительности этого решения с использованием одной карты NVidia GeForce 8800 GTX показывают небольшое увеличение производительности для меньших последовательностей, но небольшое снижение производительности для больших. Однако те же тесты, запущенные на двух картах NVidia GeForce 8800 GTX, почти в два раза быстрее, чем реализация Farrar для всех протестированных размеров последовательностей.
Теперь доступна более новая реализация SW на GPU CUDA, которая быстрее предыдущих версий, а также снимает ограничения на длину запросов. См. CUDASW++.
Было сообщено об одиннадцати различных реализациях ПО на базе CUDA, три из которых сообщают об ускорении в 30 раз. [22]
Наконец, другие реализации алгоритма Смита-Уотермана с ускорением на GPU можно найти в NVIDIA Parabricks , программном пакете NVIDIA для анализа генома. [23]
В 2000 году быстрая реализация алгоритма Смита-Уотермана с использованием технологии SIMD (одна инструкция, несколько данных ) , доступной в процессорах Intel Pentium MMX и аналогичной технологии, была описана в публикации Рогнеса и Сиберга. [24] В отличие от подхода Возняка (1997), новая реализация была основана на векторах, параллельных последовательности запросов, а не на диагональных векторах. Компания Sencel Bioinformatics подала заявку на патент, охватывающий этот подход. Sencel продолжает развивать программное обеспечение и предоставляет исполняемые файлы для академического использования бесплатно.
Векторизация алгоритма SSE2 (Farrar, 2007) теперь доступна, обеспечивая 8-16-кратное ускорение на процессорах Intel/AMD с расширениями SSE2. [14] При запуске на процессоре Intel с использованием микроархитектуры Core реализация SSE2 достигает 20-кратного увеличения. Реализация SSE2 Фаррара доступна как программа SSEARCH в пакете сравнения последовательностей FASTA . SSEARCH включена в набор программ поиска сходства Европейского института биоинформатики .
Датская биоинформатическая компания CLC bio достигла ускорения почти в 200 раз по сравнению со стандартными программными реализациями с SSE2 на процессоре Intel Core 2 Duo с тактовой частотой 2,17 ГГц, согласно общедоступному документу.
Ускоренная версия алгоритма Смита-Уотермана на серверах Linux на базе Intel и Advanced Micro Devices (AMD) поддерживается пакетом GenCore 6, предлагаемым Biocceleration. Тесты производительности этого программного пакета показывают ускорение скорости до 10 раз по сравнению со стандартной реализацией программного обеспечения на том же процессоре.
В настоящее время CLC bio является единственной компанией в области биоинформатики, предлагающей решения как SSE, так и FPGA, ускоряющие алгоритм Смита-Уотермана. Компания достигла ускорения более чем в 110 раз по сравнению со стандартными программными реализациями с помощью CLC Bioinformatics Cube. [ необходима цитата ]
Самую быструю реализацию алгоритма на процессорах с SSSE3 можно найти в программном обеспечении SWIPE (Rognes, 2011), [25] , которое доступно по лицензии GNU Affero General Public License . Параллельно это программное обеспечение сравнивает остатки из шестнадцати различных последовательностей базы данных с одним остатком запроса. Используя последовательность запроса из 375 остатков, была достигнута скорость 106 миллиардов обновлений ячеек в секунду (GCUPS) на двухъядерной процессорной системе Intel Xeon X5650, что более чем в шесть раз быстрее, чем программное обеспечение, основанное на «полосатом» подходе Фаррара. Оно быстрее, чем BLAST при использовании матрицы BLOSUM50.
Реализация Smith–Waterman под названием dialogsw на языках C и C++ использует наборы инструкций SIMD ( SSE4.1 для платформы x86 и AltiVec для платформы PowerPC). Она выпущена под лицензией MIT с открытым исходным кодом .
В 2008 году Фаррар [26] описал портирование Striped Smith–Waterman [14] на Cell Broadband Engine и сообщил о скоростях 32 и 12 GCUPS на блейд-сервере IBM QS20 и Sony PlayStation 3 соответственно.
Быстрое расширение генетических данных бросает вызов скорости текущих алгоритмов выравнивания последовательностей ДНК. Основные потребности в эффективном и точном методе обнаружения вариантов ДНК требуют инновационных подходов для параллельной обработки в реальном времени.
{{cite journal}}: CS1 maint: multiple names: authors list (link){{cite web}}: CS1 maint: archived copy as title (link), "Архивная копия" (PDF) . Архивировано из оригинала (PDF) 2008-07-05 . Получено 2007-10-17 .{{cite web}}: CS1 maint: archived copy as title (link), и "Архивная копия" (PDF) . Архивировано из оригинала (PDF) 2011-07-20 . Получено 2007-10-17 .{{cite web}}: CS1 maint: archived copy as title (link){{cite journal}}: Цитировать журнал требует |journal=( помощь )