Эволюционная генетика человека изучает, как один человеческий геном отличается от другого человеческого генома, эволюционное прошлое, которое дало начало человеческому геному, и его нынешние эффекты. Различия между геномами имеют антропологические , медицинские , исторические и судебно-медицинские последствия и приложения. Генетические данные могут дать важные сведения об эволюции человека .
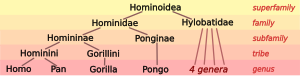
Биологи классифицируют людей , наряду с несколькими другими видами , как человекообразных обезьян (вид в семействе гоминидов ). Современные гоминиды включают два отдельных вида шимпанзе ( бонобо , Pan paniscus , и шимпанзе , Pan troglodytes ), два вида горилл ( западная горилла , Gorilla gorilla , и восточная горилла , Gorilla graueri ), и два вида орангутанов ( борнейский орангутан , Pongo pygmaeus , и суматранский орангутан , Pongo abelii ). Человекообразные обезьяны с семейством гиббонов Hylobatidae образуют надсемейство человекообразных обезьян Hominoidea .
Обезьяны, в свою очередь, принадлежат к отряду приматов (>400 видов), наряду с обезьянами Старого Света , обезьянами Нового Света и другими. Данные как митохондриальной ДНК (мтДНК), так и ядерной ДНК (яДНК) указывают на то, что приматы принадлежат к группе Euarchontoglires , вместе с Rodentia , Lagomorpha , Dermoptera и Scandentia . [1] Это дополнительно подтверждается Alu-подобными короткими вкрапленными ядерными элементами (SINE), которые были обнаружены только у членов Euarchontoglires. [2]
Филогенетическое дерево обычно выводится из последовательностей ДНК или белков популяций. Часто для изучения демографии древних людей используются митохондриальная ДНК или последовательности Y-хромосомы . Эти однолокусные источники ДНК не рекомбинируют и почти всегда наследуются от одного родителя, за исключением только одного известного исключения в мтДНК. [3] Индивиды из более близких географических регионов, как правило, более похожи, чем индивиды из более отдаленных регионов. Расстояние на филогенетическом дереве можно использовать для приблизительного указания:
Отделение людей от их ближайших родственников, нечеловеческих африканских обезьян (шимпанзе и горилл), широко изучалось более века. Были рассмотрены пять основных вопросов:
Как обсуждалось ранее, разные части генома показывают разную дивергенцию последовательностей между разными гоминоидами . Также было показано, что дивергенция последовательностей между ДНК людей и шимпанзе сильно различается. Например, дивергенция последовательностей варьируется от 0% до 2,66% между некодирующими, неповторяющимися геномными областями людей и шимпанзе. [8] Процент нуклеотидов в геноме человека (hg38), которые имели точные совпадения один к одному в геноме шимпанзе (pantro6), составил 84,38%. Кроме того, генные деревья, полученные путем сравнительного анализа сегментов ДНК, не всегда соответствуют видовому дереву. Подводя итог:
Время расхождения людей и других обезьян представляет большой интерес. Одно из первых молекулярных исследований, опубликованное в 1967 году, измеряло иммунологические расстояния (ID) между различными приматами. [10] По сути, исследование измеряло силу иммунологического ответа, который антиген одного вида (альбумин человека) вызывает в иммунной системе другого вида (человека, шимпанзе, гориллы и обезьян Старого Света ). Близкородственные виды должны иметь схожие антигены и, следовательно, более слабый иммунологический ответ на антигены друг друга. Иммунологический ответ вида на его собственные антигены (например, человека на человека) был установлен равным 1.
ID между людьми и гориллами был определен как 1,09, между людьми и шимпанзе — как 1,14. Однако расстояние до шести различных обезьян Старого Света в среднем составляло 2,46, что указывает на то, что африканские обезьяны более тесно связаны с людьми, чем с обезьянами. Авторы считают, что время расхождения между обезьянами Старого Света и гоминоидами составило 30 миллионов лет назад (MYA), основываясь на данных об ископаемых, и иммунологическое расстояние, как предполагалось, увеличивалось с постоянной скоростью. Они пришли к выводу, что время расхождения людей и африканских обезьян составляет примерно ~5 MYA. Это был удивительный результат. Большинство ученых в то время считали, что люди и высшие обезьяны разошлись гораздо раньше (>15 MYA).
Горилла была, с точки зрения идентификации, ближе к человеку, чем к шимпанзе; однако, разница была настолько незначительной, что трихотомию не удалось разрешить с уверенностью. Более поздние исследования, основанные на молекулярной генетике, смогли разрешить трихотомию: шимпанзе филогенетически ближе к человеку, чем к горилле. Однако некоторые времена расхождения, оцененные позже (с использованием гораздо более сложных методов в молекулярной генетике), существенно не отличаются от самой первой оценки в 1967 году, но недавняя статья [11] оценивает его в 11–14 млн лет назад.

Современные методы определения времени расхождения используют выравнивание последовательностей ДНК и молекулярные часы . Обычно молекулярные часы калибруются, предполагая, что орангутан отделился от африканских обезьян (включая людей) 12-16 млн лет назад. Некоторые исследования также включают некоторых обезьян Старого Света и устанавливают время их расхождения с гоминоидов в 25-30 млн лет назад. Обе точки калибровки основаны на очень небольшом количестве ископаемых данных и подвергались критике. [12]
Если эти даты будут пересмотрены, то время расхождения, оцененное по молекулярным данным, также изменится. Однако относительное время расхождения вряд ли изменится. Даже если мы не можем точно назвать абсолютное время расхождения, мы можем быть уверены, что время расхождения между шимпанзе и людьми примерно в шесть раз короче, чем между шимпанзе (или людьми) и обезьянами.
В одном исследовании (Takahata et al. , 1995) было использовано 15 последовательностей ДНК из разных областей генома человека и шимпанзе и 7 последовательностей ДНК человека, шимпанзе и гориллы . [13] Они определили, что шимпанзе более тесно связаны с людьми, чем гориллы. Используя различные статистические методы, они оценили время расхождения человек-шимпанзе в 4,7 млн лет назад, а время расхождения между гориллами и людьми (и шимпанзе) в 7,2 млн лет назад.
Кроме того, они оценили эффективную численность популяции общего предка людей и шимпанзе в ~100 000. Это было несколько удивительно, поскольку сегодня эффективная численность популяции людей оценивается всего в ~10 000. Если это правда, то это означает, что человеческая линия претерпела бы колоссальное сокращение своей эффективной численности популяции (и, следовательно, генетического разнообразия) в ходе своей эволюции. (см. Теорию катастрофы Тоба )

В другом исследовании (Chen & Li, 2001) секвенировали 53 неповторяющихся межгенных сегмента ДНК человека, шимпанзе , гориллы и орангутана . [8] Когда последовательности ДНК были объединены в одну длинную последовательность, сгенерированное дерево соседнего присоединения поддерживало кладу Homo - Pan со 100% бутстрапом (то есть, люди и шимпанзе являются наиболее близкими родственными видами из четырех). Когда три вида довольно тесно связаны друг с другом (например, человек, шимпанзе и горилла), деревья, полученные из данных о последовательностях ДНК, могут не совпадать с деревом, представляющим видообразование (дерево видов).
Чем короче межузловой промежуток времени (T IN ), тем чаще встречаются неконгруэнтные генные деревья. Эффективный размер популяции (N e ) межузловой популяции определяет, как долго генетические линии сохраняются в популяции. Более высокий эффективный размер популяции приводит к более неконгруэнтным генным деревьям. Поэтому, если известен межузловой промежуток времени, можно рассчитать предковый эффективный размер популяции общего предка человека и шимпанзе.
При индивидуальном анализе каждого сегмента 31 поддержал клад Homo - Pan , 10 поддержали клад Homo - Gorilla и 12 поддержали клад Pan - Gorilla . Используя молекулярные часы, авторы подсчитали, что гориллы разделились первыми 6,2-8,4 млн лет назад, а шимпанзе и люди разделились 1,6-2,2 млн лет спустя (межузловой временной промежуток) 4,6-6,2 млн лет назад. Межузловой временной промежуток полезен для оценки предкового эффективного размера популяции общего предка людей и шимпанзе.
Экономный анализ показал , что 24 локуса поддерживали кладу Homo - Pan , 7 поддерживали кладу Homo - Gorilla , 2 поддерживали кладу Pan - Gorilla и 20 не дали разрешения. Кроме того, они взяли 35 локусов кодирования белков из баз данных. Из них 12 поддерживали кладу Homo - Pan , 3 - кладу Homo - Gorilla , 4 - кладу Pan - Gorilla и 16 не дали разрешения. Таким образом, только ~70% из 52 локусов, которые дали разрешение (33 межгенных, 19 кодирующих белок), поддерживают «правильное» дерево видов. На основе доли локусов, которые не соответствовали видовому дереву, и рассчитанного ранее межузлового промежутка времени эффективная популяция общего предка человека и шимпанзе оценивалась примерно в 52 000–96 000 особей. Это значение не такое высокое, как в первом исследовании (Такахата), но все же намного выше современной эффективной численности популяции людей.
Третье исследование (Ян, 2002) использовало тот же набор данных, что и Чэнь и Ли, но оценило эффективную популяцию предков «всего» в ~12 000–21 000, используя другой статистический метод. [14]
Люди и шимпанзе идентичны на 99,1% на уровне кодирования, с 99,4% сходства на несинонимичном уровне и 98,4% на синонимичном уровне. [15] Выравниваемые последовательности в геномах людей и шимпанзе различаются примерно на 35 миллионов однонуклеотидных замен. Кроме того, около 3% полных геномов различаются делециями, вставками и дупликациями. [16]
Поскольку скорость мутаций относительно постоянна, примерно половина этих изменений произошла в человеческой линии. Только очень малая часть этих фиксированных различий дала начало различным фенотипам людей и шимпанзе, и их обнаружение является большой проблемой. Подавляющее большинство различий нейтральны и не влияют на фенотип . [ необходима цитата ]
Молекулярная эволюция может действовать по-разному, через эволюцию белков, потерю генов, дифференциальную регуляцию генов и эволюцию РНК. Считается, что все они сыграли определенную роль в эволюции человека.
Множество различных мутаций могут инактивировать ген, но лишь немногие изменят его функцию определенным образом. Инактивирующие мутации, таким образом, будут легко доступны для отбора, чтобы действовать на них. Таким образом, потеря гена может быть распространенным механизмом эволюционной адаптации (гипотеза «меньше-значит-больше»). [17]
80 генов были утеряны в человеческой линии после отделения от последнего общего предка с шимпанзе. 36 из них были для обонятельных рецепторов . Гены, участвующие в хеморецепции и иммунном ответе, представлены в избыточном количестве. [18] Другое исследование подсчитало, что было утеряно 86 генов. [19]
Ген кератина волос типа I был утерян в человеческой линии. Кератины являются основным компонентом волос. У людей все еще есть девять функциональных генов кератина волос типа I, но потеря этого конкретного гена могла вызвать истончение волос на теле человека. Основываясь на предположении о постоянных молекулярных часах, исследование предсказывает, что потеря гена произошла относительно недавно в эволюции человека — менее 240 000 лет назад, но и неандерталец Виндия, и высокопокрытая последовательность денисовцев содержат те же преждевременные стоп-кодоны, что и современные люди, и, следовательно, датировка должна быть более 750 000 лет назад. [20]
Стедман и др. (2004) заявили, что потеря гена саркомерного миозина MYH16 в человеческой линии привела к уменьшению жевательных мышц . Они подсчитали, что мутация, которая привела к инактивации (делеция двух пар оснований), произошла 2,4 миллиона лет назад, до появления Homo ergaster / erectus в Африке. Последующий период был отмечен сильным увеличением емкости черепа , что способствовало предположению, что потеря гена могла устранить эволюционное ограничение на размер мозга в роде Homo . [21]
По другой оценке, потеря гена MYH16 произошла 5,3 миллиона лет назад, задолго до появления Homo . [22]
Сегментные дупликации (SD или LCR ) сыграли свою роль в создании новых генов приматов и формировании генетической изменчивости человека.
Когда геном человека сравнили с геномами пяти видов приматов, включая шимпанзе , гориллу , орангутана , гиббона и макаку, было обнаружено, что существует около 20 000 специфических для человека вставок, которые считаются регуляторными. Хотя большинство вставок кажутся нейтральными с точки зрения приспособленности, небольшое количество было идентифицировано в положительно отобранных генах, показывающих ассоциации с нейронными фенотипами, а некоторые связаны с фенотипами, связанными с зубным и сенсорным восприятием. Эти результаты намекают на, по-видимому, важную роль специфических для человека вставок в недавней эволюции людей. [23]
Ускоренные человеческие регионы — это области генома, которые различаются между людьми и шимпанзе в большей степени, чем это можно объяснить генетическим дрейфом с течением времени, поскольку два вида имели общего предка. Эти регионы демонстрируют признаки того, что они подвергались естественному отбору, что привело к эволюции отчетливо человеческих черт. Два примера — HAR1F , который, как полагают, связан с развитием мозга, и HAR2 (он же HACNS1 ), который, возможно, сыграл роль в развитии противопоставленного большого пальца .
Также была выдвинута гипотеза, что большая часть различий между людьми и шимпанзе объясняется регуляцией экспрессии генов , а не различиями в самих генах. Анализы консервативных некодирующих последовательностей , которые часто содержат функциональные и, таким образом, положительно выбранные регуляторные области, рассматривают эту возможность. [24]
Когда проект последовательности генома обыкновенного шимпанзе ( Pan troglodytes ) был опубликован летом 2005 года, 2400 миллионов оснований (из ~3160 миллионов оснований) были секвенированы и собраны достаточно хорошо, чтобы их можно было сравнить с человеческим геномом. [16] 1,23% этой последовательности отличались заменами одного основания. Из них 1,06% или меньше, как считалось, представляли фиксированные различия между видами, а остальные были вариантными участками у людей или шимпанзе. Другой тип различий, называемый инделями (вставки/делеции), объяснял гораздо меньше различий (15%), но вносил ~1,5% уникальной последовательности в каждый геном, поскольку каждая вставка или делеция может включать от одного основания до миллионов оснований. [16]
В сопутствующей статье рассматривались сегментные дупликации в двух геномах, [25] вставки и делеции которых в геноме составляют большую часть последовательности инделей. Они обнаружили, что в общей сложности 2,7% эухроматической последовательности были дифференциально дуплицированы в одной или другой линии.
Расхождение последовательностей в целом имеет следующий характер: человек-шимпанзе < человек-горилла << человек-орангутанг, что подчеркивает близкое родство между людьми и африканскими обезьянами. Элементы Alu расходятся быстро из-за высокой частоты динуклеотидов CpG, которые мутируют примерно в 10 раз чаще, чем средний нуклеотид в геноме. Скорость мутаций выше в мужской зародышевой линии , поэтому расхождение в хромосоме Y , которая наследуется исключительно от отца, выше, чем в аутосомах . Хромосома X наследуется в два раза чаще через женскую зародышевую линию, чем через мужскую, и поэтому показывает немного более низкое расхождение последовательностей. Расхождение последовательностей области Xq13.3 удивительно низкое между людьми и шимпанзе. [26]
Мутации, изменяющие аминокислотную последовательность белков (K a ), встречаются реже всего. Фактически ~29% всех ортологичных белков идентичны у человека и шимпанзе. Типичный белок отличается всего двумя аминокислотами. [16] Меры расхождения последовательностей, показанные в таблице, учитывают только различия в заменах, например, от A ( аденина ) к G ( гуанину ). Однако последовательности ДНК могут также отличаться вставками и делециями ( инделами ) оснований. Обычно их удаляют из выравниваний перед выполнением расчета расхождения последовательностей.
Международная группа ученых завершила черновой вариант последовательности генома неандертальца в мае 2010 года. Результаты указывают на некоторое скрещивание между современными людьми ( Homo sapiens ) и неандертальцами ( Homo neanderthalensis ) , поскольку геномы неафриканских людей имеют на 1–4% больше общего с неандертальцами, чем геномы африканцев к югу от Сахары. Неандертальцы и большинство современных людей разделяют непереносимость лактозы вариант гена лактазы , который кодирует фермент, неспособный расщеплять лактозу в молоке после отлучения от груди. Современные люди и неандертальцы также разделяют вариант гена FOXP2, связанный с развитием мозга и речью у современных людей, что указывает на то, что неандертальцы, возможно, могли говорить. У шимпанзе есть два аминокислотных различия в FOXP2 по сравнению с человеческим и неандертальским FOXP2. [27] [28] [29]
Homo sapiens , как полагают, появился около 300 000 лет назад. Он распространился по всей Африке, а после 70 000 лет назад по всей Евразии и Океании. Исследование 2009 года выявило 14 «предковых кластеров населения», наиболее отдаленным из которых является народ Сан в Южной Африке. [30] [31]
С их быстрым распространением в различных климатических зонах, и особенно с доступностью новых источников пищи с одомашниванием крупного рогатого скота и развитием сельского хозяйства , человеческие популяции подвергались значительному селективному давлению с момента их расселения. Например, считается, что предки жителей Восточной Азии подверглись процессам отбора по ряду аллелей, включая варианты генов EDAR , ADH1B , ABCC1 и ALDH2 .
Восточноазиатские типы ADH1B, в частности, связаны с одомашниванием риса и, таким образом, могли возникнуть после развития выращивания риса примерно 10 000 лет назад. [32] Несколько фенотипических черт, характерных для жителей Восточной Азии, обусловлены единственной мутацией гена EDAR , датируемой примерно 35 000 лет назад. [33]
По состоянию на 2017 год [update]База данных полиморфизма отдельных нуклеотидов ( dbSNP ), в которой перечислены SNP и другие варианты, перечислила в общей сложности 324 миллиона вариантов, обнаруженных в секвенированных человеческих геномах. [34] Разнообразие нуклеотидов , средняя доля нуклеотидов, которые различаются между двумя особями, оценивается в пределах от 0,1% до 0,4% для современных людей (по сравнению с 2% между людьми и шимпанзе). [35] [36] Это соответствует различиям генома в нескольких миллионах участков; проект 1000 геномов аналогичным образом обнаружил, что «типичный [индивидуальный] геном отличается от референтного человеческого генома на 4,1–5,0 миллионов участков… затрагивая 20 миллионов оснований последовательности». [37]
В феврале 2019 года ученые обнаружили доказательства, основанные на генетических исследованиях с использованием искусственного интеллекта (ИИ), которые предполагают существование неизвестного вида предков человека, а не неандертальца , денисовца или гибрида человека (вроде Денни (гибридного гоминина) ), в геноме современных людей . [38] [39]
В марте 2019 года китайские ученые сообщили о введении гена MCPH1, связанного с мозгом человека , в лабораторных макак-резусов , в результате чего трансгенные обезьяны показали лучшие результаты и быстрее отвечали на «тесты на кратковременную память, включающие сопоставление цветов и форм», по сравнению с контрольными нетрансгенными обезьянами, по словам исследователей. [40] [41]
В мае 2023 года ученые сообщили, основываясь на генетических исследованиях, о более сложном пути эволюции человека, чем считалось ранее. Согласно исследованиям, люди эволюционировали из разных мест и в разное время в Африке, а не из одного места и периода времени. [42] [43]
31 августа 2023 года исследователи сообщили, основываясь на генетических исследованиях, что «бутылочное горлышко» популяции предков человека произошло «около 930 000 и 813 000 лет назад... продолжалось около 117 000 лет и привело предков человека к вымиранию». [44] [45]
{{cite journal}}: CS1 maint: numeric names: authors list (link)