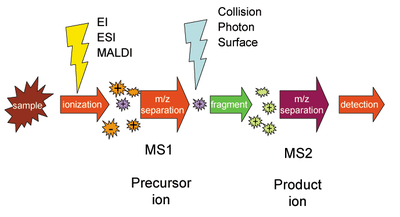
Мониторинг выбранной реакции ( SRM ), также называемый мониторингом множественной реакции ( MRM ), представляет собой метод, используемый в тандемной масс-спектрометрии , в котором ион определенной массы выбирается на первой ступени тандемного масс-спектрометра , а ионный продукт реакции фрагментации исходных ионов выбирается на второй ступени масс-спектрометра для обнаружения. [1]
Общий случай SRM можно представить следующим образом:
где ион-предшественник ABCD + выбирается первым этапом масс-спектрометрии (MS1), диссоциирует на молекулу AB и ион-продукт CD + , а последний выбирается вторым этапом масс-спектрометрии (MS2) и обнаруживается. Пара ионов-предшественника и продукта называется SRM «переходом». [2]
Последовательный мониторинг реакции ( ПМР ) представляет собой последовательное применение трех или более стадий масс-спектрометрии к ПМР, представленное в простом случае следующим образом:
где ABCD + выбирается MS1, диссоциирует на молекулу AB и ион CD + . [3] Ион выбирается на втором этапе масс-спектрометрии MS2, затем подвергается дальнейшей фрагментации с образованием иона D + , который выбирается на третьем этапе масс-спектрометрии MS3 и обнаруживается.
Мониторинг множественных реакций ( MRM ) представляет собой применение выбранного мониторинга реакций к нескольким ионам-продуктам из одного или нескольких ионов-предшественников, [3] [4] например
где ABCD + выбирается MS1 и диссоциирует двумя путями, образуя либо AB + , либо CD + . Ионы последовательно выбираются MS2 и детектируются. Мониторинг параллельных реакций ( PRM ) представляет собой применение SRM с параллельным детектированием всех переходов в одном анализе с использованием масс-спектрометра высокого разрешения. [5]
SRM может использоваться для целенаправленной количественной протеомики с помощью масс-спектрометрии . [6] После ионизации , например, в источнике электрораспыления , пептидный предшественник сначала изолируется для получения существенной популяции ионов в основном предполагаемого вида. Затем эта популяция фрагментируется для получения ионов-продуктов, чье содержание сигнала указывает на содержание пептида в образце. Этот эксперимент можно проводить на тройных квадрупольных масс-спектрометрах , где разрешающий по массе Q 1 изолирует предшественника, q 2 действует как ячейка столкновений, а разрешающий по массе Q 3 циклически проходит через ионы-продукты, которые обнаруживаются при выходе из последнего квадруполя электронным умножителем . Пару предшественник/продукт часто называют переходом . Большая работа направлена на то, чтобы обеспечить выбор переходов, имеющих максимальную специфичность.
Используя изотопную маркировку с использованием пептидов с тяжелой меткой (например, D , 13 C или 15 N ) в сложной матрице в качестве стандартов концентрации , SRM можно использовать для построения калибровочной кривой , которая может обеспечить абсолютную количественную оценку (т. е. количество копий на клетку ) нативного легкого пептида и, соответственно, его родительского белка . [2]
SRM использовался для идентификации белков, кодируемых генами дикого типа и мутантными генами ( мутантные белки ), и количественной оценки их абсолютных копий в опухолях и биологических жидкостях, тем самым отвечая на основные вопросы об абсолютном числе копий белков в одной клетке, что будет иметь важное значение при цифровом моделировании клеток млекопитающих и человеческого организма, а также об относительных уровнях генетически аномальных белков в опухолях, и доказывая свою полезность для диагностических приложений. [7] [8] SRM также использовался в качестве метода запуска сканирования полных ионов продуктов пептидов для a) подтверждения специфичности перехода SRM или b) обнаружения определенных посттрансляционных модификаций , которые находятся ниже предела обнаружения стандартных анализов MS. [9] В 2017 году SRM была разработана как высокочувствительная и воспроизводимая платформа для обнаружения целевых белков на основе масс-спектрометрии (под названием «SAFE-SRM»), и было продемонстрировано, что новый конвейер на основе SRM имеет значительные преимущества в клинических протеомных приложениях по сравнению с традиционными конвейерами SRM, и он продемонстрировал значительно улучшенную диагностическую эффективность по сравнению с методами диагностики белковых биомаркеров на основе антител, такими как ELISA . [10]