Проект 1000 геномов ( 1KGP ), который проводился с января 2008 по 2015 год, был международным исследовательским усилием по созданию самого подробного каталога генетических вариаций человека на тот момент. Ученые планировали секвенировать геномы по крайней мере тысячи анонимных здоровых участников из ряда различных этнических групп в течение следующих трех лет, используя достижения в недавно разработанных технологиях . В 2010 году проект завершил свою пилотную фазу, которая была подробно описана в публикации в журнале Nature . [1] В 2012 году секвенирование 1092 геномов было объявлено в публикации Nature . [2] В 2015 году две статьи в Nature сообщили о результатах и завершении проекта, а также о возможностях для будущих исследований. [3] [4]
Было выявлено множество редких вариаций, ограниченных близкородственными группами, и проанализировано восемь классов структурных вариаций. [5]
Проект объединил многопрофильные исследовательские группы из институтов по всему миру, включая Китай , Италию , Японию , Кению , Нигерию , Перу , Великобританию и США, которые внесли свой вклад в набор данных о последовательностях и в уточненную карту генома человека, свободно доступную через публичные базы данных как для научного сообщества, так и для широкой общественности. [2]
Международный ресурс образцов генома был создан для размещения и расширения набора данных после завершения проекта. [6]

После завершения проекта «Геном человека» достижения в области генетики популяций человека и сравнительной геномики позволили глубже понять генетическое разнообразие. [7] Понимание структурных вариаций (вставок/делеций ( инделей ), вариаций числа копий (CNV), ретроэлементов ), однонуклеотидных полиморфизмов (SNP) и естественного отбора было улучшено. [8] [9] [10] [11]
Разнообразие генетических вариаций человека, таких как индели, было обнаружено и исследовано, а также вариации генома человека [ необходима ссылка ]
Он также был направлен на предоставление доказательств, которые могут быть использованы для изучения влияния естественного отбора на различия в популяциях. Модели полиморфизмов ДНК могут быть использованы для надежного обнаружения сигнатур отбора и могут помочь идентифицировать гены, которые могут лежать в основе вариаций в устойчивости к болезням или метаболизме лекарств. [12] [13] Такие идеи могли бы улучшить понимание фенотипических вариаций , генетических нарушений и менделевского наследования и их влияния на выживание и/или воспроизводство различных человеческих популяций.
Проект «1000 геномов» был разработан для того, чтобы заполнить пробел в знаниях между редкими генетическими вариантами, которые оказывают сильное влияние преимущественно на простые признаки (например, муковисцидоз , болезнь Хантингтона ), и распространенными генетическими вариантами, которые оказывают умеренное влияние и связаны со сложными признаками (например, когнитивные способности , диабет , заболевания сердца ). [14]
Основной целью этого проекта было создание полного и подробного каталога генетических вариаций человека , который может быть использован для ассоциативных исследований, связывающих генетические вариации с заболеваниями. Консорциум стремился обнаружить >95 % вариантов (например, SNP, CNV, индели) с малыми частотами аллелей всего 1 % по всему геному и 0,1–0,5 % в генных регионах, а также оценить популяционные частоты, фоны гаплотипа и модели неравновесного сцепления аллелей вариантов. [15]
Вторичные цели включали поддержку лучшего выбора SNP и зонда для платформ генотипирования в будущих исследованиях и улучшение референтной последовательности человека . Ожидалось, что завершенная база данных станет полезным инструментом для изучения регионов, находящихся под отбором, вариаций в нескольких популяциях и понимания основных процессов мутации и рекомбинации . [15]
Геном человека состоит приблизительно из 3 миллиардов пар оснований ДНК и, по оценкам, несет около 20 000 генов , кодирующих белки . При разработке исследования консорциуму необходимо было решить несколько критических вопросов, касающихся метрик проекта, таких как технологические проблемы, стандарты качества данных и покрытие последовательностей. [15]
В течение следующих трех лет [ требуется разъяснение ] ученые из Института Сэнгера , BGI Shenzhen и Сети крупномасштабного секвенирования Национального института исследований генома человека планировали секвенировать минимум 1000 человеческих геномов. Из-за большого объема требуемых данных о последовательностях набор дополнительных участников продолжался. [14]
Почти 10 миллиардов оснований должны были быть секвенированы в день в течение двухлетней фазы производства, что эквивалентно более чем двум человеческим геномам каждые 24 часа. Предполагаемый набор данных о последовательностях должен был включать 6 триллионов оснований ДНК, в 60 раз больше данных о последовательностях, чем было опубликовано в базах данных ДНК в то время. [14]
Для определения окончательного дизайна полного проекта должны были быть проведены три пилотных исследования в течение первого года проекта. Первый пилотный проект предполагает генотипирование 180 человек из 3 основных географических групп при низком охвате (2×). Для второго пилотного исследования геномы двух нуклеарных семей (оба родителя и взрослый ребенок) будут секвенированы с глубоким охватом (20× на геном). Третье пилотное исследование включает секвенирование кодирующих областей ( экзонов ) 1000 генов у 1000 человек с глубоким охватом (20×). [14] [15]
Было подсчитано, что проект, вероятно, обойдется более чем в 500 миллионов долларов, если будут использоваться стандартные технологии секвенирования ДНК. Несколько новых технологий (например, Solexa , 454 , SOLiD ) должны были быть применены, что снизило ожидаемые затраты до 30–50 миллионов долларов. Основная поддержка будет предоставлена Wellcome Trust Sanger Institute в Хинкстоне, Англия; Beijing Genomics Institute , Шэньчжэнь (BGI Shenzhen), Китай; и NHGRI , частью Национальных институтов здравоохранения (NIH). [14]
В соответствии с принципами Форт-Лодердейла Архивировано 28 декабря 2013 г. на Wayback Machine , все данные о последовательности генома (включая вызовы вариантов) находятся в свободном доступе по мере развития проекта и могут быть загружены по ftp с веб-страницы проекта 1000 геномов.
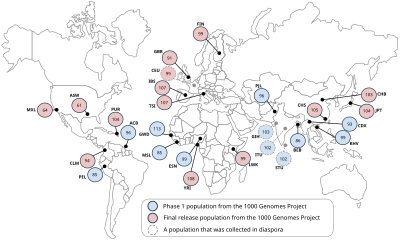
На основе общих целей проекта, образцы будут выбраны для обеспечения мощности в популяциях, где проводятся исследования ассоциаций для распространенных заболеваний. Кроме того, образцы не должны иметь медицинскую или фенотипическую информацию, поскольку предлагаемый каталог будет базовым ресурсом по человеческим вариациям. [15]
Для пилотных исследований будут секвенированы образцы человеческого генома из коллекции HapMap . Будет полезно сосредоточиться на образцах, которые имеют дополнительные доступные данные (такие как последовательность ENCODE , генотипы по всему геному, последовательность фосмидного конца, анализы структурных вариаций и экспрессия генов ), чтобы иметь возможность сравнить результаты с результатами других проектов. [15]
Соблюдая обширные этические процедуры, проект 1000 геномов затем будет использовать образцы от добровольных доноров. В исследование будут включены следующие популяции: йоруба в Ибадане (YRI), Нигерия ; японцы в Токио (JPT); китайцы в Пекине (CHB); жители Юты с происхождением из северной и западной Европы (CEU); лухья в Вебуе , Кения (LWK); масаи в Киньяве, Кения (MKK); тосканцы в Италии (TSI); перуанцы в Лиме , Перу (PEL); индейцы гуджарати в Хьюстоне (GIH); китайцы в столичном Денвере (CHD); люди мексиканского происхождения в Лос-Анджелесе (MXL); и люди африканского происхождения на юго-западе Соединенных Штатов (ASW). [14]
* Население, собранное в диаспоре
Данные, полученные в рамках проекта «1000 геномов», широко используются сообществом генетиков, что делает первый проект «1000 геномов» одной из самых цитируемых статей в биологии. [17] Для поддержки этого сообщества пользователей в июле 2012 года проект провел аналитическую встречу сообщества, на которой обсуждались ключевые открытия проекта, их влияние на популяционную генетику и исследования заболеваний человека, а также резюме других крупномасштабных исследований секвенирования. [18]
Пилотный этап состоял из трех проектов:
Было обнаружено, что в среднем каждый человек несет около 250–300 вариантов потери функции в аннотированных генах и 50–100 вариантов, ранее связанных с наследственными расстройствами. На основе двух трио подсчитано, что скорость мутации de novo зародышевой линии составляет приблизительно 10 −8 на основание на поколение. [1]