Cre-рекомбиназа — это фермент тирозиновой рекомбиназы, полученный из бактериофага P1 . Фермент использует механизм, подобный топоизомеразе I, для осуществления событий сайт-специфической рекомбинации . Фермент (38 кДа) является членом семейства интеграз сайт-специфической рекомбиназы и, как известно, катализирует событие сайт-специфической рекомбинации между двумя сайтами распознавания ДНК ( сайты LoxP ). Этот сайт распознавания loxP из 34 пар оснований (п. н.) состоит из двух палиндромных последовательностей по 13 п. н. , которые фланкируют спейсерную область из 8 п. н. Продукты Cre-опосредованной рекомбинации на сайтах loxP зависят от местоположения и относительной ориентации сайтов loxP. Два отдельных вида ДНК, оба содержащие сайты loxP, могут подвергаться слиянию в результате Cre-опосредованной рекомбинации. Последовательности ДНК, обнаруженные между двумя сайтами loxP, называются « флоксированными ». В этом случае продукты рекомбинации, опосредованной Cre, зависят от ориентации сайтов loxP. ДНК, обнаруженная между двумя сайтами loxP, ориентированными в одном направлении, будет вырезана в виде кольцевой петли ДНК, в то время как промежуточная ДНК между двумя сайтами loxP, ориентированными в противоположных направлениях, будет инвертирована. [1] Фермент не требует дополнительных кофакторов (таких как АТФ ) или вспомогательных белков для своей функции. [2]
Фермент играет важную роль в жизненном цикле бактериофага P1, например, в циклизации линейного генома и разделении димерных хромосом , которые образуются после репликации ДНК . [3]
Рекомбиназа Cre является широко используемым инструментом в области молекулярной биологии . Уникальная и специфическая система рекомбинации фермента используется для манипулирования генами и хромосомами в огромном диапазоне исследований, таких как исследования по выключению или включению генов . Способность фермента эффективно работать в широком диапазоне клеточных сред (включая млекопитающих, растения, бактерии и дрожжи) позволяет использовать систему рекомбинации Cre-Lox в огромном количестве организмов, что делает ее особенно полезным инструментом в научных исследованиях. [4]
Исследования, проведенные в 1981 году Штернбергом и Гамильтоном, продемонстрировали, что бактериофаг « P1 » имел уникальную систему сайт-специфической рекомбинации. Фрагменты EcoRI генома бактериофага P1 были получены и клонированы в лямбда-векторы . Было обнаружено, что фрагмент EcoRI размером 6,5 кб (фрагмент 7) допускает эффективные события рекомбинации. [5] Механизм этих событий рекомбинации был известен как уникальный, поскольку они происходили в отсутствие бактериальных белков RecA и RecBCD . Компоненты этой системы рекомбинации были выяснены с помощью исследований делеционного мутагенеза . Эти исследования показали, что для эффективных событий рекомбинации требуются как продукт гена P1, так и сайт рекомбинации. Продукт гена P1 был назван Cre ( c yclization re combination), а сайт рекомбинации был назван loxP ( locus of crossing ( x ) over, P 1). [5] Белок Cre был очищен в 1983 году и, как было установлено, представляет собой белок массой 35 000 Да. [2] Для рекомбиназной активности очищенного белка не требуются высокоэнергетические кофакторы, такие как АТФ или вспомогательные белки. [2] Ранние исследования также продемонстрировали, что Cre связывается с неспецифическими последовательностями ДНК, имея при этом в 20 раз более высокую аффинность к последовательностям loxP, а результаты ранних исследований футпринтинга ДНК также предполагают, что молекулы Cre связывают сайты loxP как димеры . [2]
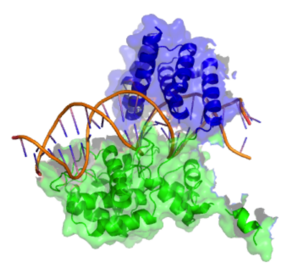

Рекомбиназа Cre состоит из 343 аминокислот , которые образуют два отдельных домена. Аминоконцевой домен охватывает остатки 20–129, и этот домен содержит 5 альфа-спиральных сегментов, связанных серией коротких петель. Спирали A и E участвуют в образовании тетрамера рекомбиназы, при этом известно, что C-концевая область спирали E образует контакты с C-концевым доменом соседних субъединиц. Спирали B и D образуют прямые контакты с большой бороздкой ДНК loxP. Считается, что эти две спирали образуют три прямых контакта с основаниями ДНК на участке loxP. Карбоксиконцевой домен фермента состоит из аминокислот 132–341 и содержит активный центр фермента. Общая структура этого домена имеет большое структурное сходство с каталитическим доменом других ферментов того же семейства, таких как λ-интеграза и HP1-интеграза. Этот домен преимущественно спиральный по структуре с 9 отдельными спиралями (F−N). Концевая спираль (N) выступает из основного тела карбокси-домена, и эта спираль, как полагают, играет роль в посредничестве взаимодействий с другими субъединицами. Кристаллические структуры показывают, что эта конечная спираль N зарывает свою гидрофобную поверхность в акцепторный карман соседней субъединицы Cre. [6]
Эффект двухдоменной структуры заключается в формировании С-образного зажима, который захватывает ДНК с противоположных сторон. [3]

Активный центр фермента Cre состоит из консервативных остатков каталитической триады Arg 173, His 289, Arg 292, а также консервативных нуклеофильных остатков Tyr 324 и Trp 315. В отличие от некоторых ферментов рекомбиназы, таких как рекомбиназа Flp, Cre не образует общего активного центра между отдельными субъединицами, и все остатки, которые вносят вклад в активный центр, находятся на одной субъединице. Следовательно, когда две молекулы Cre связываются с одним сайтом loxP, присутствуют два активных центра. Рекомбинация, опосредованная Cre, требует образования синапса, в котором два комплекса Cre-LoxP ассоциируются, образуя то, что известно как тетрамер синапса, в котором присутствуют 4 различных активных центра. [6] Tyr 324 действует как нуклеофил , образуя ковалентную 3'-фосфотирозиновую связь с субстратом ДНК. Разрезной фосфат (фосфат, нацеленный на нуклеофильную атаку в месте расщепления) координируется боковыми цепями 3 аминокислотных остатков каталитической триады ( Arg 173, His 289 и Trp 315). Азот индола триптофана 315 также образует водородную связь с этим разрезным фосфатом. (примечание: гистидин занимает этот сайт в других членах семейства тирозиновых рекомбиназ и выполняет ту же функцию). Эта реакция расщепляет ДНК и освобождает 5'-гидроксильную группу. Этот процесс происходит в активном центре двух из четырех субъединиц рекомбиназы, присутствующих в тетрамере синапса. Если 5'-гидроксильные группы атакуют 3'-фосфотирозиновую связь, одна пара цепей ДНК обменяет свои действия, образуя промежуточное соединение Холлидея . [3]
Рекомбиназа Cre играет важную роль в жизненном цикле бактериофага P1 . При инфицировании клетки система Cre-loxP используется для того, чтобы вызвать кольцевание ДНК P1. В дополнение к этому Cre также используется для разрешения димерной лизогенной ДНК P1, которая образуется во время деления клетки фага. [7]
Индуцируемая активация Cre достигается с помощью варианта CreER (рецептор эстрогена), который активируется только после доставки тамоксифена . [8] Это делается посредством слияния мутировавшего домена связывания лиганда рецептора эстрогена с рекомбиназой Cre, в результате чего Cre становится специфически активированным тамоксифеном. В отсутствие тамоксифена CreER приведет к перемещению мутировавшей рекомбиназы в цитоплазму. Белок останется в этом месте в своем неактивированном состоянии до тех пор, пока не будет дан тамоксифен. После введения тамоксифена он метаболизируется в 4-гидрокситамоксифен, который затем связывается с ER и приводит к транслокации CreER в ядро, где он затем может расщеплять сайты lox. [9] Важно отметить, что иногда флуоресцентные репортеры могут активироваться в отсутствие тамоксифена из-за утечки нескольких молекул рекомбиназы Cre в ядро, что в сочетании с очень чувствительными репортерами приводит к непреднамеренной маркировке клеток. [10] CreER(T2) был разработан для минимизации тамоксифен-независимой рекомбинации и максимизации чувствительности к тамоксифену.
В последние годы рекомбиназа Cre была улучшена путем преобразования в предпочтительные кодоны млекопитающих , удаления известных криптических сайтов сплайсинга , измененного стоп-кодона и снижения содержания CpG для снижения риска эпигенетического молчания у млекопитающих . [11] Также был выявлен ряд мутантов с повышенной точностью. [12]