Технологии сайт-специфической рекомбиназы представляют собой инструменты генной инженерии , которые используют ферменты рекомбиназы для замены целевых участков ДНК.
В конце 1980-х годов генное таргетирование в эмбриональных стволовых клетках мышей (ЭСК) позволило передавать мутации в зародышевую линию мышей и стало новым вариантом для изучения генетической основы регуляторных сетей, существующих в геноме. Тем не менее, классическое генное таргетирование оказалось ограниченным в нескольких отношениях, поскольку функции генов необратимо разрушались геном-маркером, который должен был быть введен для отбора рекомбинантных ЭСК. Эти ранние шаги привели к появлению животных, у которых мутация присутствовала во всех клетках организма с самого начала, что приводило к сложным фенотипам и/или ранней летальности. Была очевидная необходимость в методах, ограничивающих эти мутации определенными точками развития и определенными типами клеток. Эта мечта стала реальностью, когда группы в США смогли ввести бактериофаг и дрожжевые системы сайт-специфической рекомбинации (SSR-) в клетки млекопитающих, а также в клетки мышей. [1] [2] [3]
Обычные стратегии генной инженерии требуют постоянной модификации целевого генома. Для этого необходимо вложить большие усилия в разработку маршрутов, применяемых для доставки трансгенов. Хотя для биотехнологических целей случайная интеграция все еще распространена, она может привести к непредсказуемой экспрессии генов из-за переменного числа копий трансгена, отсутствия контроля над сайтами интеграции и связанными с ними мутациями. Молекулярные требования в области стволовых клеток гораздо более строгие. Здесь гомологичная рекомбинация (HR) может, в принципе, обеспечить специфичность процесса интеграции, но для эукариот она скомпрометирована крайне низкой эффективностью. Хотя мегануклеазы, эффекторные нуклеазы, подобные цинковым пальцам и активаторам транскрипции (ZFN и TALEN), являются фактическими инструментами, поддерживающими HR, именно доступность сайт-специфичных рекомбиназ (SSR) послужила толчком к рациональному построению линий клеток с предсказуемыми свойствами. В настоящее время обе технологии, HR и SSR, можно объединить в высокоэффективные «технологии тегирования и обмена». [4]
Было идентифицировано множество систем сайт-специфической рекомбинации для выполнения этих перестроек ДНК для различных целей, но почти все они принадлежат к одному из двух семейств, тирозиновых рекомбиназ (YR) и сериновых рекомбиназ (SR), в зависимости от их механизма . Эти два семейства могут опосредовать до трех типов перестроек ДНК (интеграция, вырезание/разрешение и инверсия) по различным маршрутам реакции в зависимости от их происхождения и архитектуры. [5]
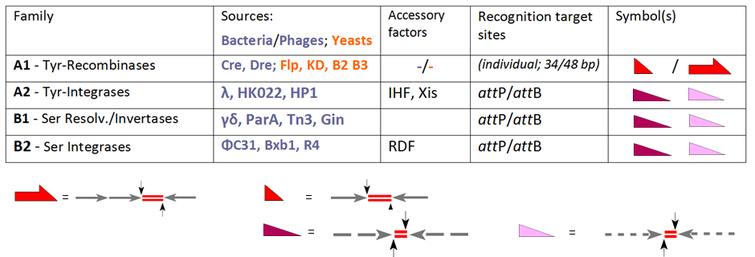
Основоположником семейства YR является лямбда-интеграза, кодируемая бактериофагом λ , что позволяет интегрировать фаговую ДНК в бактериальный геном. Общей чертой этого класса является консервативный тирозиновый нуклеофил, атакующий разрезаемый ДНК-фосфат с образованием 3'-фосфотирозиновой связи. Ранние члены семейства SR являются близкородственными резолвазами/ДНК-инвертазами из бактериальных транспозонов Tn3 и γδ, которые полагаются на каталитический серин, ответственный за атаку разрезаемого фосфата с образованием 5'-фосфосериновой связи. Однако эти бесспорные факты были скомпрометированы изрядной долей путаницы в то время, когда на сцену вышли другие члены, например, рекомбиназы YR Cre и Flp (способные к интеграции, вырезанию/разрешению, а также инверсии), которые, тем не менее, приветствовались как новые члены «семейства интеграз». Обратными примерами являются PhiC31 и родственные SR, которые изначально были введены как резолвазы/инвертазы, хотя при отсутствии вспомогательных факторов интеграция является их единственной функцией. В настоящее время стандартная активность каждого фермента определяет его классификацию, резервируя общий термин «рекомбиназа» для членов семейства, которые, по сути, включают все три пути, INT, RES и INV:
Наша таблица расширяет выбор обычных систем SSR и группирует их в соответствии с их производительностью. Все эти ферменты рекомбинируют два целевых сайта, которые либо идентичны (подсемейство A1), либо различны (ферменты, полученные из фага в A2, B1 и B2). [6] В то время как для A1 эти сайты имеют индивидуальные обозначения (« FRT » в случае Flp-рекомбиназы, loxP для Cre-рекомбиназы), термины « att P» и « att B» (сайты прикрепления на фаговой и бактериальной части соответственно) действительны в других случаях. В случае подсемейства A1 мы имеем дело с короткими (обычно 34 п.н.) сайтами, состоящими из двух (почти) идентичных плеч по 13 п.н. (стрелки), фланкирующих 8-п.н. спейсер (область кроссовера, обозначенная красными дублетами линий). [7] Обратите внимание, что для Flp существует альтернативный сайт из 48 пар оснований с тремя плечами, каждое из которых вмещает единицу Flp (так называемый «протомер»). Сайты att P и att B следуют схожим архитектурным правилам, но здесь плечи показывают лишь частичную идентичность (обозначенную пунктирными линиями) и различаются в обоих случаях. Эти особенности объясняют соответствующие различия:
Для того, чтобы упорядочить эту главу, следующие реализации будут сосредоточены на двух рекомбиназах (Flp и Cre) и только одной интегразе (PhiC31), поскольку их спектр охватывает инструменты, которые в настоящее время в основном используются для направленных модификаций генома. Это будет сделано в рамках следующего обзора.

Режим интеграции/разрешения и инверсии (INT/RES и INV) зависит от ориентации целевых участков рекомбиназы (RTS) среди этих пар att P и att B. Раздел C показывает, в упрощенном виде, способ, которым обмен кассетами, опосредованный рекомбиназой (RMCE), может быть достигнут с помощью синхронных двойных реципрокных кроссоверов (а не интеграции с последующим разрешением). [8] [9]
Tyr-Recombinases обратимы, в то время как Ser-Integrase однонаправлена. Следует отметить, как обратимая интеграция/разрешение Flp (Tyr-рекомбиназы) модулируется версиями FRT из 48 п.н. (вместо минимальных 34 п.н.) : дополнительное плечо из 13 п.н. служит «посадочным путем» Flp, способствуя формированию синаптического комплекса, как в контексте функций Flp-INT, так и Flp-RMCE (см. соответствующие равновесные ситуации). В то время как предотвратить (энтропийно-обусловленную) реверсию интеграции в разделе A для Cre едва ли возможно, а для Flp ее трудно достичь, RMCE может быть завершен, если донорская плазмида предоставлена в избытке из-за бимолекулярного характера как прямой, так и обратной реакции. Размещение обоих сайтов FRT в обратном порядке приведет к равновесию обеих ориентаций для вставки (зеленая стрелка). В отличие от Flp, Ser интеграза PhiC31 (нижние изображения) приводит к однонаправленной интеграции, по крайней мере, при отсутствии фактора направленности рекомбиназы (RDF-). [10] Относительно Flp-RMCE, который требует двух различных («гетероспецифических») мутантов FRT -спейсера, партнер реакции ( att B) первого реагирующего сайта att P поражается произвольно, так что нет контроля над направлением, в котором донорская кассета входит в цель (ср. альтернативные продукты). Также в отличие от Flp-RMCE , несколько различных целей RMCE не могут быть смонтированы параллельно из-за отсутствия гетероспецифических (неперекрестно взаимодействующих) комбинаций att P/ att B.
Рекомбиназа Cre (Cre) способна рекомбинировать определенные последовательности ДНК без необходимости в кофакторах . Фермент распознает 34 пары оснований ДНК-последовательности, называемые loxP («локус кроссинговера в фаге P1»). В зависимости от ориентации целевых участков по отношению друг к другу, Cre будет интегрировать/вырезать или инвертировать последовательности ДНК. При вырезании (называемым «разрешением» в случае кольцевого субстрата) определенного участка ДНК нормальная экспрессия гена значительно нарушается или прекращается. [11]
Из-за выраженной разрешающей активности Cre одним из его первоначальных применений было удаление генов lox P-фланкированных («floxed»), что приводит к клеточно-специфическому нокауту такого floxed гена после того, как Cre становится экспрессированным в интересующей ткани. Современные технологии включают методы, которые позволяют как пространственно, так и временно контролировать активность Cre. Обычный метод, облегчающий пространственный контроль генетических изменений, включает выбор тканеспецифического промотора для управления экспрессией Cre. Размещение Cre под контролем такого промотора приводит к локализованной тканеспецифической экспрессии. В качестве примера Леоне и др. поместили транскрипционную единицу под контроль регуляторных последовательностей гена миелинового протеолипидного белка (PLP), что приводит к индуцированному удалению целевых последовательностей генов в олигодендроцитах и шванновских клетках . [12] Конкретный фрагмент ДНК, распознаваемый Cre, остается нетронутым в клетках, которые не экспрессируют ген PLP; Это, в свою очередь, облегчает эмпирическое наблюдение за локальными эффектами изменений генома в миелиновой оболочке, окружающей нервные волокна в центральной нервной системе (ЦНС) и периферической нервной системе (ПНС). [13] Избирательная экспрессия Cre была достигнута также во многих других типах клеток и тканях.
Для контроля временной активности реакции эксцизии были разработаны формы Cre, которые используют преимущества различных доменов связывания лигандов. Одна из успешных стратегий для индукции специфической временной активности Cre включает слияние фермента с мутировавшим доменом связывания лигандов для человеческого рецептора эстрогена (ERt). После введения тамоксифена ( антагониста рецептора эстрогена ) конструкция Cre-ERt способна проникать в ядро и вызывать целевую мутацию. ERt связывает тамоксифен с большей аффинностью, чем эндогенные эстрогены , что позволяет Cre-ERt оставаться цитоплазматическим у животных, не получавших тамоксифен. Временной контроль активности SSR тамоксифеном позволяет индуцировать генетические изменения позже в эмбриогенезе и/или во взрослых тканях. [12] Это позволяет исследователям обходить эмбриональную летальность, продолжая изучать функцию целевых генов.
Недавние расширения этих общих концепций привели к созданию «Cre-zoo», т.е. коллекций сотен штаммов мышей, у которых определенные гены могут быть удалены путем целенаправленной экспрессии Cre. [3]

В своем естественном хозяине (S. cerevisiae) система Flp/ FRT обеспечивает репликацию «плазмиды 2μ» путем инверсии сегмента, который фланкирован двумя идентичными, но противоположно ориентированными сайтами FRT (активность «флиппазы»). Эта инверсия изменяет относительную ориентацию репликативных вилок внутри плазмиды, обеспечивая «катящийся круг» — амплификацию кольцевой сущности 2μ до того, как мультимерные промежуточные продукты будут разрешены для высвобождения множественных мономерных продуктов. В то время как минимальные сайты FRT размером 34 п.н. способствуют вырезанию/разрешению в той же степени, что и аналоговые сайты lox P для Cre, естественные, более протяженные варианты FRT размером 48 п.н. обеспечивают более высокую степень интеграции, преодолевая определенные беспорядочные взаимодействия, как описано для фаговых ферментов, таких как Cre- [5] и PhiC31. [6] Дополнительным преимуществом является тот факт, что простые правила могут быть применены для создания гетероспецифических сайтов FRT , которые подвергаются кроссинговерам с равными партнерами, но не с FRT дикого типа . Эти факты позволили с 1994 года разработать и постоянно совершенствовать стратегии обмена кассетами с помощью рекомбиназы (RMCE-), позволяющие производить чистую замену целевой кассеты на входящую донорскую кассету. [6]
На основе технологии RMCE в рамках программы EUCOMM (European Conditional Mouse Mutagenesis) был разработан определенный ресурс предварительно охарактеризованных штаммов ES, который поддается дальнейшей разработке, на основе уже существующих установок "FlExing" (Flp-опосредованное удаление/инверсия) на основе Cre и/или Flp [6] , включающих действия по удалению и инверсии. Начатый в 2005 году, этот проект был в первую очередь сосредоточен на мутагенезе насыщения для обеспечения полной функциональной аннотации генома мыши (координируемой Международным консорциумом по нокаутированию мышей, IKMC) с конечной целью мутировать все гены белков посредством захвата и нацеливания генов в мышиных ES-клетках. [14] Эти усилия знаменуют вершину различных стратегий «тегирования и обмена», которые посвящены маркировке отдельного геномного сайта таким образом, что «тег» может служить адресом для введения новой (или изменения существующей) генетической информации. Шаг маркировки сам по себе может быть направлен на определенные классы сайтов интеграции, используя предпочтения ретровирусов или даже сайт-специфических интеграз, таких как PhiC31, которые оба действуют по существу однонаправленным образом.
Традиционные трудоемкие процедуры «маркировки и обмена» основывались на двух последовательных шагах гомологичной рекомбинации (HR-), первый из которых («HR1») вводил метку, состоящую из гена маркера селекции. Затем «HR2» использовался для замены маркера на «GOI». В первой реакции («нокаут») ген был помечен селектируемым маркером, как правило, путем вставки кассеты hygtk ([+/-]), обеспечивающей устойчивость к G418. На следующем этапе «нок-ин» маркированная геномная последовательность была заменена гомологичными геномными последовательностями с определенными мутациями. Затем клоны клеток могли быть изолированы по их устойчивости к ганцикловиру из-за потери гена HSV-tk, т. е. («отрицательный отбор»). Эта обычная двухэтапная процедура маркировки и обмена [15] могла быть упрощена после появления RMCE, которая могла бы взять на себя и повысить эффективность этапа нока-ин.
Без особых сомнений, интегразы Ser являются текущими инструментами выбора для интеграции трансгенов в ограниченное число хорошо изученных геномных акцепторных сайтов, которые в основном (но не всегда) имитируют сайт att P фага, поскольку они привлекают донорный вектор, содержащий att B. На данный момент наиболее заметным членом является PhiC31-INT с доказанным потенциалом в контексте геномов человека и мыши.
В отличие от вышеупомянутых рекомбиназ Tyr, PhiC31-INT как таковая действует однонаправленно, прочно фиксируя донорный вектор на геномно закрепленной цели. Очевидное преимущество этой системы заключается в том, что она может полагаться на немодифицированные, нативные att P (акцептор) и донорные сайты att B. Дополнительные преимущества (вместе с определенными осложнениями) могут возникнуть из-за того, что геномы мыши и человека сами по себе содержат ограниченное количество эндогенных целей (так называемые « att P-псевдосайты»). Имеющаяся информация предполагает, что значительные требования к последовательности ДНК позволяют интегразе распознавать меньше сайтов, чем ретровирусные или даже основанные на транспозазе интеграционные системы, открывая ее карьеру в качестве превосходного транспортного средства для транспортировки и вставки в ряд хорошо известных геномных сайтов, некоторые из которых обладают свойствами так называемой «безопасной гавани». [10]
Используя факт специфических ( att P x att B) путей рекомбинации, RMCE становится возможным без необходимости в синтетических, гетероспецифических att -сайтах. Однако это очевидное преимущество достигается за счет определенных недостатков, таких как отсутствие контроля над типом или направленностью входящей (донорской) кассеты. [6] Дополнительные ограничения налагаются тем фактом, что необратимость не допускает стандартные настройки мультиплексирования RMCE, включая реакции «серийного RMCE», т. е. повторные обмены кассетами в заданном геномном локусе .
Аннотация геномов человека и мыши привела к идентификации >20 000 генов, кодирующих белки, и >3 000 некодирующих генов РНК, которые направляют развитие организма от оплодотворения через эмбриогенез до взрослой жизни. Хотя отмечен значительный прогресс, актуальность редких вариантов генов остается центральной темой исследований.
В качестве одной из важнейших платформ для работы с функциями генов позвоночных в больших масштабах были созданы геномные генетические ресурсы мутантных мышиных ES-клеток. С этой целью в Европе и Северной Америке были основаны четыре международные программы, направленные на насыщающий мутагенез мышиного генома (EUCOMM, KOMP, NorCOMM и TIGM). Координируемые Международным консорциумом по нокаутированным мышам (IKSC), эти репозитории ES-клеток доступны для обмена между международными исследовательскими подразделениями. Текущие ресурсы включают мутации в 11 539 уникальных генах, 4 414 из которых условные. [14]
Соответствующие технологии в настоящее время достигли уровня, позволяющего распространить их на другие виды млекопитающих и стволовые клетки человека, в первую очередь те, которые имеют статус iPS (индуцированных плюрипотентных) .