Секвенирование отдельных клеток исследует информацию о последовательности нуклеиновых кислот из отдельных клеток с помощью оптимизированных технологий секвенирования нового поколения , обеспечивая более высокое разрешение клеточных различий и лучшее понимание функции отдельной клетки в контексте ее микроокружения. [1] Например, при раке секвенирование ДНК отдельных клеток может дать информацию о мутациях, переносимых небольшими популяциями клеток. В процессе разработки секвенирование РНК, экспрессируемых отдельными клетками, может дать представление о существовании и поведении различных типов клеток. [2] В микробных системах популяция одного и того же вида может оказаться генетически клональной. Тем не менее, одноклеточное секвенирование РНК или эпигенетические модификации могут выявить межклеточную изменчивость, которая может помочь популяциям быстро адаптироваться к выживанию в меняющихся условиях окружающей среды. [3]
Типичная человеческая клетка состоит примерно из 2х3,3 миллиардов пар оснований ДНК и 600 миллионов оснований мРНК. Обычно для секвенирования ДНК или РНК используется смесь миллионов клеток с использованием традиционных методов, таких как секвенирование по Сэнгеру или секвенирование Illumina . Путем глубокого секвенирования ДНК и РНК из одной клетки можно широко исследовать клеточные функции. [1] Как и типичные эксперименты по секвенированию следующего поколения, протоколы секвенирования отдельных клеток обычно включают следующие этапы: выделение одной клетки, извлечение и амплификация нуклеиновой кислоты, подготовка библиотеки секвенирования , секвенирование и биоинформационный анализ данных. Выполнить секвенирование отдельных клеток сложнее, чем секвенирование целых клеток. Минимальное количество исходных материалов из одной клетки приводит к тому, что деградация, потеря образцов и загрязнение оказывают заметное влияние на качество данных секвенирования. Кроме того, из-за пикограммного уровня количества используемых нуклеиновых кислот [4] во время подготовки образцов для секвенирования отдельных клеток часто требуется тяжелая амплификация, что приводит к неравномерному охвату, шуму и неточной количественной оценке данных секвенирования.
Недавние технические усовершенствования делают секвенирование отдельных клеток многообещающим инструментом для решения ряда, казалось бы, недоступных проблем. Например, гетерогенные образцы, редкие типы клеток, взаимоотношения клеточных линий, мозаицизм соматических тканей, анализ микробов, которые невозможно культивировать, а также эволюцию заболевания — все это можно выяснить с помощью секвенирования отдельных клеток. [5] Секвенирование одиночных клеток было выбрано издательской группой Nature Publishing Group в качестве метода 2013 года. [6]
Секвенирование генома одноклеточной ДНК включает выделение одной клетки, амплификацию всего генома или интересующей области, создание библиотек секвенирования, а затем применение секвенирования ДНК следующего поколения (например, Illumina , Ion Torrent , MGI). Секвенирование одноклеточной ДНК широко применяется в системах млекопитающих для изучения нормальной физиологии и заболеваний. Разрешение одиночных клеток может раскрыть роль генетического мозаицизма или внутриопухолевой генетической гетерогенности в развитии рака или реакции на лечение. [7] В контексте микробиомов геном одного одноклеточного организма называется единым амплифицированным геномом (SAG). Достижения в секвенировании одноклеточной ДНК позволили собрать геномные данные от некультивируемых видов прокариот, присутствующих в сложных микробиомах. [8] Хотя SAG характеризуются низкой полнотой и значительной погрешностью, последние вычислительные достижения позволили собрать почти полные геномы из составных SAG. [9] Данные, полученные от микроорганизмов, могут определить процессы культивирования в будущем. [10] Некоторые из инструментов сборки генома, используемых при секвенировании отдельных клеток, включают SPAdes , IDBA-UD, Cortex и HyDA. [11]

Опубликован список из более чем 100 различных методов омики отдельных клеток . [12]
Множественная амплификация смещения (MDA) — широко используемый метод, позволяющий амплифицировать фемтограммы ДНК бактерий до микрограмм для секвенирования. Реагенты, необходимые для реакций MDA, включают: случайные праймеры и ДНК-полимеразу бактериофага phi29. В 30-градусной изотермической реакции ДНК амплифицируется с включенными реагентами. Когда полимеразы производят новые цепи, происходит реакция замещения цепи, в результате которой из каждой матрицы ДНК синтезируется множество копий. При этом пряди, которые были наращены ранее, сместятся. Продукты MDA имеют длину около 12 КБ и варьируются примерно до 100 КБ, что позволяет использовать их в секвенировании ДНК. [10] В 2017 году было введено значительное усовершенствование этого метода, получившее название WGA-X, за счет использования термостабильного мутанта полимеразы phi29, что привело к лучшему восстановлению генома из отдельных клеток, особенно из клеток с высоким содержанием G + C. . [13] MDA также был реализован в микрожидкостной системе на основе капель для достижения высокопараллельной амплификации всего генома одной клетки. Инкапсулируя отдельные клетки в капли для захвата и амплификации ДНК, этот метод обеспечивает снижение систематической ошибки и повышение производительности по сравнению с традиционным MDA. [14]
Другой распространенный метод — MALBAC . [15] Как и в случае с MDA, этот метод начинается с изотермической амплификации, но праймеры фланкируются «общей» последовательностью для последующей ПЦР-амплификации. По мере генерации предварительных ампликонов общая последовательность способствует самолигированию и образованию «петлей», предотвращающих дальнейшую амплификацию. В отличие от MDA, сильно разветвленная сеть ДНК не образуется. Вместо этого петли денатурируются в другом температурном цикле, что позволяет амплифицировать фрагменты с помощью ПЦР. MALBAC также был реализован в микрофлюидном устройстве, но эффективность амплификации не была существенно улучшена за счет инкапсуляции в нанолитровые капли. [16]
Сравнивая MDA и MALBAC, MDA обеспечивает лучшее покрытие генома, но MALBAC обеспечивает более равномерное покрытие по всему геному. MDA может быть более эффективным для идентификации SNP , тогда как MALBAC предпочтительнее для обнаружения вариантов количества копий. Хотя выполнение MDA с помощью микрофлюидного устройства заметно снижает предвзятость и загрязнение, химический состав, используемый в MALBAC, не демонстрирует такого же потенциала для повышения эффективности.
Методом, особенно подходящим для обнаружения структурных вариаций генома , является секвенирование цепей матрицы одноклеточной ДНК (также известное как Strand-seq). [17] Используя принцип трехканальной обработки отдельных клеток, который использует совместное моделирование ориентации чтения, глубины чтения и фазы гаплотипа, Strand-seq позволяет обнаруживать полный спектр классов соматических структурных вариаций ≥200 КБ в размер. Strand-seq преодолевает ограничения методов, основанных на амплификации всего генома, для идентификации классов соматических генетических вариаций в отдельных клетках [18] , поскольку он невосприимчив к считываемым химерам, приводящим к возникновению артефактов (подробно обсуждаемых в разделе ниже), и менее эффективен. пострадали от отсева. Выбор метода зависит от цели секвенирования, поскольку каждый метод имеет разные преимущества. [7]
MDA геномов отдельных клеток приводит к крайне неравномерному покрытию генома, т.е. к относительному перепредставлению и недопредставленности различных областей матрицы, что приводит к потере некоторых последовательностей. Этот процесс состоит из двух компонентов: а) стохастическое чрезмерное и недостаточное усиление случайных областей; и б) систематическое смещение в отношении регионов с высоким %GC. Стохастический компонент можно устранить путем объединения одноклеточных реакций MDA из одного и того же типа клеток, используя флуоресцентную гибридизацию in situ (FISH) и/или подтверждение после секвенирования. [10] Смещение MDA в отношении областей с высоким содержанием %GC можно устранить с помощью термостабильных полимераз, например, в процессе под названием WGA-X. [13]
Однонуклеотидные полиморфизмы (SNP), которые составляют значительную часть генетических вариаций в геноме человека , и вариации числа копий (CNV), создают проблемы при секвенировании одной клетки, а также ограниченное количество ДНК, экстрагируемой из одной клетки. Из-за небольшого количества ДНК точный анализ ДНК создает проблемы даже после амплификации, поскольку охват невелик и подвержен ошибкам. При использовании MDA средний охват генома составляет менее 80%, и SNP, которые не охвачены считыванием секвенирования, будут исключены. Кроме того, MDA показывает высокий процент выпадения аллелей , не обнаруживая аллелей в гетерозиготных образцах. В настоящее время используются различные алгоритмы SNP, но ни один из них не предназначен для секвенирования одиночных клеток. MDA с CNV также создает проблему выявления ложных CNV, скрывающих настоящие CNV. Чтобы решить эту проблему, когда на основе ложных CNV можно генерировать шаблоны, алгоритмы могут обнаруживать и устранять этот шум, создавая истинные варианты. [19]
Strand-seq преодолевает ограничения методов, основанных на амплификации всего генома, для вызова генетических вариантов: поскольку Strand-seq не требует операций чтения (или пар чтения), пересекающих границы (или точки останова) CNV или классов структурных вариантов со сбалансированным копированием, он требует меньше усилий. восприимчивы к обычным артефактам одноклеточных методов, основанным на амплификации всего генома, которые включают в себя пропуски вызова вариантов из-за отсутствия чтений в точке останова варианта и химеру чтения. [7] [18] Strand-seq обнаруживает полный спектр классов структурных вариаций размером не менее 200 КБ, включая циклы разрыв-слияние-мостик и события хромотрипсиса , а также сбалансированные инверсии, а также сбалансированные или несбалансированные транслокации по числу копий. [ 18 ] «Вызовы структурных вариантов, сделанные Strand-seq, разрешаются гаплотипом длины хромосомы , что обеспечивает дополнительную специфичность вызова вариантов . ), и этот метод не обнаруживает варианты размером менее 200 КБ, такие как вставки мобильных элементов .
Микробиомы являются одной из основных целей геномики отдельных клеток из-за сложности культивирования большинства микроорганизмов в большинстве сред. Геномика одиночных клеток — мощный способ получить последовательности микробного генома без культивирования. Этот подход широко применялся к морским, почвенным, подповерхностным, организменным и другим типам микробиомов для решения широкого спектра вопросов, связанных с микробной экологией, эволюцией, общественным здравоохранением и биотехнологическим потенциалом. [20] [21] [22] [23] [24] [25] [26] [27] [28]
Секвенирование рака также является новым применением scDNAseq. Свежие или замороженные опухоли можно достаточно хорошо проанализировать и классифицировать в отношении SCNA, SNV и перестроек, используя подходы полногеномной ДНК. [29] СкДНКсек рака особенно полезен для изучения глубины сложности и сложных мутаций, присутствующих в амплифицированных терапевтических мишенях, таких как гены рецепторных тирозинкиназ (EGFR, PDGFRA и т. д.), когда традиционные подходы на популяционном уровне к объемной опухоли не могут разрешить проблему. закономерности одновременного возникновения этих мутаций в отдельных клетках опухоли. Такое перекрытие может обеспечить избыточность активации пути и устойчивости опухолевых клеток.

Секвенирование метилома одноклеточной ДНК позволяет количественно оценить метилирование ДНК . В природе встречается несколько известных типов метилирования, в том числе 5-метилцитозин (5mC), 5-гидроксиметилцитозин (5hmC), 6-метиладенин (6mA) и 4-метилцитозин 4mC (4mC). У эукариот, особенно у животных, 5mC широко распространен по геному и играет важную роль в регуляции экспрессии генов путем репрессии мобильных элементов . [31] Секвенирование 5mC в отдельных клетках может выявить, как эпигенетические изменения в генетически идентичных клетках из одной ткани или популяции приводят к появлению клеток с разными фенотипами.
Бисульфитное секвенирование стало золотым стандартом обнаружения и секвенирования 5mC в отдельных клетках. [32] Обработка ДНК бисульфитом превращает остатки цитозина в урацил, но оставляет остатки 5-метилцитозина незатронутыми. Следовательно, ДНК, обработанная бисульфитом, сохраняет только метилированные цитозины. Чтобы получить показания метилома, обработанную бисульфитом последовательность выравнивают по немодифицированному геному. Полногеномное бисульфитное секвенирование было достигнуто в отдельных клетках в 2014 году. [33] Этот метод позволяет избежать потери ДНК, связанной с типичной процедурой, когда адаптеры секвенирования добавляются до фрагментации бисульфита. Вместо этого адаптеры добавляются после обработки и фрагментации ДНК бисульфитом, что позволяет амплифицировать все фрагменты с помощью ПЦР. [34] Используя глубокое секвенирование, этот метод захватывает ~ 40% общего количества CpG в каждой клетке. С помощью существующей технологии ДНК не может быть амплифицирована до обработки бисульфитом, поскольку метки 5mC не будут копироваться полимеразой.
Еще одним методом является бисульфитное секвенирование с уменьшенным представлением отдельных клеток (scRRBS). [35] Этот метод использует тенденцию метилированных цитозинов группироваться на CpG-островках (CGI) для обогащения областей генома с высоким содержанием CpG. Это снижает стоимость секвенирования по сравнению с полногеномным бисульфитным секвенированием, но ограничивает охват этого метода. Когда RRBS применяется к объемным образцам, обнаруживается большинство сайтов CpG в промоторах генов, но сайты в промоторах генов составляют только 10% сайтов CpG во всем геноме. [36] В одиночных клетках обнаруживается 40% сайтов CpG из общего образца. Чтобы увеличить охват, этот метод также можно применить к небольшому пулу одиночных ячеек. В образце из 20 объединенных одиночных клеток было обнаружено 63% сайтов CpG из основной выборки. Объединение отдельных клеток является одной из стратегий увеличения покрытия метиломов, но за счет сокрытия гетерогенности в популяции клеток.
Хотя бисульфитное секвенирование остается наиболее широко используемым подходом для обнаружения 5 мС, химическая обработка является жесткой, фрагментирует и разрушает ДНК. Этот эффект усугубляется при переходе от объемных образцов к одиночным клеткам. Другие методы обнаружения метилирования ДНК включают чувствительные к метилированию ферменты рестрикции. Ферменты рестрикции также позволяют обнаруживать другие типы метилирования, например, 6 мА с помощью DpnI. [37] Секвенирование на основе нанопор также предлагает путь для прямого секвенирования метилирования без фрагментации или модификации исходной ДНК. Секвенирование нанопор использовалось для секвенирования метиломов бактерий, в которых доминируют 6mA и 4mC (в отличие от 5mC у эукариот), но этот метод еще не масштабирован до отдельных клеток. [38]
Секвенирование метилирования одноклеточной ДНК широко используется для изучения эпигенетических различий в генетически сходных клетках. Чтобы проверить эти методы во время их разработки, данные о метиломе отдельных клеток смешанной популяции были успешно классифицированы с помощью иерархической кластеризации для идентификации различных типов клеток. [35] Другое применение — изучение отдельных клеток во время первых нескольких клеточных делений на ранних стадиях развития, чтобы понять, как разные типы клеток возникают из одного эмбриона. [39] Полногеномное бисульфитное секвенирование отдельных клеток также использовалось для изучения редких, но высокоактивных типов клеток при раке, таких как циркулирующие опухолевые клетки (ЦОК). [40]

Секвенирование одноклеточного хроматина, доступного для транспозазы, отображает доступность хроматина по всему геному. Транспозаза вставляет адаптеры секвенирования непосредственно в открытые области хроматина, позволяя амплифицировать и секвенировать эти области. [41]
Стандартные методы, такие как микрочипы и объемное секвенирование РНК, анализируют экспрессию РНК в больших популяциях клеток. Эти измерения могут скрыть критические различия между отдельными клетками в популяциях смешанных клеток. [42] [43]
Секвенирование одноклеточной РНК (scRNA-seq) обеспечивает профили экспрессии отдельных клеток и считается золотым стандартом для определения состояний и фенотипов клеток по состоянию на 2020 год. [44] Хотя невозможно получить полную информацию о каждой РНК, экспрессируемой каждым В клетке из-за небольшого количества доступного материала закономерности экспрессии генов можно определить с помощью анализа кластеризации генов . [45] Это может выявить редкие типы клеток в популяции клеток, которые, возможно, никогда раньше не наблюдались. Например, одна группа ученых, проводившая scRNA-секвенирование на опухолевой ткани нейробластомы, выявила редкую раковую клетку паннейробластомы, которая может быть привлекательной для новых подходов к терапии. [46]
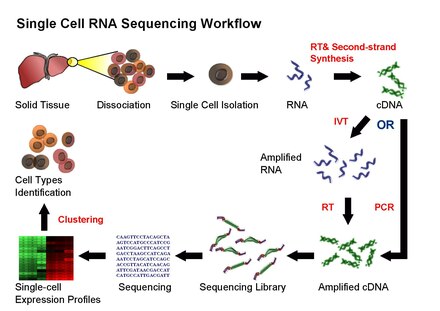
Текущие протоколы секвенирования scRNA включают выделение отдельных клеток и их РНК, а затем выполнение тех же шагов, что и массовое секвенирование РНК: обратную транскрипцию (RT), амплификацию, создание библиотеки и секвенирование. Ранние методы разделяли отдельные клетки в отдельные лунки; более поздние методы инкапсулируют отдельные клетки в капли в микрофлюидном устройстве, где происходит реакция обратной транскрипции, превращающая РНК в кДНК. Каждая капля несет «штрих-код» ДНК, который однозначно маркирует кДНК, полученные из одной клетки. После завершения обратной транскрипции кДНК из многих клеток можно смешать для секвенирования, поскольку транскрипты из конкретной клетки идентифицируются по уникальному штрих-коду. [47] [48]
Задачи scRNA-Seq включают сохранение первоначального относительного содержания мРНК в клетке и выявление редких транскриптов. [49] Этап обратной транскрипции имеет решающее значение, поскольку эффективность реакции RT определяет, какая часть клеточной популяции РНК в конечном итоге будет проанализирована секвенатором. Процессивность обратных транскриптаз и используемые стратегии прайминга могут влиять на производство полноразмерной кДНК и создание библиотек, смещенных в сторону 3'- или 5'-конца генов .
На этапе амплификации в настоящее время для амплификации кДНК используется либо ПЦР, либо транскрипция in vitro (IVT). Одним из преимуществ методов на основе ПЦР является возможность генерировать полноразмерную кДНК. Однако различная эффективность ПЦР для определенных последовательностей (например, содержания GC и структуры snapback) также может экспоненциально усиливаться, создавая библиотеки с неравномерным покрытием. С другой стороны, хотя библиотеки, созданные с помощью IVT, могут избежать смещения последовательностей, вызванного ПЦР, определенные последовательности могут транскрибироваться неэффективно, что приводит к выпадению последовательностей или образованию неполных последовательностей. [1] [42] Опубликовано несколько протоколов scRNA-seq: Tang et al., [50] STRT, [51] SMART-seq, [52] SORT-seq, [53] CEL-seq, [54] RAGE . -сек, [55] Кварц-сек. [56] и C1-CAGE. [57] Эти протоколы различаются стратегиями обратной транскрипции, синтеза и амплификации кДНК, а также возможностью использования штрих-кодов, специфичных для последовательности (т. е. UMI ), или способностью обрабатывать объединенные образцы. [58]
В 2017 году были представлены два подхода для одновременного измерения одноклеточной экспрессии мРНК и белка с помощью меченных олигонуклеотидами антител, известных как REAP-seq [59] и CITE-seq. [60] Сбор клеточного содержимого после электрофизиологической записи с использованием патч-клампа также позволил разработать метод Patch-Seq , который неуклонно завоевывает популярность в нейробиологии. [61]
Эта платформа секвенирования одноклеточной РНК позволяет анализировать транскриптомы по клеткам с помощью микрофлюидного разделения для захвата отдельных клеток и подготовки библиотек кДНК для секвенирования следующего поколения (NGS) . [62] Платформа на основе капель позволяет массово параллельно секвенировать мРНК в большом количестве отдельных клеток путем захвата одной клетки в капле масла. [63]
В целом, на первом этапе отдельные клетки захватываются отдельно и лизируются, затем выполняется обратная транскрипция (ОТ) мРНК и получается библиотека кДНК . Для отбора мРНК проводят RT с использованием одноцепочечной последовательности дезокситиминового (олиго дТ) праймера , который специфически связывает поли(А)-хвост молекул мРНК. Впоследствии амплифицированную библиотеку кДНК используют для секвенирования. [64]
Итак, первым шагом метода является инкапсуляция одной клетки и подготовка библиотеки. Клетки инкапсулируются в гелевые шарики в эмульсии (GEM) с помощью автомата. Для формирования этих пузырьков автомат использует микрофлюидный чип и объединяет все компоненты с маслом. Каждый функциональный GEM содержит одну ячейку, один шарик геля и реагенты RT. На гелевых шариках связываются олигнонуклеотиды, состоящие из 4 отдельных частей: праймер для ПЦР (необходим для секвенирования); 10X олигонуклеотиды со штрих-кодом; Последовательность уникального молекулярного идентификатора (UMI); Последовательность PolydT (которая позволяет захватывать полиаденилированные молекулы мРНК). [65] Внутри каждой реакционной везикулы GEM одна клетка лизируется и подвергается обратной транскрипции. кДНК из одной и той же клетки идентифицируются благодаря общему штрих-коду 10X. Кроме того, количество UMI отражает уровень экспрессии генов, и их анализ позволяет обнаружить высоковариабельные гены. Эти данные часто используются либо для классификации клеточных фенотипов, либо для идентификации новой субпопуляции. [66]
Последним шагом платформы является секвенирование. Созданные библиотеки можно напрямую использовать для секвенирования всего транскриптома отдельной клетки или рабочих процессов целевого секвенирования. Секвенирование проводят с использованием метода секвенирования красителей Illumina . Этот метод секвенирования основан на принципе секвенирования путем синтеза (SBS) и использовании обратимого красителя-терминатора, который позволяет идентифицировать каждый отдельный нуклеотид. Для считывания последовательностей транскриптов на одном конце и штрих-кода и UMI на другом конце требуются считыватели парных концов. [67]
Платформа на основе капель позволяет обнаруживать редкие типы клеток благодаря своей высокой пропускной способности. Фактически, в одном образце из одной клеточной суспензии захватывается от 500 до 10 000 клеток. Протокол выполняется легко и обеспечивает высокую скорость восстановления клеток — до 65%. Глобальный рабочий процесс капельной платформы занимает 8 часов и поэтому быстрее, чем метод на основе Microwell (BD Rhapsody), который занимает 10 часов. Однако он имеет некоторые ограничения, такие как необходимость свежих образцов и окончательное обнаружение только 10% мРНК.
Основное различие между методом, основанным на каплях, и методом, основанным на микролунках, заключается в методе разделения клеток. [64]
Большинство методов секвенирования РНК зависят от захвата хвоста поли(А) для обогащения мРНК и истощения обильных и неинформативных рРНК. Таким образом, они часто ограничиваются секвенированием полиаденилированных молекул мРНК. Однако недавние исследования теперь начинают понимать важность неполи(А) РНК, таких как длинные некодирующие РНК и микроРНК, в регуляции экспрессии генов. Small-seq — это одноклеточный метод, который захватывает небольшие РНК (<300 нуклеотидов), такие как микроРНК, фрагменты тРНК и малые ядрышковые РНК в клетках млекопитающих. [68] Этот метод использует комбинацию «олигонуклеотидных масок» (которые ингибируют захват очень распространенных молекул 5.8S рРНК) и выбора размера для исключения крупных видов РНК, таких как другие очень распространенные молекулы рРНК. Для нацеливания на более крупные неполи(А) РНК, такие как длинная некодирующая мРНК, гистоновая мРНК, кольцевая РНК и энхансерная РНК, выбор размера неприменим для истощения очень распространенных молекул рибосомальной РНК (18S и 28s рРНК). [69] Одноклеточный RamDA-Seq — это метод, который достигает этого путем выполнения обратной транскрипции со случайным праймированием (амплификация со случайным смещением) в присутствии «не столь случайных» (NSR) праймеров, специально разработанных для предотвращения праймирования молекулы рРНК. [70] Хотя этот метод успешно захватывает полноразмерные полные транскрипты РНК для секвенирования и обнаруживает множество неполи(А) РНК с высокой чувствительностью, он имеет некоторые ограничения. Праймеры NSR были тщательно разработаны в соответствии с последовательностями рРНК конкретного организма (мыши), и разработка новых наборов праймеров для других видов потребовала бы значительных усилий. Недавно основанный на CRISPR метод под названием scDASH (истощение одноклеточных обильных последовательностей путем гибридизации) продемонстрировал другой подход к истощению последовательностей рРНК из одноклеточных библиотек тотальных РНК-seq. [71]
Бактерии и другие прокариоты в настоящее время не поддаются секвенированию одноклеточной РНК из-за отсутствия полиаденилированной мРНК. Таким образом, разработка методов секвенирования одноклеточной РНК, которые не зависят от захвата хвоста поли(А), также будет способствовать проведению исследований микробиома с разрешением одиночных клеток. Массовые исследования бактерий обычно применяют общее истощение рРНК, чтобы преодолеть недостаток полиаденилированной мРНК у бактерий, но на уровне отдельных клеток общее количество РНК, обнаруженное в одной клетке, слишком мало. [69] Отсутствие полиаденилированной мРНК и нехватка тотальной РНК, обнаруженной в отдельных бактериальных клетках, являются двумя важными барьерами, ограничивающими внедрение scRNA-seq в бактериях.
scRNA-Seq становится широко используемым в биологических дисциплинах, включая биологию развития , [72] неврологию , [73] онкологию , [74] [75] [76] иммунологию , [77] [78] сердечно-сосудистые исследования [79] [80] и Инфекционное заболевание . [81] [82]
Используя методы машинного обучения , данные массового секвенирования РНК были использованы для увеличения соотношения сигнал/шум в scRNA-Seq. В частности, ученые использовали профили экспрессии генов из наборов данных по раку , чтобы построить сети коэкспрессии , а затем применили их к профилям экспрессии генов в отдельных клетках, получив более надежный метод обнаружения наличия мутаций в отдельных клетках с использованием уровней транскриптов. [83]
Некоторые методы scRNA-seq также применялись к одноклеточным микроорганизмам. SMART-seq2 использовался для анализа одноклеточных эукариотических микробов, но, поскольку он основан на захвате хвоста поли(А), он не применялся в прокариотических клетках. [84] Микрофлюидные подходы, такие как устройства Drop-seq и Fluidigm IFC-C1, использовались для секвенирования отдельных малярийных паразитов или отдельных дрожжевых клеток. [85] [86] Исследование одноклеточных дрожжей стремилось охарактеризовать гетерогенную толерантность к стрессу в изогенных дрожжевых клетках до и после того, как дрожжи подверглись солевому стрессу. Анализ отдельных клеток нескольких факторов транскрипции с помощью scRNA-seq выявил гетерогенность в популяции. Эти результаты показывают, что регулирование варьируется среди членов популяции, чтобы увеличить шансы на выживание для части населения.
Первый анализ транскриптома одиночных клеток у прокариотических видов был выполнен с использованием фермента терминаторной экзонуклеазы для избирательного расщепления рРНК и амплификации мРНК по катящемуся кругу (RCA). [87] В этом методе концы одноцепочечной ДНК лигировались вместе, образуя кольцо, а полученная петля затем использовалась в качестве матрицы для линейной амплификации РНК. Библиотеку конечных продуктов затем анализировали с помощью микрочипа с низкой погрешностью и хорошим охватом. Однако RCA не тестировался с помощью секвенирования РНК, в котором обычно используется секвенирование следующего поколения. Секвенирование одноклеточной РНК бактерий было бы очень полезно для изучения микробиомов. Это позволит решить проблемы, возникающие при использовании традиционных подходов массовой метатранскриптомики, такие как неспособность уловить виды, присутствующие в низкой численности, и неспособность устранить гетерогенность среди клеточных популяций.
scRNA-Seq предоставил значительную информацию о развитии эмбрионов и организмов, включая червя Caenorhabditis elegans [88] и регенеративной планарии Schmidtea mediterranea [89] [90] и аксолотля Ambystoma mexicanum . [91] [92] Первыми позвоночными животными, картографированными таким способом, были рыбки данио [93] [94] [95] и Xenopus laevis . [96] В каждом случае изучались несколько стадий эмбриона, что позволяло картировать весь процесс развития на клеточной основе. Наука признала эти достижения « Прорывом года 2018» . [97]
Был создан атлас молекулярных клеток семенников мышей для определения токсичности яичек в препубертатном периоде, индуцированной BDE47, с использованием подхода ScRNA-seq, что дает новое понимание наших механизмов и путей, лежащих в основе BDE47-ассоциированного повреждения яичек, с разрешением отдельных клеток. [98]
Существует несколько способов изолировать отдельные клетки перед амплификацией и секвенированием всего генома. Сортировка клеток, активируемая флуоресценцией (FACS), является широко используемым подходом. Отдельные клетки также можно собирать с помощью микроманипуляций, например, путем серийного разведения или с помощью патч-пипетки или нанотрубки для сбора одной клетки. [15] [99] Преимуществами микроманипуляций являются простота и низкая стоимость, но они трудоемки и подвержены ошибочной идентификации типов клеток под микроскопом. Микродиссекция с лазерным захватом (LCM) также может использоваться для сбора отдельных клеток. Хотя LCM сохраняет информацию о пространственном расположении выбранной клетки в ткани, трудно захватить целую отдельную клетку, не собрав при этом материалы из соседних клеток. [42] [100] [101] Высокопроизводительные методы выделения одиночных клеток также включают микрофлюидику . И FACS, и микрофлюидика являются точными, автоматическими и способны изолировать объективные образцы. Однако оба метода требуют сначала отделения клеток от их микроокружения, тем самым вызывая нарушения транскрипционных профилей при анализе экспрессии РНК. [102] [103]
Вообще говоря, для типичного эксперимента по секвенированию РНК в массовых клетках (RNA-seq) генерируются десять миллионов чтений, и ген с числом чтений, превышающим порог в 50 чтений на КБ на миллион чтений (RPKM), считается экспрессированным. Для гена длиной 1 КБ это соответствует 500 чтениям и минимальному коэффициенту вариации (CV) 4% в предположении распределения Пуассона . Для типичной клетки млекопитающего, содержащей 200 000 мРНК, необходимо объединить данные секвенирования по меньшей мере 50 отдельных клеток, чтобы достичь этого минимального значения CV. Однако из-за эффективности обратной транскрипции и другого шума, введенного в экспериментах, для точного анализа экспрессии и идентификации типа клеток требуется больше клеток. [42]