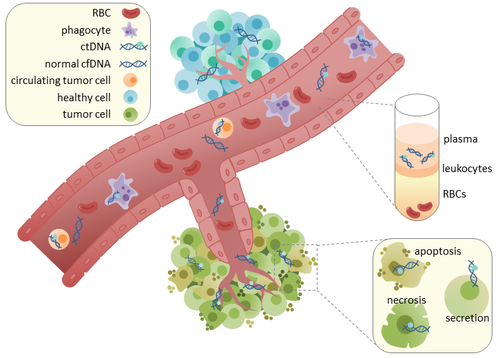
Циркулирующая опухолевая ДНК (ctDNA) — это фрагментированная ДНК, полученная из опухоли в кровотоке, которая не связана с клетками. ctDNA не следует путать с бесклеточной ДНК ( cfDNA ), более широким термином, который описывает ДНК, свободно циркулирующую в кровотоке, но не обязательно опухолевого происхождения. Поскольку ctDNA может отражать весь геном опухоли , она приобрела популярность благодаря своей потенциальной клинической полезности; « жидкие биопсии » в виде заборов крови могут проводиться в различные временные точки для мониторинга прогрессирования опухоли на протяжении всего курса лечения. [1] [2]
Недавние исследования заложили основу для вывода экспрессии генов из cfDNA (и ctDNA), при этом EPIC-seq стал заметным достижением. [3] Этот метод существенно поднял планку для неинвазивного вывода уровней экспрессии отдельных генов, тем самым расширив применимость анализа для характеристики заболеваний, гистологической классификации и мониторинга эффективности лечения. [3] [4] [5]
ctDNA происходит непосредственно из опухоли или из циркулирующих опухолевых клеток (CTCs), [6] , что описывает жизнеспособные, неповрежденные опухолевые клетки, которые отделяются от первичных опухолей и попадают в кровоток или лимфатическую систему. Точный механизм высвобождения ctDNA неясен. Биологические процессы, которые, как предполагается, участвуют в высвобождении ctDNA, включают апоптоз и некроз из умирающих клеток или активное высвобождение из жизнеспособных опухолевых клеток. [7] [8] [9] [10] [11] Исследования как на людях (здоровых, так и на больных раком) [12] и на мышах с ксенотрансплантацией [13] показывают, что размер фрагментированной cfDNA преимущественно составляет 166 п.н., что соответствует длине ДНК, обернутой вокруг нуклеосомы плюс линкер. Фрагментация этой длины может указывать на апоптотическую фрагментацию ДНК , предполагая, что апоптоз может быть основным методом высвобождения ctDNA. Фрагментация cfDNA изменяется в плазме больных раком. [14] [15] В здоровой ткани инфильтрирующие фагоциты отвечают за очистку апоптотических или некротических клеточных остатков, включая cfDNA. [16] ctDNA у здоровых пациентов присутствует только в низких концентрациях, но более высокие уровни ctDNA у онкологических больных могут быть обнаружены с увеличением размеров опухоли. [17] Это, возможно, происходит из-за неэффективной инфильтрации иммунных клеток в опухолевые участки, что снижает эффективное очищение ctDNA из кровотока. [16] Сравнение мутаций в ctDNA и ДНК, выделенной из первичных опухолей тех же пациентов, выявило наличие идентичных генетических изменений, связанных с раком. [18] [19] Это привело к возможности использования ctDNA для более раннего выявления рака и последующего лечения. [20]
Когда кровь собирается в пробирки с ЭДТА и хранится, лейкоциты начинают лизироваться и высвобождать геномную ДНК дикого типа в образец в количествах, обычно во много раз превышающих количество ctDNA. [21] Это затрудняет обнаружение мутаций или других биомаркеров ctDNA. [22] Использование коммерчески доступных пробирок для стабилизации клеток может предотвратить или отсрочить лизис лейкоцитов, тем самым уменьшая эффект разбавления ctDNA. [23] Шервуд и др. продемонстрировали превосходное обнаружение мутаций KRAS в соответствующих образцах, собранных как в пробирках с ЭДТА K3, так и в пробирках Streck BCT. [23] Преимущества пробирок для стабилизации клеток могут быть реализованы в ситуации, когда кровь не может быть немедленно переработана в плазму.
Другие процедуры также могут уменьшить количество «загрязняющей» ДНК дикого типа и сделать обнаружение цДНК более осуществимым: [23]
Главное преимущество анализа ctDNA заключается в том, что он извлекается неинвазивным способом посредством забора крови. Для получения cfDNA или ctDNA обычно требуется сбор приблизительно 3 мл крови в пробирки, покрытые EDTA . Использование EDTA важно для снижения коагуляции крови. Фракции плазмы и сыворотки крови можно разделить с помощью этапа центрифугирования. ctDNA или cfDNA могут быть впоследствии извлечены из этих фракций. Хотя сыворотка, как правило, имеет более высокие уровни cfDNA, это в первую очередь связано с ДНК из лимфоцитов. [25] Высокие уровни загрязнения cfDNA являются неоптимальными, поскольку это может снизить чувствительность обнаружения ctDNA. Поэтому в большинстве исследований для выделения ctDNA используется плазма. Затем плазма снова обрабатывается центрифугированием для удаления остаточных неповрежденных клеток крови. Супернатант используется для извлечения ДНК, которое можно выполнить с помощью коммерчески доступных наборов. [ необходима цитата ]
Анализ ctDNA после извлечения требует использования различных методов амплификации и секвенирования. Эти методы можно разделить на две основные группы в зависимости от того, является ли целью опрос всех генов в нецелевом подходе или цель заключается в мониторинге определенных генов и мутаций в целевом подходе. [ необходима цитата ]
Для обнаружения новых мутаций в ДНК опухоли при мониторинге бремени болезни или отслеживании устойчивости к лекарствам могут потребоваться подходы секвенирования всего генома или всего экзома. [26] Нецелевые подходы также полезны в исследованиях для наблюдения за гетерогенностью опухоли или для обнаружения новых мишеней для лекарств. Однако, хотя нецелевые методы могут быть необходимы в определенных приложениях, они более дороги и имеют более низкое разрешение. Это затрудняет обнаружение редких мутаций или ситуаций, когда присутствуют низкие уровни ctDNA (например, минимальная остаточная болезнь). Кроме того, могут возникнуть проблемы с различением ДНК из опухолевых клеток и ДНК из нормальных клеток при использовании подхода с использованием всего генома. [ необходима цитата ]
Секвенирование всего генома или экзома обычно использует высокопроизводительные технологии секвенирования ДНК . Ограничение секвенирования только всем экзомом может снизить расходы и увеличить скорость, но за счет потери информации о мутациях в некодирующих регуляторных областях ДНК. [27] Хотя простое изучение полиморфизмов ДНК посредством секвенирования не отличает ДНК от опухолевых или нормальных клеток, эту проблему можно решить путем сравнения с контрольным образцом нормальной ДНК (например, ДНК, полученной с помощью буккального мазка ). Важно, что секвенирование всего генома и всего экзома полезно для первоначального обнаружения мутаций. Это дает информацию для использования более чувствительных целевых методов, которые затем можно использовать для целей мониторинга заболеваний.
Секвенирование всего генома позволяет восстановить структурные свойства cfDNA, размер фрагментов и их паттерны фрагментации. Эти уникальные паттерны могут быть важным источником информации для улучшения обнаружения ctDNA или локализации ткани происхождения этих фрагментов. [28] Выбор размера коротких фрагментов (<150bp) с помощью методов in vitro или in silico может улучшить восстановление мутаций и аберраций числа копий. [15]
Этот метод был первоначально разработан лабораторией Берта Фогельштейна , Луиса Диаса и Виктора Велкулеску в Университете Джонса Хопкинса . [29] В отличие от обычного кариотипирования , где краситель используется для окрашивания хромосомных полос с целью визуализации хромосом, цифровое кариотипирование использует последовательности ДНК локусов по всему геному для расчета вариации числа копий . [29] Вариации числа копий распространены при раковых заболеваниях и описывают ситуации, когда потеря гетерозиготности гена может привести к снижению функции из-за более низкой экспрессии или дупликации гена, что приводит к сверхэкспрессии.
После того, как весь геном секвенирован с использованием высокопроизводительного метода секвенирования, такого как Illumina HiSeq, к данным применяется персонализированный анализ перестроенных концов (PARE) для анализа хромосомных перестроек и транслокаций. Первоначально эта техника была разработана для анализа ДНК солидных опухолей, но была модифицирована для приложений ctDNA. [29]
Правильная эпигенетическая маркировка необходима для нормальной экспрессии генов и функционирования клеток, а аберрантные изменения в эпигенетических паттернах являются отличительной чертой рака. [30] Нормальный эпигенетический статус поддерживается в клетке, по крайней мере частично, посредством метилирования ДНК . [31] Измерение аберрантных паттернов метилирования в ctDNA возможно благодаря стабильному метилированию областей ДНК, называемых « островками CpG ». Метилирование ctDNA можно обнаружить с помощью обработки бисульфитом . Обработка бисульфитом химически преобразует неметилированные цитозины в урацил, оставляя метилированные цитозины немодифицированными. Затем ДНК секвенируют, и можно идентифицировать любые изменения в паттерне метилирования ДНК. Гидроксиметилирование ДНК является аналогично связанной меткой, которая, как было показано, является прогностическим маркером здоровых и болезненных состояний в cfDNA, включая рак. [32] [33] )
При целевом подходе секвенирование ctDNA может быть направлено на генетическую панель, созданную на основе мутационных горячих точек для интересующего рака. Это особенно важно для информирования о лечении в ситуациях, когда мутации идентифицированы в мишенях, поддающихся лечению лекарственными средствами. [27] Персонализация целевого анализа ctDNA для каждого пациента также возможна путем объединения жидких биопсий со стандартными первичными биопсиями тканей. Полногеномное или полноэкзомное секвенирование первичной биопсии опухоли позволяет обнаружить генетические мутации, специфичные для опухоли пациента, и может использоваться для последующего целевого секвенирования ctDNA пациента. Самая высокая чувствительность обнаружения ctDNA достигается посредством целевого секвенирования определенных однонуклеотидных полиморфизмов (SNP). Часто мутирующие гены, такие как онкогены, которые обычно имеют мутации горячих точек, являются хорошими кандидатами для подходов целевого секвенирования. Напротив, большинство генов-супрессоров опухолей имеют широкий спектр возможных мутаций потери функции по всему гену и, как таковые, не подходят для целевого секвенирования. [ необходима цитата ]
Целевые подходы имеют преимущество амплификации ctDNA посредством полимеразной цепной реакции (ПЦР) или цифровой ПЦР . Это особенно важно при анализе ctDNA не только потому, что в кровотоке циркулирует относительно низкий уровень ДНК, но и потому, что ctDNA составляет небольшую долю от общего количества выделенной бесклеточной ДНК. [27] Таким образом, амплификация интересующих областей может радикально повысить чувствительность обнаружения ctDNA. Однако амплификация посредством ПЦР может вносить ошибки, учитывая присущий ДНК-полимеразам уровень ошибок. Ошибки, вносимые во время секвенирования, также могут снижать чувствительность обнаружения мутаций ctDNA. [ необходима цитата ]
Капельная цифровая ПЦР (ddPCR) происходит от цифровой полимеразной цепной реакции , первоначально названной группой Берта Фогельштейна в Университете Джонса Хопкинса . Капельная цифровая ПЦР использует генератор капель для разделения отдельных фрагментов ДНК на капли с использованием эмульсии масло/вода. Затем в каждой капле происходят отдельные полимеразные цепные реакции с использованием выбранных праймеров против областей ctDNA и продолжается до конечной точки. Наличие интересующих последовательностей измеряется флуоресцентными зондами, которые связываются с амплифицированной областью. ddPCR позволяет проводить высококоличественную оценку частот аллелей и мутантов в ctDNA, но ограничена количеством флуоресцентных зондов, которые можно использовать в одном анализе (до 5). [34] Чувствительность анализа может варьироваться в зависимости от количества анализируемой ДНК и составляет около 1 из 10 000. [34] Специфичность следует повышать за счет использования либо модифицированных зондов, связывающих малую бороздку (MGB), либо альтернативных вариантов, таких как запертые нуклеиновые кислоты (LNA).
Бусины, эмульгирование, амплификация и магниты (BEAMing) — это метод, который основан на цифровой ПЦР капель для идентификации мутаций в ctDNA с помощью проточной цитометрии. [35] После того, как ctDNA извлечена из крови, ПЦР выполняется с праймерами, разработанными для нацеливания на интересующие регионы. Эти праймеры также содержат определенные последовательности ДНК или метки. Амплифицированная ДНК смешивается с покрытыми стрептавидином магнитными шариками и эмульгируется в капли. Биотинилированные праймеры, разработанные для связывания с метками, используются для амплификации ДНК. Биотинилирование позволяет амплифицированной ДНК связываться с магнитными шариками, которые покрыты стрептавидином. После завершения ПЦР связанные с ДНК шарики разделяются с помощью магнита. Затем ДНК на шариках денатурируют и дают ей гибридизоваться с флуоресцентными олигонуклеотидами, специфичными для каждой матрицы ДНК. Затем полученные комплексы шарик-ДНК анализируются с помощью проточной цитометрии. Эта техника способна захватывать частоты аллелей и мутаций благодаря сопряжению с ddPCR. Однако, в отличие от ddPCR, большее количество последовательностей ДНК может быть исследовано благодаря гибкости использования флуоресцентно связанных зондов. Еще одним преимуществом этой системы является то, что изолированная ДНК также может быть использована для секвенирования ниже по течению. [36] Чувствительность составляет от 1,6 в 10 4 до 4,3 в 10 5 . [34]
Персонализированное профилирование рака с помощью глубокого секвенирования (CAPP-Seq) было первоначально описано группами Эша Ализаде и Максимилиана Дина в Стэнфордском университете . Этот метод использует биотинилированные олигонуклеотидные селекторные зонды для нацеливания последовательностей ДНК, имеющих отношение к обнаружению ctDNA. [37] Публично доступные базы данных по раку были использованы для создания библиотеки зондов против рецидивирующих мутаций при раке путем вычисления их индекса рецидива. Протокол был оптимизирован для низких уровней ДНК, наблюдаемых в коллекции ctDNA. Затем изолированная ДНК подвергается глубокому секвенированию для повышения чувствительности. Этот метод позволяет исследовать сотни участков ДНК. Сообщается, что чувствительность обнаружения ctDNA с помощью CAPP-Seq составляет 2,5 молекулы на 1 000 000. [38]
Глубокое секвенирование меченых ампликонов (TAM-Seq) позволяет проводить целевое секвенирование целых генов для обнаружения мутаций в ctDNA. [39] Сначала выполняется общий этап амплификации с использованием праймеров, которые охватывают весь интересующий ген в секциях 150-200bp. Затем используется система микрофлюидики для прикрепления адаптеров с уникальным идентификатором к каждому ампликону для дальнейшей амплификации ДНК в параллельных одноплексных реакциях. Было показано, что эта методика успешно идентифицирует мутации, разбросанные в гене-супрессоре опухоли TP53 у пациентов с прогрессирующим раком яичников. Чувствительность этой методики составляет 1 из 50.
Безопасное секвенирование (Safe-Seq) было первоначально описано Бертом Фогельштейном и его группой в Университете Джонса Хопкинса . Safe-Seq снижает частоту ошибок массового параллельного секвенирования, чтобы повысить чувствительность к редким мутантам. [40] Это достигается путем добавления последовательности уникального идентификатора (UID) к каждому шаблону ДНК. Затем ДНК амплифицируется с использованием добавленных UID и секвенируется. Все молекулы ДНК с одинаковым UID (семейство UID) должны иметь одинаковую сообщаемую последовательность ДНК, поскольку они были амплифицированы из одной молекулы. Однако мутации могут быть введены посредством амплификации, или неправильные назначения оснований могут быть вызваны на этапах секвенирования и анализа. Наличие UID позволяет отделить эти методологические ошибки от истинных мутаций ctDNA. Мутация считается «супермутантом», если 95% секвенированных прочтений совпадают. Чувствительность этого подхода составляет 9 на 1 миллион. [34]
Дуплексное секвенирование является улучшением одиночных UID, добавленных в технике Safe-Seq. [41] В дуплексном секвенировании рандомизированная двухцепочечная ДНК действует как уникальные метки и прикрепляется к инвариантному спейсеру. Метки прикрепляются к обоим концам фрагмента ДНК (α и β метки), что приводит к двум уникальным шаблонам для ПЦР — одна цепь с α-меткой на 5'-конце и β-меткой на 3'-конце, а другая цепь с β-меткой на 5'-конце и α-меткой на 3'-конце. Затем эти фрагменты ДНК амплифицируются с помощью праймеров против инвариантных последовательностей меток. Амплифицированная ДНК секвенируется и анализируется. ДНК с дуплексными адаптерами сравнивается, и мутации принимаются только в том случае, если между обеими цепями есть консенсус. Этот метод учитывает как ошибки секвенирования, так и ошибки ранней стадии амплификации ПЦР. Чувствительность подхода к обнаружению мутантов составляет 1 из 10^7.
Интегрированное цифровое подавление ошибок (iDES) улучшает анализ CAPP-Seq ctDNA, чтобы уменьшить ошибки и, следовательно, повысить чувствительность обнаружения. [38] Сообщалось в 2016 году, что iDES объединяет CAPP-Seq с технологией дуплексного штрихкодирования секвенирования и с вычислительным алгоритмом, который удаляет стереотипные ошибки, связанные с шагом гибридизации CAPP-Seq. Метод также интегрирует дуплексное секвенирование, где это возможно, и включает методы для более эффективного восстановления дуплекса из бесклеточной ДНК. Чувствительность этой улучшенной версии CAPP-Seq составляет 4 на 100 000 копий.
Исследования по секвенированию всего генома проводились на ctDNA, присутствующей у разных пациентов с резистентным к лечению раком предстательной железы (в подавляющем большинстве случаев, а в некоторых случаях и метастатическим ), раком мочевого пузыря и у контрольных пациентов, у которых эта ДНК не была обнаружена, включая соматические мутации и структурные перестройки в их геномах.
Этот новый и многообещающий метод предоставил информацию о резистентности к лечению ингибиторами сигнализации андрогеновых рецепторов , внутриопухолевой гетерогенности (благодаря филогенетической эволюции и молекулярной хронологии), хромосомной нестабильности , вкладе ctDNA в метастазирование через глобальные транскриптомные паттерны (с учетом нуклеосом, присутствующих в сайтах начала транскрипции (TSS) и сайтах связывания AR (ARB). Таким образом, можно наблюдать геномную и транскриптомную эволюцию ctDNA, проводимую у живых пациентов, у которых развивается резистентность к лечению, поэтому секвенирование ctDNA является элементарным для выявления клинически значимых различий в фенотипе рака и для того, чтобы увидеть, как терапия влияет на пациентов. Кроме того, относительная однородность изменений генов-драйверов среди метастазов подтверждает, что геномные и функциональные изменения при раке предстательной железы являются общими для ctDNA и ткани.
Это делает ctDNA мощным новым инструментом для обнаружения генетических мутаций в геномном масштабе у пациентов, страдающих метастатическим раком, для наблюдения за клинической значимостью клонального состава этих опухолей, чтобы лучше понять контроль рака. Эта субклональная реконструкция, основанная на ctDNA благодаря секвенированию всего генома, представляет собой уникальный набор проблем и возможностей для научных исследований в области онкологии. Кроме того, серийная ctDNA выявляет обусловленный лечением отбор для аугментации андрогенных рецепторов, поскольку она увеличивает размерность данных.
Необходимы дальнейшие исследования для понимания того, как расположение и размер метастазов в зависимости от опухолевой нагрузки влияют на циркулирующую ДНК опухоли, а также для выбора новых методов для выбора других поражений, которые отражают клинически доминирующее заболевание. [42]
Одной из проблем использования ctDNA в качестве биомаркера рака является то, можно ли отличить ctDNA с помощью cfDNA от нормальных клеток. cfDNA выделяется незлокачественными клетками во время нормального клеточного оборота, а также во время таких процедур, как хирургия , радиотерапия или химиотерапия . Считается, что лейкоциты являются основными источниками cfDNA в сыворотке. [27]
Клиническая полезность ctDNA для обнаружения первичного заболевания частично ограничена чувствительностью современных технологий для обнаружения небольших опухолей с низким уровнем присутствующей ctDNA и априори неизвестными соматическими мутациями. [17] [34]
Доказательства заболевания традиционными методами визуализации, такими как КТ , ПЭТ или МРТ, могут отсутствовать после резекции опухоли. Таким образом, анализ ctDNA представляет собой потенциальный путь для обнаружения минимальной остаточной болезни (MRD) и, следовательно, возможности рецидива опухоли в случаях, когда объемные опухоли отсутствуют при обычных методах визуализации. [17] Сравнение обнаружения MRD с помощью КТ по сравнению с ctDNA ранее проводилось у лиц со II стадией рака толстой кишки; в этом исследовании исследователи смогли обнаружить ctDNA у лиц, у которых не было признаков клинической злокачественности с помощью КТ, что предполагает, что обнаружение ctDNA имеет большую чувствительность для оценки MRD. [27] Однако авторы признают, что анализ ctDNA не лишен ограничений; образцы плазмы, собранные после операции, могли предсказать рецидив только через 36 месяцев в 48% случаев. [27] Впоследствии были разработаны анализы ctDNA как для колоректального рака [43] , так и для меланомы . [44]
Вопрос о том, можно ли использовать измерение количества или качества ctDNA для определения результатов у людей с раком, был предметом исследования. По состоянию на 2015 год это было очень неопределенно. [45] Хотя некоторые исследования показали тенденцию к более высоким уровням ctDNA у людей с метастатическим раком высокой стадии, нагрузка ctDNA не всегда коррелирует с традиционной стадией рака. [34] По состоянию на 2013 год казалось маловероятным, что ctDNA будет иметь клиническую полезность в качестве единственного предиктора прогноза. [46]
Возникновение опухолей, устойчивых к лекарственным препаратам, из-за внутриопухолевой и межопухолевой гетерогенности является проблемой эффективности лечения. Незначительный генетический клон в опухоли может расширяться после лечения, если он несет мутацию, устойчивую к лекарственным препаратам. Первоначальные биопсии могут пропустить эти клоны из-за низкой частоты или пространственного разделения клеток в опухоли. Например, поскольку биопсия забирает только небольшую часть опухоли, клоны, которые находятся в другом месте, могут остаться незамеченными. Это может ввести в заблуждение исследования, которые фокусируются на изучении роли гетерогенности опухоли в прогрессировании рака и рецидиве. Использование ctDNA в исследованиях может развеять эти опасения, поскольку оно может обеспечить более репрезентативный «скриншот» генетического разнообразия рака как в первичных, так и в метастатических участках. Например, было показано, что ctDNA полезна при изучении клональной эволюции рака пациента до и после схем лечения. [47] Раннее выявление рака все еще остается сложной задачей, но недавний прогресс в анализе эпигенетических особенностей cfDNA или паттерна фрагментации раскрывает улучшение чувствительности жидкой биопсии. [28] Кроме того, анализ ctDNA является новым инструментом для понимания клонального состава метастатических опухолей, обнаружения различных мутаций в геномном масштабе, изучения субклонального разнообразия, которое влияет на прогноз заболевания, поскольку могут быть обнаружены различные резистентные фенотипы и появление новых механизмов геномной и транскриптомной резистентности к лечению. [42]
Внедрение ctDNA в клиническую практику во многом затруднено из-за отсутствия стандартизированных методов обработки и анализа ctDNA. Стандартизация методов сбора образцов (включая время сбора), последующей обработки (извлечение и амплификация ДНК), количественной оценки и проверки должны быть установлены до того, как анализ ctDNA станет рутинным клиническим анализом. Кроме того, может потребоваться создание панели «стандартных» опухолеассоциированных биомаркеров, учитывая разрешение текущих методов секвенирования и обнаружения ctDNA. Секвенирование опухолеспецифических аберраций из образцов плазмы также может помочь исключить загрязняющую cfDNA из анализа; повышенные уровни cfDNA из нормальных клеток могут быть отнесены к причинам, не связанным с раком. [27] Эти методы секвенирования также могут определять клональную эволюцию рака, гетерогенность опухоли и механизмы устойчивости к лекарствам, участвующие в раке. [42]