Молекулярное моделирование охватывает все методы, теоретические и вычислительные, используемые для моделирования или имитации поведения молекул . [1] Эти методы используются в области вычислительной химии , дизайна лекарств , вычислительной биологии и материаловедения для изучения молекулярных систем, начиная от небольших химических систем и заканчивая большими биологическими молекулами и материальными сборками. Простейшие расчеты можно выполнить вручную, но для молекулярного моделирования любой системы разумного размера неизбежно требуются компьютеры. Общей чертой методов молекулярного моделирования является описание молекулярных систем на атомистическом уровне. Это может включать в себя рассмотрение атомов как мельчайших отдельных единиц ( подход молекулярной механики ) или явное моделирование протонов и нейтронов с их кварками, антикварками и глюонами и электронов с их фотонами ( подход квантовой химии ).
Молекулярная механика является одним из аспектов молекулярного моделирования, поскольку она предполагает использование классической механики ( механики Ньютона ) для описания физической основы моделей. Молекулярные модели обычно описывают атомы (ядро и электроны вместе) как точечные заряды с соответствующей массой. Взаимодействия между соседними атомами описываются пружинистыми взаимодействиями (представляющими химические связи ) и силами Ван-дер-Ваальса . Для описания последнего обычно используют потенциал Леннарда -Джонса . Электростатические взаимодействия рассчитываются на основе закона Кулона . Атомам присваиваются координаты в декартовом пространстве или во внутренних координатах , а также им могут быть присвоены скорости в динамическом моделировании. Скорости атомов связаны с температурой системы, макроскопической величиной. Коллективное математическое выражение называется потенциальной функцией и связано с внутренней энергией системы (U), термодинамической величиной, равной сумме потенциальной и кинетической энергий. Методы, которые минимизируют потенциальную энергию, называются методами минимизации энергии (например, наискорейший спуск и сопряженный градиент ), а методы, моделирующие поведение системы с распространением времени, называются молекулярной динамикой .
Эта функция, называемая потенциальной функцией , вычисляет потенциальную энергию молекулы как сумму энергетических членов, которые описывают отклонение длин связей, валентных и торсионных углов от равновесных значений, а также членов для несвязанных пар атомов, описывающих ван дер Ваальса и электростатические взаимодействия. Набор параметров, состоящий из равновесных длин связей, валентных углов, значений частичных зарядов, силовых констант и параметров Ван-дер-Ваальса, вместе называется силовым полем . Различные реализации молекулярной механики используют разные математические выражения и разные параметры потенциальной функции . [2] Общие силовые поля, используемые сегодня, были разработаны с использованием химической теории, экспериментальных справочных данных и квантовых расчетов высокого уровня. Метод, называемый минимизацией энергии, используется для поиска положений нулевого градиента для всех атомов, другими словами, локального минимума энергии. Состояния с более низкой энергией более стабильны и обычно исследуются из-за их роли в химических и биологических процессах. С другой стороны, моделирование молекулярной динамики рассчитывает поведение системы как функцию времени. Это включает в себя решение законов движения Ньютона, главным образом второго закона . Интегрирование законов движения Ньютона с использованием различных алгоритмов интегрирования приводит к траекториям атомов в пространстве и времени. Сила, действующая на атом, определяется как отрицательный градиент функции потенциальной энергии. Метод минимизации энергии полезен для получения статической картины для сравнения состояний подобных систем, а молекулярная динамика дает информацию о динамических процессах с присущим учетом температурных эффектов.
Молекулы можно моделировать либо в вакууме, либо в присутствии растворителя, например воды. Моделирование систем в вакууме называется моделированием газовой фазы , а моделирование, включающее присутствие молекул растворителя, называется явным моделированием растворителя . В другом типе моделирования влияние растворителя оценивается с использованием эмпирического математического выражения; это называется неявным сольватационным моделированием.
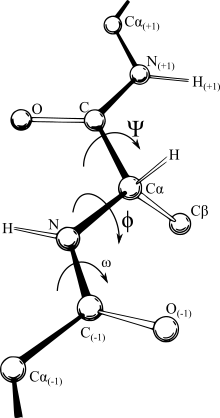
Большинство силовых полей зависят от расстояния, что дает наиболее удобное выражение для этих декартовых координат. Однако сравнительно жесткий характер связей, которые возникают между конкретными атомами и, по сути, определяют, что подразумевается под обозначением « молекула» , делают внутреннюю систему координат наиболее логичным представлением. В некоторых областях представление IC (длина связи, угол между связями и угол закручивания связи, как показано на рисунке) называется Z-матрицей или представлением угла кручения. К сожалению, непрерывные движения в декартовом пространстве часто требуют прерывистых угловых ветвей во внутренних координатах, что затрудняет работу с силовыми полями во внутреннем координатном представлении, и, наоборот, простое перемещение атома в декартовом пространстве может не быть прямой траекторией из-за к запретам взаимосвязанных связей. Таким образом, программы вычислительной оптимизации очень часто переключаются между представлениями во время своих итераций. Это может существенно повлиять на время расчета самого потенциала, а в молекулах с длинными цепями привести к кумулятивной числовой неточности. Хотя все алгоритмы преобразования дают математически идентичные результаты, они различаются по скорости и числовой точности. [3] В настоящее время самым быстрым и точным преобразованием в декартову систему является метод естественного расширения системы отсчета (NERF). [3]
Методы молекулярного моделирования регулярно используются для исследования структуры, динамики, свойств поверхности и термодинамики неорганических, биологических и полимерных систем. Сегодня в базах данных легко доступно большое количество молекулярных моделей силового поля. [4] [5] Типы биологической активности, которые были исследованы с помощью молекулярного моделирования, включают сворачивание белков , ферментативный катализ , стабильность белков, конформационные изменения, связанные с биомолекулярными функциями, а также молекулярное распознавание белков, ДНК и мембранных комплексов. [6]