Кольцевая хромосома — хромосома у бактерий , архей , митохондрий и хлоропластов , имеющая форму молекулы кольцевой ДНК, в отличие от линейной хромосомы большинства эукариот .
Большинство хромосом прокариот содержат кольцевую молекулу ДНК. Это имеет главное преимущество, поскольку не имеет свободных концов ( теломер ) ДНК . Напротив, большинство эукариот имеют линейную ДНК, требующую сложных механизмов для поддержания стабильности теломер и репликации ДНК . Однако кольцевая хромосома имеет тот недостаток, что после репликации две кольцевые хромосомы потомства могут оставаться взаимосвязанными или запутанными, и их необходимо расцепить, чтобы каждая клетка унаследовала одну полную копию хромосомы во время деления клетки .

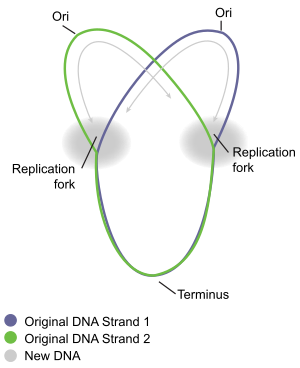
Кольцевая репликация хромосом бактерий лучше всего изучена на примере хорошо изученных бактерий Escherichia coli и Bacillus subtilis . Репликация хромосом происходит в три основных этапа: инициация, удлинение и терминация. Стадия инициации начинается с упорядоченной сборки белков-инициаторов в исходной области хромосомы, называемой oriC . Эти этапы сборки регулируются, чтобы гарантировать, что репликация хромосомы происходит только один раз в каждом клеточном цикле. Во время фазы удлинения репликации ферменты , которые были собраны в oriC во время инициации, продвигаются вдоль каждого плеча ( реплихоры ) хромосомы в противоположных направлениях от oriC, реплицируя ДНК для создания двух идентичных копий. Этот процесс известен как двунаправленная репликация. Вся совокупность молекул, участвующих в репликации ДНК на каждом плече, называется реплисомой . В авангарде реплисомы находится ДНК-хеликаза , которая раскручивает две нити ДНК, создавая движущуюся репликационную вилку . Две раскрученные одиночные нити ДНК служат матрицами для ДНК-полимеразы , которая движется с геликазой (вместе с другими белками) для синтеза комплементарной копии каждой нити. Таким образом, создаются две идентичные копии исходной ДНК. В конце концов, две репликационные вилки, движущиеся по кольцевой хромосоме, встречаются в определенной зоне хромосомы, примерно напротив oriC, называемой терминальной областью. Затем ферменты удлинения разбираются, и две «дочерние» хромосомы разделяются до завершения деления клетки.

Источник репликации E. coli , называемый oriC, состоит из последовательностей ДНК , которые распознаются белком DnaA , который высококонсервативен среди различных видов бактерий . Связывание DnaA с источником инициирует регулируемое привлечение других ферментов и белков , что в конечном итоге приведет к созданию двух полных реплисом для двунаправленной репликации. [1]
Элементы последовательности ДНК в oriC , которые важны для его функции, включают DnaA-боксы, 9-мерный повтор с высококонсервативной консенсусной последовательностью 5' – TTATCCACA – 3', [2] , которые распознаются белком DnaA. Белок DnaA играет решающую роль в инициации репликации хромосомной ДНК. [3] Связанный с АТФ и с помощью бактериальных гистон -подобных белков [HU] DnaA затем раскручивает богатую АТ область вблизи левой границы oriC , которая несет три 13-мерных мотива, [4] и открывает двухцепочечную ДНК для входа других репликационных белков. [5]
Этот регион также содержит четыре последовательности элементов раскручивания ДНК «GATC» , которые распознаются ДНК-аденинметилазой (Dam), ферментом, который модифицирует основание аденина, когда эта последовательность неметилирована или полуметилирована. Метилирование аденинов важно , поскольку оно изменяет конформацию ДНК, способствуя разделению нитей, [6] и, по-видимому, этот регион ori C имеет естественную тенденцию к раскручиванию. [7]
Затем DnaA привлекает репликативную геликазу DnaB из комплекса DnaB-DnaC в раскрученную область для формирования комплекса препрайминга. [8] После того , как DnaB перемещается в вершину каждой репликационной вилки, геликаза раскручивает родительскую ДНК и на мгновение взаимодействует с праймазой . [9]
Для продолжения репликации ДНК необходимы одноцепочечные связывающие белки, которые не позволяют одиночным цепям ДНК образовывать вторичные структуры и предотвращают их повторный отжиг . Кроме того, ДНК-гираза необходима для снятия топологического напряжения, создаваемого действием геликазы DnaB.
Когда репликативная вилка движется по кругу, образуется структура, имеющая форму греческой буквы тета Ө. Джон Кейрнс продемонстрировал тета-структуру репликации хромосом E. coli в 1963 году, используя инновационный метод визуализации репликации ДНК. В своем эксперименте он радиоактивно пометил хромосому, выращивая свои культуры в среде, содержащей 3H- тимидин . Нуклеозидное основание было равномерно включено в бактериальную хромосому. Затем он изолировал хромосомы, осторожно лизируя клетки, и помещал их на сетку электронного микрографа (ЭМ), которую он экспонировал рентгеновской пленкой в течение двух месяцев. Этот эксперимент наглядно демонстрирует модель тета-репликации кольцевых бактериальных хромосом. [10]
Как описано выше, репликация бактериальных хромосом происходит двунаправленным образом. Это было впервые продемонстрировано путем специфической маркировки реплицирующихся бактериальных хромосом радиоактивными изотопами . Затем области ДНК, подвергающиеся репликации во время эксперимента, были визуализированы с помощью авторадиографии и изучения проявленной пленки под микроскопом. Это позволило исследователям увидеть, где происходит репликация. Первые убедительные наблюдения двунаправленной репликации были получены в ходе исследований B. subtilis. [11] Вскоре после этого было показано, что хромосома E. coli также реплицируется двунаправленно. [12]
Холофермент ДНК-полимеразы III E. coli представляет собой комплекс 900 кДа, обладающий по существу димерной структурой. Каждая мономерная единица имеет каталитическое ядро, димеризационную субъединицу и компонент процессивности . [13] ДНК-полимераза III использует один набор своих основных субъединиц для непрерывного синтеза ведущей цепи , в то время как другой набор основных субъединиц циклически переходит от одного фрагмента Оказаки к следующему на петлевой отстающей цепи . Синтез ведущей цепи начинается с синтеза короткого праймера РНК в точке начала репликации ферментом Праймазой ( белок DnaG ).
Затем к этому праймеру добавляются дезоксинуклеотиды с помощью одного димера ДНК-полимеразы III в интегрированном комплексе с геликазой DnaB. Синтез ведущей цепи затем продолжается непрерывно, в то время как ДНК одновременно раскручивается на репликационной вилке. Напротив, синтез отстающей цепи осуществляется короткими фрагментами Оказаки. Сначала праймер РНК синтезируется праймазой, и, как и при синтезе ведущей цепи, ДНК Pol III связывается с праймером РНК и добавляет дезоксирибонуклеотиды .
Когда синтез фрагмента Оказаки завершен, репликация останавливается, и основные субъединицы ДНК Pol III диссоциируют от скользящего β-зажима [скользящий хлопок B является процессивной субъединицей ДНК Pol III]. [14] РНК-праймер удаляется и заменяется ДНК ДНК-полимеразой I [которая также обладает корректирующей экзонуклеазной активностью], а оставшийся надрез запечатывается ДНК-лигазой , которая затем лигирует эти фрагменты, образуя отстающую цепь.
Значительная часть (10-15%) репликативных вилок, возникающих в oriC, сталкивается с повреждением ДНК или разрывом цепи, когда клетки выращиваются в обычных лабораторных условиях (без экзогенной обработки, повреждающей ДНК). [15] Встречающиеся повреждения ДНК обычно обрабатываются ферментами рекомбинационной репарации, что позволяет продолжить прогрессирование репликативной вилки. [15]

Терминация — это процесс слияния репликационных вилок и разборки реплисом с образованием двух отдельных и полных молекул ДНК . Это происходит в терминальной области, примерно напротив oriC на хромосоме (рис. 5). Терминальная область содержит несколько участков терминации репликации ДНК, или участков «Ter». Специальный белок «терминатора репликации» должен быть связан с сайтом Ter, чтобы он остановил репликацию. Каждый сайт Ter имеет полярность действия, то есть он останавливает репликационную вилку, приближающуюся к сайту Ter с одного направления, но позволяет вилке беспрепятственно двигаться через сайт Ter с другого направления. Расположение сайтов Ter образует две противоположные группы, которые заставляют две вилки встречаться друг с другом в области, которую они охватывают. Такое расположение называется «ловушкой репликационной вилки». [16]
Сайты Ter специфически взаимодействуют с белком-терминатором репликации, называемым Tus в E. coli . [17] Комплекс Tus-Ter препятствует активности раскручивания ДНК DnaB в зависимости от ориентации. [18]
Репликация ДНК, разделяющая противоположные репликационные вилки, оставляет завершенные хромосомы соединенными в виде « катенанов » или топологически взаимосвязанных кругов. Круги связаны не ковалентно, а механически, поскольку они переплетены и каждый ковалентно замкнут. Сцепленные круги требуют действия топоизомераз для разделения кругов (декатенации). В E. coli ДНК-топоизомераза IV играет главную роль в разделении сцепленных хромосом, временно разрывая обе нити ДНК одной хромосомы и позволяя другой хромосоме пройти через разрыв.
Была некоторая путаница относительно роли ДНК-гиразы в декатенации. Чтобы определить номенклатуру, существует два типа топоизомераз: тип I производит временные одноцепочечные разрывы в ДНК, а тип II производит временные двухцепочечные разрывы. В результате фермент типа I удаляет супервитки из ДНК по одной за раз, тогда как фермент типа II удаляет супервитки по две за раз. Топо I как прокариот , так и эукариот является топоизомеразой типа I. Эукариотическая топо II, бактериальная гираза и бактериальная топо IV относятся к типу II.
ДНК-гираза также обладает активностью топоизомеразы типа II; таким образом, поскольку она является гомологом топоизомеразы IV (также обладающей активностью топоизомеразы II), мы ожидаем сходства в функциях двух белков. Первоначальная роль ДНК-гиразы заключается в том, чтобы вводить отрицательные супервитки в ДНК, тем самым ослабляя положительные супервитки, которые образуются во время репликации ДНК. Топоизомераза IV также ослабляет положительные супервитки, поэтому ДНК-гираза и топоизомераза IV играют почти одинаковую роль в удалении положительных супервитков перед транслоцирующей ДНК-полимеразой, позволяя репликации ДНК продолжаться без помех со стороны топологического напряжения. [19]
ДНК-гираза — не единственный фермент, ответственный за декатенацию. В эксперименте Зехидриха , Ходурского и Коццарелли в 1997 году было обнаружено, что топоизомераза IV является единственной важной декатеназой промежуточных продуктов репликации ДНК у бактерий. [20] Когда была ингибирована только ДНК-гираза, большинство катенанов были несвязанными. Однако, когда была ингибирована только топоизомераза IV, декатенация была почти полностью заблокирована. Это говорит о том, что топоизомераза IV является основным белком для декатенации взаимосвязанных хромосом in vivo , при этом ДНК-гираза играет второстепенную роль.
Несколько групп бактерий, включая Brucella , Paracoccus denitrificans и Vibrio , имеют несколько кольцевых хромосом.
В основу статьи положена статья Имальды Девапаранам и Дэвида Трайба, предоставленная в соответствии с условиями лицензирования CC by SA из университетского курса на кафедре микробиологии и иммунологии Мельбурнского университета в 2007 году. [ необходима ссылка ] В этой статье использованы материалы из статьи Citizendium «Репликация кольцевой бактериальной хромосомы», которая лицензирована в соответствии с лицензией Creative Commons Attribution-ShareAlike 3.0 Unported , но не в соответствии с лицензией GFDL .