Инвертированный повтор (или IR ) представляет собой одноцепочечную последовательность нуклеотидов , за которой следует ее обратный комплемент . [1] Промежуточная последовательность нуклеотидов между исходной последовательностью и обратным комплементом может иметь любую длину, включая нулевую. Например, 5'---TTACGnnnnnn CGTAA---3' представляет собой инвертированную повторную последовательность. Когда промежуточная длина равна нулю, составная последовательность является палиндромной последовательностью . [2]
Как инвертированные повторы, так и прямые повторы представляют собой типы нуклеотидных последовательностей , которые встречаются повторно. Эти повторяющиеся последовательности ДНК часто варьируются от пары нуклеотидов до целого гена , в то время как близость повторяющихся последовательностей варьируется между широко распространенными и простыми тандемными массивами . [3] Короткие тандемные повторяющиеся последовательности могут существовать в виде всего нескольких копий в небольшом регионе до тысяч копий, рассеянных по всему геному большинства эукариот . [4] Повторяющиеся последовательности с примерно 10–100 парами оснований известны как минисателлиты , в то время как более короткие повторяющиеся последовательности, имеющие в основном 2–4 пары оснований, известны как микросателлиты . [5] Наиболее распространенные повторы включают динуклеотидные повторы, которые имеют основания AC на одной цепи ДНК и GT на комплементарной цепи. [3] Некоторые элементы генома с уникальными последовательностями функционируют как экзоны , интроны и регуляторная ДНК. [6] Хотя наиболее известными локусами повторяющихся последовательностей являются центромера и теломера , [6] большая часть повторяющихся последовательностей в геноме обнаружена среди некодирующей ДНК . [5]
Инвертированные повторы имеют ряд важных биологических функций. Они определяют границы в транспозонах и указывают области, способные к самокомплементарному спариванию оснований (области в пределах одной последовательности, которые могут образовывать пары оснований друг с другом). Эти свойства играют важную роль в нестабильности генома [7] и способствуют не только клеточной эволюции и генетическому разнообразию [8], но также мутациям и болезням [9] . Для того чтобы подробно изучить эти эффекты, был разработан ряд программ и баз данных, помогающих в обнаружении и аннотации инвертированных повторов в различных геномах.
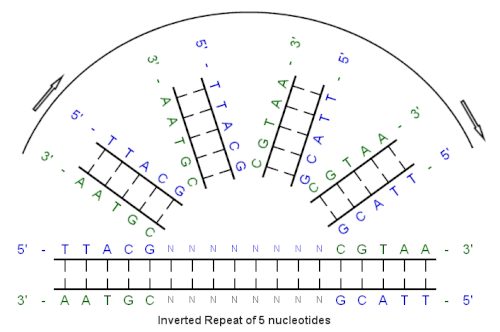
Начиная с этой начальной последовательности: 5'-TTACG-3'
Комплемент, созданный путем спаривания оснований, выглядит следующим образом: 3'-AATGC-5'
Обратный комплемент: 5'-CGTAA-3'
А инвертированная повторяющаяся последовательность выглядит так: 5'---TTACGnnnnnn CGTAA---3'
«nnnnnn» представляет собой любое количество промежуточных нуклеотидов.
Прямой повтор происходит, когда последовательность повторяется с тем же шаблоном ниже по течению. [1] Нет инверсии и обратного комплемента, связанных с прямым повтором. Нуклеотидная последовательность, написанная жирным шрифтом, обозначает повторяющуюся последовательность. Она может иметь или не иметь промежуточных нуклеотидов.
С лингвистической точки зрения типичный прямой повтор можно сравнить с рифмованием, например, «time on ad ime ».
Прямой повтор без промежуточных нуклеотидов между начальной последовательностью и ее копией ниже по течению — это тандемный повтор . Нуклеотидная последовательность, выделенная жирным шрифтом, обозначает повторяющуюся последовательность.
С лингвистической точки зрения типичный тандемный повтор можно сравнить с заиканием или намеренным повторением слов, например, «пока-пока».
Инвертированная повторяющаяся последовательность без промежуточных нуклеотидов между исходной последовательностью и ее нисходящим обратным комплементом является палиндромом . [1]
ПРИМЕР:
Шаг 1: начать с инвертированного повтора: 5' TTACGnnnnnnCGTAA 3'
Шаг 2: удалить промежуточные нуклеотиды: 5' TTACGCGTAA 3'
Эта полученная последовательность является палиндромной, поскольку она является обратным комплементом самой себя. [1]
Разнообразные повторы по всему геному происходят из мобильных элементов , которые, как теперь понимают, «прыгают» по разным геномным участкам, не перенося свои исходные копии. [10] Последующее перемещение одних и тех же последовательностей в течение многочисленных поколений обеспечивает их множественность по всему геному. [10] Ограниченная рекомбинация последовательностей между двумя различными элементами последовательности, известная как консервативная сайт-специфическая рекомбинация (CSSR), приводит к инверсиям сегмента ДНК, основанным на расположении последовательностей распознавания рекомбинации на донорской ДНК и реципиентной ДНК. [10] Опять же, ориентация двух рекомбинирующих сайтов в молекуле донорской ДНК относительно асимметрии промежуточных последовательностей расщепления ДНК, известной как область кроссинговера, имеет решающее значение для образования либо инвертированных повторов, либо прямых повторов. [10] Таким образом, рекомбинация, происходящая в паре инвертированных сайтов, инвертирует последовательность ДНК между двумя сайтами. [10] Были обнаружены очень стабильные хромосомы со сравнительно меньшим количеством инвертированных повторов, чем прямых повторов, что предполагает связь между стабильностью хромосом и количеством повторов. [11]
Терминальные инвертированные повторы были обнаружены в ДНК различных эукариотических транспозонов, хотя их источник остается неизвестным. [12] Инвертированные повторы в основном обнаруживаются в точках начала репликации клеточного организма и органелл, которые варьируются от фаговых плазмид, митохондрий и эукариотических вирусов до клеток млекопитающих. [13] Точки начала репликации фага G4 и других родственных фагов включают сегмент из почти 139 нуклеотидных оснований, которые включают три инвертированных повтора, которые необходимы для начала репликации. [13]
В значительной степени части нуклеотидных повторов довольно часто наблюдаются как часть редких комбинаций ДНК. [14] Три основных повтора, которые в основном встречаются в определенных конструкциях ДНК, включают в себя очень точные гомопурин-гомопиримидиновые инвертированные повторы, которые иначе называются H-палиндромами, обычное явление в тройных спиральных H-конформациях, которые могут включать либо TAT, либо CGC-нуклеотидные триады. Другие можно описать как длинные инвертированные повторы, имеющие тенденцию образовывать шпильки и крестообразные, и, наконец, прямые тандемные повторы, которые обычно существуют в структурах, описываемых как скользящая петля, крестообразная и левосторонняя Z-ДНК. [14]
Прошлые исследования показывают, что повторы являются общей чертой эукариот, в отличие от прокариот и архей . [14] Другие отчеты показывают, что независимо от сравнительной нехватки повторяющихся элементов в прокариотических геномах, они, тем не менее, содержат сотни или даже тысячи больших повторов. [15] Текущий геномный анализ, по-видимому, предполагает существование большого избытка совершенных инвертированных повторов во многих прокариотических геномах по сравнению с эукариотическими геномами. [16]

Для количественной оценки и сравнения инвертированных повторов между несколькими видами, а именно археями, см. [17]
Псевдоузлы — это распространенные структурные мотивы, обнаруженные в РНК. Они образованы двумя вложенными стебель-петлями, так что стебель одной структуры образован петлей другой. Среди псевдоузлов существует множество топологий складывания и большое разнообразие длин петель, что делает их структурно разнообразной группой. [18]
Инвертированные повторы являются ключевым компонентом псевдоузлов, как можно увидеть на иллюстрации естественного псевдоузла, обнаруженного в компоненте РНК человеческой теломеразы . [19] В эту структуру вовлечены четыре различных набора инвертированных повторов. Наборы 1 и 2 являются стеблем стебля-петли A и являются частью петли для стебля-петли B. Аналогично, наборы 3 и 4 являются стеблем для стебля-петли B и являются частью петли для стебля-петли A.
Псевдоузлы играют ряд различных ролей в биологии. Псевдоузел теломеразы на иллюстрации имеет решающее значение для активности этого фермента. [ 19] Рибозим вируса гепатита дельта (HDV) сворачивается в структуру с двойным псевдоузлом и самостоятельно расщепляет свой кольцевой геном, производя РНК длиной в один геном. Псевдоузлы также играют роль в запрограммированном сдвиге рамки рибосомы , обнаруженном в некоторых вирусах и необходимом для репликации ретровирусов . [18]
Инвертированные повторы играют важную роль в рибопереключателях , которые являются регуляторными элементами РНК, которые контролируют экспрессию генов, которые производят мРНК, частью которой они являются. [10] Упрощенный пример рибопереключателя флавинмононуклеотида (ФМН) показан на иллюстрации. Этот рибопереключатель существует в транскрипте мРНК и имеет несколько структур стебель-петля выше по течению от кодирующей области . Однако на иллюстрации показаны только ключевые стебель-петли, которые были значительно упрощены, чтобы помочь показать роль инвертированных повторов. В этом рибопереключателе есть несколько инвертированных повторов, как показано зеленым (желтый фон) и синим (оранжевый фон).
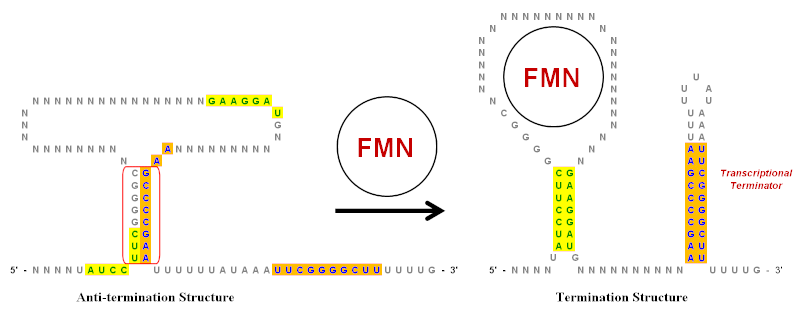
При отсутствии FMN структура Anti-termination является предпочтительной конформацией для транскрипта мРНК. Она создается путем спаривания оснований инвертированной области повтора, обведенной красным. Когда FMN присутствует, он может связываться с петлей и предотвращать образование структуры Anti-termination. Это позволяет двум различным наборам инвертированных повторов образовывать пары оснований и формировать структуру Termination. [20] Стебель-петля на 3'-конце является терминатором транскрипции , поскольку последовательность, непосредственно следующая за ней, представляет собой цепочку урацилов (U). Если эта стебель-петля образуется (из-за присутствия FMN) по мере того, как растущая цепь РНК выходит из комплекса РНК-полимеразы , она создаст достаточно структурного напряжения, чтобы вызвать диссоциацию цепи РНК и, таким образом, завершить транскрипцию. Диссоциация происходит легко, поскольку спаривание оснований между U в РНК и A в цепи шаблона является самым слабым из всех пар оснований. [10] Таким образом, при более высоких уровнях концентрации ФМН подавляет собственную транскрипцию, увеличивая образование терминальной структуры.
Инвертированные повторы часто описываются как «горячие точки» эукариотической и прокариотической геномной нестабильности. [7] Считается, что длинные инвертированные повторы оказывают большое влияние на стабильность генома различных организмов. [21] Это проиллюстрировано на примере E. coli , где геномные последовательности с длинными инвертированными повторами редко реплицируются, а скорее быстро удаляются. [21] Опять же, длинные инвертированные повторы, наблюдаемые у дрожжей, в значительной степени способствуют рекомбинации в пределах одной и той же и соседних хромосом, что приводит к одинаково высокой скорости делеции. [21] Наконец, очень высокая скорость делеции и рекомбинации также наблюдалась в областях хромосом млекопитающих с инвертированными повторами. [21] Сообщаемые различия в стабильности геномов взаимосвязанных организмов всегда являются признаком несоответствия в инвертированных повторах. [11] Нестабильность возникает из-за тенденции инвертированных повторов складываться в шпильковые или крестообразные структуры ДНК. Эти особые структуры могут препятствовать или запутывать репликацию ДНК и другие виды геномной активности. [7] Таким образом, инвертированные повторы приводят к особым конфигурациям как в РНК , так и в ДНК, которые в конечном итоге могут вызывать мутации и заболевания . [9]

На рисунке показан инвертированный повтор, подвергающийся крестообразной экструзии. ДНК в области инвертированного повтора раскручивается и затем рекомбинирует, образуя четырехстороннее соединение с двумя структурами стебель-петля . Крестообразная структура возникает из-за того, что инвертированные повторные последовательности самоспариваются друг с другом на своей собственной нити. [22]
Выдавленные крестообразные структуры могут приводить к мутациям со сдвигом рамки считывания , когда последовательность ДНК имеет инвертированные повторы в форме палиндрома, объединенные с областями прямых повторов с обеих сторон. Во время транскрипции проскальзывание и частичная диссоциация полимеразы от шаблонной нити могут приводить как к делеционным, так и к инсерционным мутациям. [9] Делеция происходит, когда часть раскрученной шаблонной нити образует стебель-петлю, которая «пропускается» транскрипционным аппаратом. Инсерция происходит, когда стебель-петля образуется в диссоциированной части зарождающейся (ново синтезированной) нити, заставляя часть шаблонной нити транскрибироваться дважды. [9]
Несовершенные инвертированные повторы могут приводить к мутациям через внутрицепочечное и межцепочечное переключение. [9] Кодирующая область гена антитромбина III является примером несовершенного инвертированного повтора, как показано на рисунке справа. Структура стебля-петли формируется с выступом внизу, поскольку G и T не образуют пары. Событие переключения нити может привести к замене G (в выступе) на A, что устраняет «несовершенство» в инвертированном повторе и обеспечивает более прочную структуру стебля-петли. Однако замена также создает точечную мутацию, преобразующую кодон GCA в ACA. Если за событием переключения нити следует второй раунд репликации ДНК , мутация может закрепиться в геноме и привести к заболеванию. В частности, миссенс-мутация приведет к дефектному гену и дефициту антитромбина, что может привести к развитию венозной тромбоэмболии (сгустки крови в вене). [9]
Мутации в гене коллагена могут привести к заболеванию несовершенный остеогенез , которое характеризуется хрупкостью костей. [9] На иллюстрации петля-стебель, образованная из несовершенного инвертированного повтора, мутирует с вставкой нуклеотида тимина (T) в результате меж- или внутрицепочечного переключения. Добавление T создает парное основание «совпадение» с аденином (A), который ранее был «шишкой» на левой стороне стебля. Хотя это добавление делает стебель более прочным и совершенствует инвертированный повтор, оно также создает мутацию сдвига рамки в последовательности нуклеотидов, которая изменяет рамку считывания и приведет к неправильной экспрессии гена. [9]
В следующем списке представлена информация и внешние ссылки на различные программы и базы данных для инвертированных повторов:
{{cite book}}: CS1 maint: несколько имен: список авторов ( ссылка )