PubMed — это бесплатная база данных , включающая в себя в первую очередь базу данных MEDLINE ссылок и рефератов по биологическим наукам и биомедицинским темам. Национальная медицинская библиотека США (NLM) в Национальном институте здравоохранения поддерживает базу данных как часть системы поиска информации Entrez . [1]
С 1971 по 1997 год онлайн-доступ к базе данных MEDLINE осуществлялся в основном через институциональные учреждения, такие как университетские библиотеки . [2] PubMed, впервые выпущенный в январе 1996 года, открыл эру частного, бесплатного, домашнего и офисного поиска MEDLINE. [3] Система PubMed стала предлагаться бесплатно для публики с июня 1997 года. [2]
Помимо MEDLINE, PubMed предоставляет доступ к:
Многие записи PubMed содержат ссылки на полные тексты статей, некоторые из которых находятся в свободном доступе, часто в PubMed Central [5] и локальных зеркалах, таких как Europe PubMed Central . [6]
Информация о журналах, индексированных в MEDLINE и доступных через PubMed, находится в каталоге NLM. [7]
По состоянию на 23 мая 2023 года [обновлять]в PubMed имеется более 35 миллионов ссылок и рефератов, датируемых с 1966 года, выборочно с 1865 года и очень выборочно с 1809 года. По состоянию на ту же дату [обновлять]24,6 миллиона записей PubMed перечислены с их рефератами, а 26,8 миллиона записей имеют ссылки на полнотекстовые версии (из которых 10,9 миллиона статей доступны в виде полных текстов бесплатно). [8] За последние 10 лет (по состоянию на 31 декабря 2019 года) в среднем ежегодно добавлялось около миллиона новых записей.
В 2016 году NLM изменила систему индексации, чтобы издатели могли напрямую исправлять опечатки и ошибки в статьях, проиндексированных PubMed. [9]
Сообщалось, что PubMed включает некоторые статьи, опубликованные в хищных журналах. Политики MEDLINE и PubMed по выбору журналов для включения в базу данных немного различаются. Слабости в критериях и процедурах индексации журналов в PubMed Central могут позволить публикациям из хищных журналов просочиться в PubMed. [10]
Новый интерфейс PubMed был запущен в октябре 2009 года и поощрял использование таких быстрых поисковых формулировок, как у Google; их также называют поиском «телеграмм». [11] По умолчанию результаты сортируются по «Самым последним», но это можно изменить на «Лучшее совпадение», «Дата публикации», «Первый автор», «Последний автор», «Журнал» или «Название». [12]
Дизайн и домен веб-сайта PubMed были обновлены в январе 2020 года и стали использоваться по умолчанию 15 мая 2020 года с обновленными и новыми функциями. [13] Была критическая реакция со стороны многих исследователей, которые часто используют сайт. [14]
Доступ к PubMed/MEDLINE можно получить с помощью портативных устройств, например, с помощью опции «PICO» (для специализированных клинических вопросов), созданной NLM. [15] Также доступна опция «PubMed Mobile», обеспечивающая доступ к мобильной, упрощенной версии PubMed. [16]
Простой поиск в PubMed можно выполнить, введя ключевые аспекты темы в окно поиска PubMed.
PubMed переводит эту первоначальную формулировку поиска и автоматически добавляет названия полей, соответствующие термины MeSH (Медицинские предметные рубрики), синонимы, логические операторы и «вкладывает» полученные термины соответствующим образом, значительно улучшая формулировку поиска, в частности, путем регулярного объединения (с использованием оператора OR) текстовых слов и терминов MeSH. [ необходима ссылка ]
Примеры, приведенные в руководстве PubMed [17], демонстрируют, как работает этот автоматический процесс:
Причины снохождения переводятся как («этиология»[Подзаголовок] ИЛИ «этиология»[Все поля] ИЛИ «причины»[Все поля] ИЛИ «причинность»[Термины MeSH] ИЛИ «причинность»[Все поля]) И («сомнамбулизм»[Термины MeSH] ИЛИ «сомнамбулизм»[Все поля] ИЛИ («сон»[Все поля] И «ходьба»[Все поля]) ИЛИ «хождение во сне»[Все поля])
Так же,
soft Attack Aspirin Prevention переводится как («инфаркт миокарда»[Термины MeSH] ИЛИ («миокардиальный»[Все поля] И «инфаркт»[Все поля]) ИЛИ «инфаркт миокарда»[Все поля] ИЛИ («сердце»[Все поля] И «атака»[Все поля]) ИЛИ «сердечный приступ»[Все поля]) И («аспирин»[Термины MeSH] ИЛИ «аспирин»[Все поля]) И («профилактика и контроль»[Подзаголовок] ИЛИ («профилактика»[Все поля] И «контроль»[Все поля]) ИЛИ «профилактика и контроль»[Все поля] ИЛИ «профилактика»[Все поля])
Для оптимального поиска в PubMed необходимо понимать его основной компонент, MEDLINE, и особенно контролируемый словарь MeSH (Medical Subject Headings), используемый для индексации статей MEDLINE. Они также могут потребовать сложных стратегий поиска, использования названий полей (тегов), правильного использования ограничений и других функций; библиотекари-справочники и специалисты по поиску предлагают услуги поиска. [18] [19]
Поиск в окне поиска PubMed рекомендуется только для поиска однозначных тем или новых вмешательств, для которых еще не создан заголовок MeSH, а также для поиска коммерческих марок лекарств и имен собственных. Он также полезен, когда нет подходящего заголовка или дескриптор представляет собой частичный аспект. Поиск с использованием тезауруса MeSH более точен и даст меньше нерелевантных результатов. Кроме того, он избавляет от недостатка поиска свободного текста, при котором необходимо учитывать орфографические, единственное/множественное число или сокращения. С другой стороны, статьи, недавно включенные в базу данных, которым еще не были назначены дескрипторы, не будут найдены. Поэтому, чтобы гарантировать исчерпывающий поиск, необходимо использовать комбинацию контролируемых языковых заголовков и свободных текстовых терминов. [20]
При индексации журнальной статьи извлекаются многочисленные параметры статьи и сохраняются в виде структурированной информации. К таким параметрам относятся: Тип статьи (термины MeSH, например, «Клиническое исследование»), Вторичные идентификаторы (термины MeSH), Язык, Страна журнала или история публикации (дата электронной публикации, дата публикации печатного журнала).
Параметр типа публикации позволяет осуществлять поиск по типу публикации , включая отчеты о различных видах клинических исследований. [21]
С июля 2005 года процесс индексации статей MEDLINE извлекает идентификаторы из аннотации статьи и помещает их в поле, называемое Вторичный идентификатор (SI). Поле вторичного идентификатора предназначено для хранения номеров доступа к различным базам данных молекулярных последовательностей, экспрессии генов или химических соединений и идентификаторов клинических испытаний. Для клинических испытаний PubMed извлекает идентификаторы испытаний из двух крупнейших реестров испытаний: ClinicalTrials.gov (идентификатор NCT) и Международный стандартный регистр рандомизированных контролируемых испытаний (идентификатор IRCTN). [22]
Ссылка, которая считается особенно релевантной, может быть отмечена, и могут быть идентифицированы «связанные статьи». Если релевантно, можно выбрать несколько исследований и сгенерировать связанные статьи для всех них (в PubMed или любой другой базе данных NCBI Entrez) с помощью опции «Найти связанные данные». Затем связанные статьи перечисляются в порядке «связанности». Чтобы создать эти списки связанных статей, PubMed сравнивает слова из заголовка и аннотации каждой цитаты, а также назначенные заголовки MeSH, используя мощный алгоритм взвешивания слов. [23] Функция «связанные статьи» была признана настолько точной, что авторы статьи предложили использовать ее вместо полного поиска. [24]
PubMed автоматически ссылается на термины и подзаголовки MeSH. Примерами могут служить: «плохой запах изо рта» ссылается на (и включает в поиск) «галитоз», «сердечный приступ» на «инфаркт миокарда», «рак груди» на «новообразования груди». Где это уместно, эти термины MeSH автоматически «расширяются», то есть включают более конкретные термины. Такие термины, как «уход» автоматически связываются с «Уход [MeSH]» или «Уход [Подзаголовок]». Эта функция называется «автоматическое сопоставление терминов» и по умолчанию применяется при свободном текстовом поиске, но не при точном поиске фраз (т. е. при заключении поискового запроса в двойные кавычки). [25] Эта функция делает поиск PubMed более чувствительным и позволяет избежать ложноотрицательных (пропущенных) результатов, компенсируя разнообразие медицинской терминологии. [25]
PubMed не применяет автоматическое сопоставление термина в следующих случаях: при написании фразы в кавычках (например, «почечный аллотрансплантат»), при усечении на звездочке (например, почечный аллотрансплантат*) и при поиске с метками полей (например, Рак [ti]). [20]
Дополнительная функция PubMed «Мой NCBI» (с бесплатной регистрацией) предоставляет инструменты для
и широкий спектр других опций. [26] Доступ к разделу "My NCBI" возможен с любого компьютера с доступом в Интернет. Более ранняя версия "My NCBI" называлась "PubMed Cubby". [27]
LinkOut — это средство NLM для связывания и предоставления доступа к полным текстам местных журнальных фондов. [28] Около 3200 сайтов (в основном академические учреждения) участвуют в этом средстве NLM (по состоянию на март 2010 г. [обновлять]), от Университета Ольборга в Дании до ZymoGenetics в Сиэтле. [29] Пользователи этих учреждений видят логотип своего учреждения в результатах поиска PubMed (если журнал хранится в этом учреждении) и могут получить доступ к полному тексту. Link out объединяется с Outside Tool с момента крупного обновления платформы, которое состоится летом 2019 г. [30]
В 2016 году PubMed позволяет авторам статей комментировать статьи, индексируемые PubMed. Эта функция изначально тестировалась в пилотном режиме (с 2013 года) и стала постоянной в 2016 году. [31] В феврале 2018 года PubMed Commons был закрыт из-за того, что «использование оставалось минимальным». [32] [33]
askMEDLINE, инструмент для создания запросов на естественном языке в свободном тексте для MEDLINE/PubMed, разработанный NLM, также подходит для портативных устройств. [34]
PMID (идентификатор PubMed или уникальный идентификатор PubMed) [35] — это уникальное целочисленное значение , начинающееся с , назначаемое каждой записи PubMed. PMID — это не то же самое, что PMCID ( идентификатор PubMed Central), который является идентификатором для всех работ, опубликованных в свободном доступе PubMed Central . [36]1
Присвоение PMID или PMCID публикации ничего не говорит читателю о типе или качестве контента. PMID присваиваются письмам редактору , редакционным мнениям, колонкам и любым другим материалам, которые редактор решает включить в журнал, а также рецензируемым статьям. Наличие идентификационного номера также не является доказательством того, что статьи не были отозваны из -за мошенничества, некомпетентности или неправомерного поведения. Объявлению о любых исправлениях в исходных статьях может быть присвоен PMID.
Каждое число, введенное в окно поиска PubMed, по умолчанию рассматривается как PMID. Таким образом, любая ссылка в PubMed может быть найдена с помощью PMID.
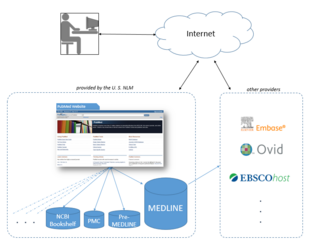
Национальная медицинская библиотека сдает в аренду информацию MEDLINE ряду частных поставщиков, таких как Embase , Ovid , Dialog , EBSCO , Knowledge Finder и многим другим коммерческим, некоммерческим и академическим поставщикам. [37] По состоянию на октябрь 2008 года [обновлять]было выдано более 500 лицензий, более 200 из них — поставщикам за пределами США. Поскольку лицензии на использование данных MEDLINE предоставляются бесплатно, NLM фактически предоставляет бесплатную испытательную площадку для широкого спектра [38] альтернативных интерфейсов и сторонних дополнений к PubMed, одной из очень немногих крупных, профессионально курируемых баз данных, которая предлагает такую возможность.
Лу определяет выборку из 28 текущих и бесплатных веб-версий PubMed, не требующих установки или регистрации, которые сгруппированы в четыре категории: [38]
Поскольку большинство из этих и других альтернатив в основном полагаются на данные PubMed/MEDLINE, взятые в аренду по лицензии у NLM/PubMed, был предложен термин «производные PubMed». [38] Без необходимости хранить около 90 ГБ исходных наборов данных PubMed, любой может писать приложения PubMed, используя программный интерфейс eutils-application, как описано в «The E-utilities In-Depth: Parameters, Syntax and More» Эрика Сэйерса, доктора философии. [49] Различные генераторы форматов цитирования, принимающие номера PMID в качестве входных данных, являются примерами веб-приложений, использующих программный интерфейс eutils-application. Примеры веб-страниц включают Citation Generator – Mick Schroeder, Pubmed Citation Generator – Ultrasound of the Week, PMID2cite и Cite this for me.
Альтернативные методы добычи данных в PubMed используют среды программирования, такие как Matlab , Python или R. В этих случаях запросы PubMed пишутся как строки кода и передаются в PubMed, а затем ответ обрабатывается непосредственно в среде программирования. Код может быть автоматизирован для систематического запроса с различными ключевыми словами, такими как болезнь, год, органы и т. д.
В дополнение к своей традиционной роли биомедицинской базы данных, PubMed стал общим ресурсом для обучения моделей биомедицинского языка . [50] Недавние достижения в этой области включают разработку таких моделей, как PubMedGPT, модель с 2,7 млрд параметров, обученная на данных PubMed Стэнфордским CRFM, и BiomedCLIP-PubMedBERT от Microsoft, которая использует пары рисунок-подпись из PubMed Central для обработки зрения-языка. Эти модели демонстрируют значительный потенциал данных PubMed в расширении возможностей ИИ в медицинских исследованиях и приложениях здравоохранения. Такие достижения подчеркивают растущее пересечение между крупномасштабным интеллектуальным анализом данных и разработкой ИИ в области биомедицины.
Данные, доступные через PubMed, можно отразить локально с помощью неофициального инструмента, такого как MEDOC. [51]
Миллионы записей PubMed дополняют различные открытые наборы данных об открытом доступе , такие как Unpaywall . Инструменты анализа данных, такие как Unpaywall Journals, используются библиотеками для помощи в отмене крупных сделок : библиотеки могут избежать подписки на материалы, уже обслуживаемые мгновенным открытым доступом через открытые архивы, такие как PubMed Central. [52]
[[Категория:Национальная медицинская библиотека США|PubMed]