
SOS -ответ — это глобальный ответ на повреждение ДНК , при котором клеточный цикл останавливается, а репарация ДНК и мутагенез индуцируются. Система включает белок RecA ( Rad51 у эукариот). Белок RecA, стимулируемый одноцепочечной ДНК, участвует в инактивации репрессора ( LexA ) генов SOS-ответа, тем самым индуцируя ответ. Это система репарации, подверженная ошибкам, которая вносит значительный вклад в изменения ДНК, наблюдаемые у широкого спектра видов.
SOS-ответ был сформулирован Эвелин Виткин . [3] [4] Позднее, характеризуя фенотипы мутировавших E. coli , она и аспирант Мирослав Радман подробно описали SOS-ответ на УФ-излучение у бактерий. [3] [5] SOS-ответ на повреждение ДНК стал основополагающим открытием, поскольку это был первый скоординированный ответ на стресс, который был объяснен. [6]
Во время нормального роста гены SOS отрицательно регулируются димерами белка -репрессора LexA . В нормальных условиях LexA связывается с консенсусной последовательностью из 20 пар оснований ( бокс SOS ) в области оператора для этих генов. Некоторые из этих генов SOS экспрессируются на определенных уровнях даже в репрессированном состоянии, в соответствии с сродством LexA к их боксу SOS. Активация генов SOS происходит после повреждения ДНК путем накопления одноцепочечных (ssDNA) областей, образующихся в репликационных вилках, где ДНК-полимераза блокируется. RecA образует нить вокруг этих областей ssDNA в зависимости от АТФ и активируется. [7] Активированная форма RecA взаимодействует с репрессором LexA, способствуя самоотщеплению репрессора LexA от оператора. [7] [8]
Как только пул LexA уменьшается, репрессия генов SOS снижается в соответствии с уровнем сродства LexA к SOS-боксам. [7] Операторы, которые слабо связывают LexA, первыми полностью экспрессируются. Таким образом, LexA может последовательно активировать различные механизмы репарации. Гены, имеющие слабый SOS-бокс (такие как lexA , recA , uvrA , uvrB и uvrD ), полностью индуцируются в ответ даже на слабые SOS-индуцирующие обработки. Таким образом, первым индуцируемым механизмом репарации SOS является нуклеотидная эксцизионная репарация (NER), целью которой является исправление повреждений ДНК без приверженности полноценному SOS-ответу. Если, однако, NER недостаточно для исправления повреждений, концентрация LexA еще больше снижается, поэтому индуцируется экспрессия генов с более сильными LexA-боксами (такими как sulA , umuD , umuC — они экспрессируются поздно). [7] SulA останавливает деление клеток [7] , связываясь с FtsZ , инициирующим белком в этом процессе. Это вызывает филаментацию и индукцию UmuDC-зависимой мутагенной репарации. В результате этих свойств некоторые гены могут частично индуцироваться в ответ даже на эндогенные уровни повреждения ДНК, в то время как другие гены, по-видимому, индуцируются только при наличии в клетке высокого или постоянного повреждения ДНК.
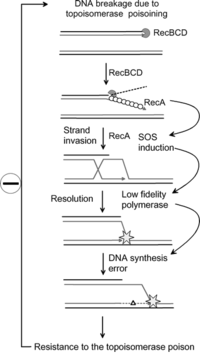
Исследования показали, что система ответа SOS может приводить к мутациям, которые могут привести к устойчивости к антибиотикам. [9] Повышенная скорость мутаций во время ответа SOS вызвана тремя низкоточными ДНК-полимеразами : Pol II , Pol IV и Pol V. [ 10] [9] Исследователи теперь нацеливаются на эти белки с целью создания лекарств, которые предотвращают восстановление SOS. Таким образом, время, необходимое патогенным бактериям для развития устойчивости к антибиотикам, может быть увеличено, тем самым улучшая долгосрочную жизнеспособность некоторых антибиотиков. [11]
Помимо генетической устойчивости, SOS-ответ может также способствовать фенотипической устойчивости. Здесь геном сохраняется, в то время как другие негенетические факторы изменяются, чтобы позволить бактериям выжить. Например, зависимая от SOS система токсина-антитоксина tisB-istR была связана с индукцией персистирующих клеток , зависящих от повреждения ДНК . [12]

В Escherichia coli различные классы агентов, повреждающих ДНК, могут инициировать реакцию SOS, как описано выше. Используя преимущество слияния оперонов, помещая оперон lac (ответственный за выработку бета-галактозидазы, белка, который расщепляет лактозу) под контроль белка, связанного с SOS, можно провести простой колориметрический анализ генотоксичности . К бактериям добавляется аналог лактозы, который затем расщепляется бета-галактозидазой, в результате чего образуется окрашенное соединение, которое можно количественно измерить с помощью спектрофотометрии . Степень развития цвета является косвенной мерой произведенной бета-галактозидазы, которая сама по себе напрямую связана с количеством повреждений ДНК.
E. coli далее модифицируются для того, чтобы иметь ряд мутаций, включая мутацию uvrA , которая делает штамм дефицитным по эксцизионной репарации, увеличивая реакцию на определенные агенты, повреждающие ДНК, а также мутацию rfa , которая делает бактерии дефицитными по липополисахариду, что позволяет улучшить диффузию определенных химических веществ в клетку для того, чтобы вызвать реакцию SOS. [13] Доступны коммерческие наборы, которые измеряют первичную реакцию клетки E. coli на генетическое повреждение и могут быть тесно связаны с тестом Эймса для определенных материалов. [14]
Цианобактерии , единственные прокариоты, способные к фотосинтезу с выделением кислорода , являются основными производителями кислородсодержащей атмосферы Земли. [15] Морские цианобактерии Prochlorococcus и Synechococcus, по-видимому, имеют систему SOS, похожую на E. coli, для восстановления ДНК, поскольку они кодируют гены, гомологичные ключевым генам SOS E. coli, таким как lexA и sulA . [16]