Просвечивающая электронная микроскопия Секвенирование ДНК — это технология секвенирования отдельных молекул , которая использует методы просвечивающей электронной микроскопии . Метод был задуман и разработан в 1960-х и 70-х годах, [1] но утратил популярность, когда была признана степень повреждения образца. [2]
Для того, чтобы ДНК была четко визуализирована под электронным микроскопом , она должна быть помечена тяжелыми атомами. Кроме того, специализированные методы визуализации и оптика с коррекцией аберраций полезны для получения разрешения, необходимого для изображения меченой молекулы ДНК. Теоретически, секвенирование ДНК с помощью просвечивающей электронной микроскопии может обеспечить чрезвычайно большую длину считывания, но проблема повреждения электронным пучком все еще может оставаться, и эта технология еще не получила коммерческого развития.
Всего через несколько лет после того, как Джеймс Уотсон и Фрэнсис Крик вывели структуру ДНК , и почти за два десятилетия до того, как Фредерик Сэнгер опубликовал первый метод быстрого секвенирования ДНК , Ричард Фейнман , американский физик, представил электронный микроскоп как инструмент, который однажды позволит биологам «увидеть порядок оснований в цепочке ДНК ». [3] Фейнман считал, что если бы электронный микроскоп можно было сделать достаточно мощным, то стало бы возможным визуализировать атомную структуру любых химических соединений, включая ДНК.
В 1970 году Альберт Крю разработал метод визуализации высокоуглового кольцевого темного поля (HAADF) в сканирующем просвечивающем электронном микроскопе . Используя этот метод, он визуализировал отдельные тяжелые атомы на тонких аморфных углеродных пленках. [4] В 2010 году Криванек и его коллеги сообщили о нескольких технических усовершенствованиях метода HAADF, включая комбинацию электронной оптики с коррекцией аберраций и низкого ускоряющего напряжения. Последнее имеет решающее значение для визуализации биологических объектов, поскольку позволяет уменьшить повреждение пучком и увеличить контрастность изображения для легких атомов. В результате можно было визуализировать замещения отдельных атомов в монослое нитрида бора. [5]
Несмотря на изобретение множества химических и флуоресцентных технологий секвенирования, электронная микроскопия все еще изучается как средство выполнения секвенирования одиночной молекулы ДНК. Например, в 2012 году сотрудничество между учеными Гарвардского университета , Университета Нью-Гемпшира и ZS Genetics продемонстрировало возможность считывать длинные последовательности ДНК с помощью этой техники, [6] однако технология секвенирования ДНК с помощью трансмиссионной электронной микроскопии все еще далека от коммерческой доступности. [7]
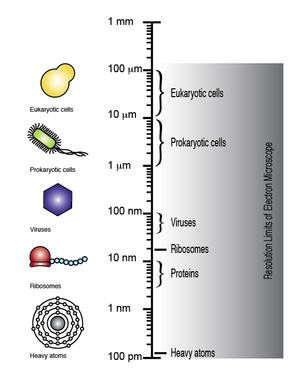
Электронный микроскоп способен достигать разрешения до 100 мкм, благодаря чему можно наблюдать микроскопические биомолекулы и структуры, такие как вирусы, рибосомы, белки, липиды, малые молекулы и даже отдельные атомы. [8]
Хотя ДНК видна при наблюдении с помощью электронного микроскопа, разрешение полученного изображения недостаточно высоко, чтобы позволить расшифровать последовательность отдельных оснований , т. е . секвенирование ДНК . Однако при дифференциальной маркировке оснований ДНК тяжелыми атомами или металлами можно как визуализировать, так и различать отдельные основания. Поэтому электронная микроскопия в сочетании с дифференциальной маркировкой ДНК тяжелыми атомами может быть использована для прямого получения изображения ДНК с целью определения ее последовательности. [7] [9] [10] [11]
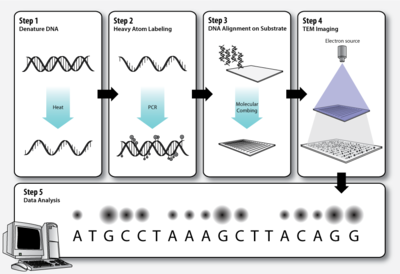
Как и в стандартной полимеразной цепной реакции (ПЦР) , двухцепочечные молекулы ДНК, подлежащие секвенированию, должны быть денатурированы , прежде чем вторая цепь может быть синтезирована с использованием меченых нуклеотидов.
Элементы, из которых состоят биологические молекулы ( C , H , N , O , P , S ), слишком легкие (низкий атомный номер, Z ), чтобы их можно было четко визуализировать как отдельные атомы с помощью просвечивающей электронной микроскопии . Чтобы обойти эту проблему, основания ДНК можно пометить более тяжелыми атомами (более высокий Z). Каждый нуклеотид помечен характерной тяжелой меткой, чтобы их можно было различить на просвечивающей электронной микрофотографии.
Молекулы ДНК должны быть растянуты на тонком твердом субстрате, чтобы порядок меченых оснований был четко виден на электронной микрофотографии. Молекулярное расчесывание — это метод, который использует силу отступающего интерфейса воздух-вода для удлинения молекул ДНК, оставляя их необратимо связанными со слоем силана после высыхания. [13] [14] Это один из способов, с помощью которого можно добиться выравнивания ДНК на твердом субстрате.

Просвечивающая электронная микроскопия (ПЭМ) создает изображения с высоким увеличением и высоким разрешением , пропуская пучок электронов через очень тонкий образец. В то время как атомное разрешение было продемонстрировано с помощью обычного ПЭМ, дальнейшее улучшение пространственного разрешения требует исправления сферических и хроматических аберраций линз микроскопа . Это было возможно только в сканирующей просвечивающей электронной микроскопии , где изображение получается путем сканирования объекта тонко сфокусированным электронным пучком, аналогично электронно-лучевой трубке . Однако достигнутое улучшение разрешения сопровождается облучением изучаемого объекта гораздо более высокими интенсивностями пучка, сопутствующим повреждением образца и связанными с этим артефактами изображения. [15] Различные методы визуализации применяются в зависимости от того, содержит ли образец тяжелые или легкие атомы:
Темные и светлые пятна на электронной микрофотографии, соответствующие дифференциально маркированным основаниям ДНК, анализируются с помощью компьютерной программы.
Секвенирование ДНК с помощью трансмиссионной электронной микроскопии пока недоступно для коммерческого использования, но большие длины считывания, которые эта технология может обеспечить в будущем, сделают ее полезной в самых разных контекстах.
При секвенировании генома его необходимо разбить на части, которые достаточно короткие, чтобы их можно было секвенировать за одно считывание. Затем эти считывания необходимо собрать обратно, как пазл, выровняв области, которые перекрываются между считываниями; этот процесс называется сборкой генома de novo . Чем больше длина считывания, которую обеспечивает платформа секвенирования, тем длиннее перекрывающиеся области и тем легче собрать геном. С вычислительной точки зрения микрофлюидное секвенирование по Сэнгеру по-прежнему является наиболее эффективным способом секвенирования и сборки геномов, для которых не существует референтной последовательности генома . Относительно большая длина считываний обеспечивает существенное перекрытие между отдельными считываниями секвенирования, что обеспечивает большую статистическую достоверность сборки. Кроме того, длинные считывания по Сэнгеру способны охватывать большинство областей повторяющейся последовательности ДНК , которые в противном случае затрудняют сборку последовательности, вызывая ложные выравнивания. Однако сборка генома de novo с помощью секвенирования по Сэнгеру чрезвычайно дорога и требует много времени. Технологии секвенирования второго поколения [19] , хотя и менее дорогие, как правило, не подходят для сборки генома de novo из-за короткой длины прочтения. В целом, технологии секвенирования третьего поколения [11] , включая секвенирование ДНК с помощью просвечивающей электронной микроскопии, направлены на улучшение длины прочтения при сохранении низкой стоимости секвенирования. Таким образом, по мере совершенствования технологий секвенирования третьего поколения быстрая и недорогая сборка генома de novo станет реальностью.
Гаплотип — это ряд связанных аллелей , которые наследуются вместе на одной хромосоме. Секвенирование ДНК может быть использовано для генотипирования всех однонуклеотидных полиморфизмов (SNP), которые составляют гаплотип. Однако короткие прочтения ДНК-секвенирования часто не могут быть фазированы; то есть гетерозиготные варианты не могут быть уверенно отнесены к правильному гаплотипу. Фактически, гаплотипирование с данными секвенирования ДНК с короткими прочтениями требует очень высокого покрытия (в среднем >50x покрытия каждого основания ДНК) для точной идентификации SNP, а также дополнительных данных о последовательностях от родителей, чтобы можно было использовать менделевскую передачу для оценки гаплотипов. [20] Технологии секвенирования, которые генерируют длинные прочтения, включая секвенирование ДНК с помощью трансмиссионной электронной микроскопии, могут захватывать целые гаплоблоки за одно прочтение. То есть гаплотипы не разбиваются между несколькими прочтениями, и генетически связанные аллели остаются вместе в данных секвенирования. Таким образом, длинные считывания делают гаплотипирование более простым и точным, что приносит пользу области популяционной генетики .
Гены обычно присутствуют в двух копиях в диплоидном геноме человека; гены, которые отклоняются от этого стандартного числа копий, называются вариантами числа копий (CNV) . Изменение числа копий может быть доброкачественным (это обычно распространенные варианты, называемые полиморфизмами числа копий) или патогенным. [21] CNV обнаруживаются с помощью флуоресцентной гибридизации in situ (FISH) или сравнительной геномной гибридизации (CGH) . Чтобы обнаружить конкретные точки разрыва, в которых происходит делеция, или обнаружить геномные поражения, внесенные событием дупликации или амплификации, CGH можно выполнить с помощью тайлингового массива ( array CGH ), или можно секвенировать вариантную область. Длинные чтения секвенирования особенно полезны для анализа дупликаций или амплификации, поскольку можно проанализировать ориентацию амплифицированных сегментов, если они захвачены в одном чтении секвенирования.
Геномика рака, или онкогеномика , является новой областью, в которой высокопроизводительная технология секвенирования ДНК второго поколения применяется для секвенирования целых раковых геномов. Анализ этих данных секвенирования коротких прочтений охватывает все проблемы, связанные со сборкой генома de novo с использованием данных коротких прочтений. [22] Кроме того, раковые геномы часто являются анеуплоидными . [23] Эти аберрации, которые по сути являются крупномасштабными вариантами числа копий, могут быть проанализированы технологиями секвенирования второго поколения с использованием частоты прочтений для оценки числа копий. [22] Однако более длинные прочтения дадут более точную картину числа копий, ориентации амплифицированных областей и SNP, присутствующих в раковых геномах.
Микробиом относится к общей коллекции микробов , присутствующих в микросреде, и их соответствующим геномам. Например, по оценкам, 100 триллионов микробных клеток колонизируют человеческое тело в любой момент времени. [24] Человеческий микробиом представляет особый интерес, поскольку эти комменсальные бактерии важны для здоровья и иммунитета человека. Большинство бактериальных геномов Земли еще не были секвенированы; осуществление проекта по секвенированию микробиома потребовало бы обширной сборки генома de novo , перспектива, которая пугает с технологиями секвенирования ДНК с коротким считыванием. [25] Более длинные считывания значительно облегчили бы сборку новых микробных геномов.
По сравнению с другими технологиями секвенирования ДНК второго и третьего поколения, секвенирование ДНК с помощью просвечивающей электронной микроскопии имеет ряд потенциальных ключевых преимуществ и недостатков, которые в конечном итоге определят его полезность и значимость как будущей технологии секвенирования ДНК.
Многие технологии секвенирования ДНК второго и третьего поколения, не относящиеся к методу Сэнгера, были разработаны или в настоящее время разрабатываются с общей целью повышения производительности и снижения затрат, чтобы можно было в полной мере реализовать персонализированную генетическую медицину.
Премия Archon X Prize for Genomics в размере 10 миллионов долларов США, финансируемая фондом X Prize Foundation (Санта-Моника, Калифорния, США), а также гранты в размере 70 миллионов долларов США, финансируемые Национальным институтом исследований генома человека Национальных институтов здравоохранения (NIH-NHGRI), способствуют быстрому росту исследовательской активности в области разработки новых технологий секвенирования ДНК. [7]
Поскольку различные подходы, методы и стратегии определяют каждую технологию секвенирования ДНК, каждая из них имеет свои сильные и слабые стороны. Сравнение важных параметров между различными технологиями секвенирования ДНК второго и третьего поколения представлено в Таблице 1.