
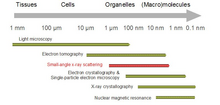
Биологическое малоугловое рассеяние — это метод малоуглового рассеяния для структурного анализа биологических материалов. Малоугловое рассеяние используется для изучения структуры различных объектов, таких как растворы биологических макромолекул, нанокомпозитов, сплавов и синтетических полимеров. [1] Малоугловое рентгеновское рассеяние ( SAXS ) и малоугловое нейтронное рассеяние ( SANS ) — это два взаимодополняющих метода, известных совместно как малоугловое рассеяние (SAS). [2] SAS — это аналогичный метод рентгеновской и нейтронной дифракции , широкоуглового рентгеновского рассеяния , а также статического рассеяния света . В отличие от других методов рентгеновского и нейтронного рассеяния, SAS дает информацию о размерах и формах как кристаллических, так и некристаллических частиц. При использовании для изучения биологических материалов, которые очень часто находятся в водном растворе, картина рассеяния усредняется по ориентации. [3] [4]
SAS-паттерны собираются под малыми углами в несколько градусов. SAS способен предоставлять структурную информацию в диапазоне разрешения от 1 до 25 нм и повторяющихся расстояний в частично упорядоченных системах размером до 150 нм. Ультрамалые углы рассеяния (USAS) позволяют разрешать даже большие размеры. Малые углы рассеяния скользящего падения (GISAS) являются мощным методом изучения слоев биологических молекул на поверхностях.
В биологических приложениях SAS используется для определения структуры частицы с точки зрения среднего размера и формы частицы. Также можно получить информацию об отношении поверхности к объему . Обычно биологические макромолекулы диспергированы в жидкости. Метод является точным, в основном неразрушающим и обычно требует лишь минимальной подготовки образца. Однако биологические молекулы всегда подвержены радиационному повреждению .
По сравнению с другими методами определения структуры, такими как ЯМР раствора или рентгеновская кристаллография , SAS позволяет преодолеть некоторые ограничения. Например, ЯМР раствора ограничен размером белка, тогда как SAS может использоваться как для малых молекул, так и для больших многомолекулярных ансамблей. Твердотельный ЯМР по-прежнему является незаменимым инструментом для определения информации на атомном уровне макромолекул более 40 кДа или некристаллических образцов, таких как амилоидные фибриллы. Определение структуры с помощью рентгеновской кристаллографии может занять несколько недель или даже лет, тогда как измерения SAS занимают дни. SAS также можно сочетать с другими аналитическими методами, такими как гель-хроматография, для изучения гетерогенных образцов. [5] Однако с помощью SAS невозможно измерить положение атомов внутри молекулы.

Концептуально эксперименты по малоугловому рассеянию просты: образец подвергается воздействию рентгеновских лучей или нейтронов , а рассеянное излучение регистрируется детектором. Поскольку измерения SAS проводятся очень близко к первичному пучку («малые углы»), для этой методики необходим высококоллимированный или сфокусированный рентгеновский или нейтронный пучок. Биологическое малоугловое рентгеновское рассеяние часто выполняется на источниках синхротронного излучения , поскольку биологические молекулы обычно рассеивают слабо, а измеряемые растворы являются разбавленными . Биологический метод SAXS использует высокую интенсивность пучков рентгеновских фотонов, обеспечиваемых синхротронными накопительными кольцами . Кривая рассеяния рентгеновских лучей или нейтронов (интенсивность в зависимости от угла рассеяния) используется для создания модели белка с низким разрешением, показанной здесь на правом рисунке. Можно также использовать данные рассеяния рентгеновских лучей или нейтронов и вписывать отдельные домены (структуры рентгеновских лучей или ЯМР ) в «оболочку SAXS».
В эксперименте по рассеянию раствор макромолекул подвергается воздействию рентгеновских лучей (с длиной волны λ обычно около 0,15 нм) или тепловых нейтронов ( λ ≈ 0,5 нм). Интенсивность рассеяния I(s) регистрируется как функция переданного импульса s ( s = 4πsinθ/λ , где 2θ — угол между падающим и рассеянным излучением). Из интенсивности раствора вычитается рассеяние только от растворителя. Случайные положения и ориентации частиц приводят к изотропному распределению интенсивности, которое для монодисперсных невзаимодействующих частиц пропорционально рассеянию от одной частицы, усредненному по всем ориентациям. Чистое рассеяние частиц пропорционально квадрату разности плотности длин рассеяния ( электронной плотности для рентгеновских лучей и ядерной/спиновой плотности для нейтронов) между частицей и растворителем — так называемый контраст. Контраст можно изменять при рассеянии нейтронов с использованием смесей H 2 O/ D 2 O или селективного дейтерирования для получения дополнительной информации. [1] Информационное содержание данных SAS проиллюстрировано здесь на рисунке справа, который показывает картины рассеяния рентгеновских лучей от белков с различными складками и молекулярными массами . При малых углах (разрешение 2-3 нм) кривые являются быстро затухающими функциями s , по существу определяемыми формой частиц, которые явно различаются. При среднем разрешении (от 2 до 0,5 нм) различия уже менее выражены, а выше разрешения 0,5 нм все кривые очень похожи. [6] Таким образом, SAS содержит информацию о грубых структурных особенностях – форме, четвертичной и третичной структуре – но не подходит для анализа атомной структуры.
Первые применения относятся к концу 1930-х годов, когда основные принципы SAXS были разработаны в фундаментальной работе Гинье после его исследований металлических сплавов. В первой монографии по SAXS Гинье и Фурне уже было показано, что метод дает не только информацию о размерах и форме частиц, но и о внутренней структуре неупорядоченных и частично упорядоченных систем.
В 1960-х годах этот метод стал приобретать все большее значение в изучении биологических макромолекул в растворе, поскольку он позволял получать структурную информацию с низким разрешением об общей форме и внутренней структуре в отсутствие кристаллов. Прорыв в экспериментах SAXS и SANS произошел в 1970-х годах благодаря доступности синхротронного излучения и источников нейтронов, которые проложили путь к изменению контраста путем замены растворителя H 2 O на D 2 O и специфическим методам дейтерирования. Было осознано, что исследования рассеяния в растворе дают, при минимальных затратах времени и усилий, полезные сведения о структуре некристаллических биохимических систем. Более того, SAXS/SANS также сделали возможными исследования межмолекулярных взаимодействий в реальном времени, включая сборку и крупномасштабные конформационные изменения в макромолекулярных сборках .
Основная сложность SAS как структурного метода заключается в извлечении трехмерной структурной информации объекта из одномерных экспериментальных данных. В прошлом только общие параметры частиц (например, объем, радиус инерции) макромолекул определялись непосредственно из экспериментальных данных, тогда как анализ в терминах трехмерных моделей ограничивался простыми геометрическими телами (например, эллипсоидами, цилиндрами и т. д.) или выполнялся на основе проб и ошибок ad hoc. Электронная микроскопия часто использовалась в качестве ограничения при построении консенсусных моделей. В 1980-х годах прогресс в других структурных методах привел к снижению интереса биохимиков к исследованиям SAS, которые делали структурные выводы всего из нескольких общих параметров или основывались на моделях проб и ошибок.
1990-е годы принесли прорыв в методах анализа данных SAXS/SANS, что открыло путь для надежного моделирования макромолекулярных комплексов ab initio , включая детальное определение формы и структуры доменов и применение методов уточнения жесткого тела. Этот прогресс сопровождался дальнейшими достижениями в области инструментального обеспечения, что позволило достичь временных разрешений в пределах мс на источниках SR третьего поколения в исследованиях сворачивания белков и нуклеиновых кислот. [1]
В 2005 году был начат четырехлетний проект S - Angle X -Ray scattering Initative for E u R ope (SAXIER) с целью объединить методы SAXS с другими аналитическими методами и создать автоматизированное программное обеспечение для быстрого анализа больших объемов данных. Проект создал единую европейскую инфраструктуру SAXS, используя самые передовые доступные методы. [7]
В эксперименте SAS хорошего качества измеряются несколько растворов с различными концентрациями исследуемой макромолекулы. Экстраполируя кривые рассеяния, измеренные при различных концентрациях, до нулевой концентрации, можно получить кривую рассеяния, которая представляет собой бесконечное разбавление. Тогда эффекты концентрации не должны влиять на кривую рассеяния. Анализ данных экстраполированной кривой рассеяния начинается с проверки начала кривой рассеяния в области около s = 0. Если область следует приближению Гинье (также известному как закон Гинье ), образец не агрегируется . Тогда форму рассматриваемой частицы можно определить различными методами, некоторые из которых описаны в следующей ссылке. [1]
Первым шагом обычно является вычисление преобразования Фурье кривой рассеяния. Преобразованная кривая может быть интерпретирована как функция распределения расстояний внутри частицы. Это преобразование также дает преимущество регуляризации входных данных. [ необходима цитата ]
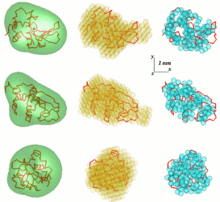
Одной из проблем анализа данных SAS является получение трехмерной структуры из одномерной картины рассеяния. Данные SAS не подразумевают единого решения. Например, многие различные белки могут иметь одну и ту же кривую рассеяния. Реконструкция трехмерной структуры может привести к большому количеству различных моделей. Чтобы избежать этой проблемы, необходимо рассмотреть ряд упрощений.
Дополнительный подход заключается в объединении данных и модели малоуглового рентгеновского и нейтронного рассеяния с программой MONSA.
Свободно доступные компьютерные программы анализа SAS интенсивно разрабатывались в EMBL . В первом общем подходе ab initio угловая огибающая функция частицы r=F(ω) , где ( r,ω ) — сферические координаты, описывается серией сферических гармоник . Таким образом, форма с низким разрешением определяется несколькими параметрами — коэффициентами этой серии, — которые соответствуют данным рассеяния. Подход был далее развит и реализован в компьютерной программе SASHA ( Small Angle Scattering Shape Determination ). [8] [9] Было показано, что при определенных обстоятельствах из данных рассеяния можно извлечь уникальную огибающую. Этот метод применим только к шаровидным частицам с относительно простой формой и без значительных внутренних полостей. Чтобы преодолеть эти ограничения, был разработан другой подход, который использует различные типы поиска Монте-Карло. DALAI_GA — элегантная программа, которая берет сферу с диаметром, равным максимальному размеру частицы Dmax, который определяется из данных рассеяния, и заполняет ее шариками. Каждая бусина принадлежит либо частице (индекс = 1), либо растворителю (индекс = 0). Таким образом, форма описывается двоичной строкой длины M. Начиная со случайной строки, генетический алгоритм ищет модель, которая соответствует данным. Ограничения компактности и связности налагаются при поиске, реализованном в программе DAMMIN . [10] [11] Если симметрия частицы известна, SASHA и DAMMIN могут использовать ее в качестве полезных ограничений. Процедура «отдай и возьми» SAXS3D и программа SASMODEL , основанные на взаимосвязанных эллипсоидах, являются подходами Монте-Карло ab initio без ограничений в пространстве поиска. [6]
Подход, который использует ансамбль фиктивных остатков (DR) и имитацию отжига для построения локально «совместимой с цепью» DR-модели внутри сферы диаметром Dmax, позволяет извлечь больше деталей из данных SAXS. Этот метод реализован в программе GASBOR . [12] [13]
Картины рассеяния раствора многодоменных белков и макромолекулярных комплексов также могут быть подогнаны с использованием моделей, построенных на основе структур высокого разрешения ( ЯМР или рентгеновских лучей ) отдельных доменов или субъединиц, предполагая, что их третичная структура сохранена. В зависимости от сложности объекта, применяются различные подходы для глобального поиска оптимальной конфигурации субъединиц, подходящей под экспериментальные данные.
Модели, основанные на Монте-Карло, содержат сотни или тысячи параметров, и требуется осторожность, чтобы избежать чрезмерной интерпретации. Обычный подход заключается в выравнивании набора моделей, полученных в результате независимых запусков реконструкции формы, для получения средней модели, сохраняющей наиболее устойчивые и, возможно, также наиболее надежные особенности (например, с помощью программы SUPCOMB ). [6] [14] [15]
Неупорядоченные поверхностные аминокислоты (« петли ») часто не наблюдаются в ЯМР и кристаллографических исследованиях и могут быть пропущены в представленных моделях. Такие неупорядоченные элементы вносят вклад в интенсивность рассеяния, и их вероятное местоположение может быть найдено путем фиксации известной части структуры и добавления недостающих частей для соответствия шаблону SAS от всей частицы. Подход фиктивных остатков был расширен, и алгоритмы для добавления недостающих петель или доменов были реализованы в программном пакете CREDO . [6]
Недавно было предложено несколько методов, которые используют данные SAXS в качестве ограничений. Авторы стремились улучшить результаты методов распознавания складок [16] и предсказания структуры белка de novo [17] . Данные SAXS предоставляют преобразование Фурье гистограммы расстояний между атомными парами (функция распределения пар) для данного белка. Это может служить структурным ограничением для методов, используемых для определения нативной конформационной складки белка. Распознавание нитей или складок предполагает, что трехмерная структура более консервативна, чем последовательность. Таким образом, очень расходящиеся последовательности могут иметь схожую структуру. Методы ab initio, с другой стороны, бросают вызов одной из самых больших проблем в молекулярной биологии, а именно, предсказать сворачивание белка «с нуля», не используя гомологичные последовательности или структуры. Используя «фильтр SAXS», авторы смогли значительно очистить набор моделей белков de novo. [17] Это было дополнительно доказано поиском гомологии структур . Было также показано, что сочетание оценок SAXS с оценками, используемыми в методах потоковой передачи, значительно улучшает производительность распознавания складок. [16] На одном примере было продемонстрировано, как приблизительная третичная структура модульных белков может быть собрана из структур доменов ЯМР высокого разрешения, используя данные SAXS, ограничивая трансляционные степени свободы. [18] Другой пример показывает, как данные SAXS могут быть объединены вместе с данными ЯМР, рентгеновской кристаллографией и электронной микроскопией для реконструкции четвертичной структуры многодоменного белка. [19]
Недавно был предложен элегантный метод решения проблемы внутренне неупорядоченных или многодоменных белков с гибкими линкерами . [20] Он допускает сосуществование различных конформаций белка, которые вместе вносят вклад в среднюю экспериментальную картину рассеяния. Первоначально EOM (метод оптимизации ансамбля) генерирует пул моделей, охватывающих пространство конфигурации белка. Затем для каждой модели рассчитывается кривая рассеяния. На втором этапе программа выбирает подмножества моделей белка. Среднее экспериментальное рассеяние рассчитывается для каждого подмножества и подгоняется под экспериментальные данные SAXS. Если наилучшее соответствие не найдено, модели перетасовываются между различными подмножествами и выполняется новый расчет среднего рассеяния и подгонка под экспериментальные данные. Этот метод был протестирован на двух белках – денатурированном лизоциме и протеинкиназе Брутона . Он дал некоторые интересные и многообещающие результаты. [20]
Покрытия биомолекул можно изучать с помощью скользящего рентгеновского и нейтронного рассеяния. IsGISAXS ( скользящее рентгеновское рассеяние с малым углом ) — это программное обеспечение, предназначенное для моделирования и анализа GISAXS из наноструктур. IsGISAXS охватывает только рассеяние частицами нанометрового размера, которые зарыты в подповерхность матрицы или поддерживаются на подложке или зарыты тонким слоем на подложке. Случай отверстий также обрабатывается. Геометрия ограничена плоскостью частиц. Сечение рассеяния разлагается в терминах интерференционной функции и форм-фактора частицы . Акцент делается на геометрии скользящего падения, которая вызывает «эффект преломления пучка». Форм-фактор частицы вычисляется в рамках приближения Борна искаженной волны (DWBA), начиная с невозмущенного состояния с резкими интерфейсами или с фактическим перпендикулярным профилем показателя преломления . Различные виды простых геометрических форм доступны с полным учетом распределения размеров и форм в приближении развязки (DA), в локальном монодисперсном приближении (LMA), а также в приближении корреляции размеров и расстояний (SSCA). Рассматриваются как неупорядоченные системы частиц, определяемые их парной корреляционной функцией частица-частица , так и двумерный кристалл или паракристалл. [21]