Геномная библиотека — это совокупность перекрывающихся фрагментов ДНК, которые вместе составляют полную геномную ДНК одного организма . ДНК хранится в популяции идентичных векторов , каждый из которых содержит разные вставки ДНК. Чтобы создать геномную библиотеку, ДНК организма извлекается из клеток , а затем расщепляется ферментом рестрикции , чтобы разрезать ДНК на фрагменты определенного размера. Затем фрагменты вставляются в вектор с помощью ДНК-лигазы . [1] Затем векторная ДНК может быть поглощена организмом-хозяином - обычно популяцией Escherichia coli или дрожжей - при этом каждая клетка содержит только одну векторную молекулу. Использование клетки-хозяина для переноса вектора позволяет легко амплифицировать и извлекать определенные клоны из библиотеки для анализа. [2]
Доступно несколько типов векторов с различной емкостью вставки. Как правило, библиотеки, созданные из организмов с более крупными геномами, требуют векторов с более крупными вставками, поэтому для создания библиотеки требуется меньше векторных молекул. Исследователи могут выбрать вектор, учитывая также идеальный размер вставки, чтобы найти желаемое количество клонов, необходимое для полного покрытия генома. [3]
Геномные библиотеки обычно используются для секвенирования . Они сыграли важную роль в секвенировании всего генома нескольких организмов, включая геном человека и несколько модельных организмов . [4] [5]
Первый геном на основе ДНК , когда-либо полностью секвенированный, был получен двукратным лауреатом Нобелевской премии Фредериком Сэнгером в 1977 году. Сэнгер и его команда ученых создали библиотеку бактериофага phi X 174 для использования в секвенировании ДНК . [6] Важность этого успеха способствовала постоянно растущему спросу на секвенирование геномов для исследования генной терапии . Теперь команды могут каталогизировать полиморфизмы в геномах и исследовать те гены-кандидаты, которые способствуют развитию таких заболеваний, как болезнь Паркинсона , болезнь Альцгеймера , рассеянный склероз , ревматоидный артрит и диабет 1 типа . [7] Это связано с развитием полногеномных ассоциативных исследований, позволяющих создавать и секвенировать геномные библиотеки. Предварительные исследования сцепления и генов-кандидатов были одними из единственных подходов. [8]
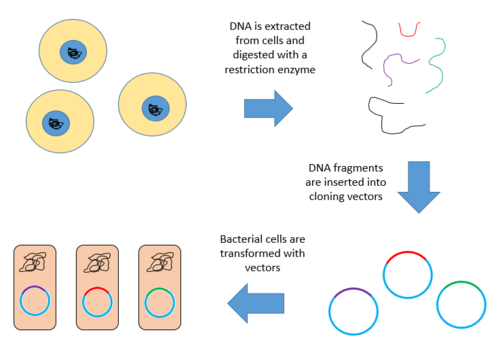
Создание геномной библиотеки предполагает создание множества молекул рекомбинантной ДНК . Геномная ДНК организма извлекается, а затем переваривается ферментом рестрикции . Для организмов с очень маленькими геномами (~10 т.п.н.) переваренные фрагменты можно разделить с помощью гель-электрофореза . Отделенные фрагменты затем могут быть вырезаны и клонированы в вектор отдельно. Однако когда большой геном переваривается ферментом рестрикции, остается слишком много фрагментов, чтобы их можно было вырезать по отдельности. Весь набор фрагментов необходимо клонировать вместе с вектором, после чего может произойти разделение клонов. В любом случае фрагменты лигируют в вектор, расщепленный тем же ферментом рестрикции. Вектор, содержащий встроенные фрагменты геномной ДНК, затем может быть введен в организм хозяина. [1]
Ниже приведены шаги по созданию геномной библиотеки из большого генома.
Ниже представлена схема описанных выше шагов.
После создания геномной библиотеки с использованием вирусного вектора, такого как лямбда-фаг , можно определить титр библиотеки. Расчет титра позволяет исследователям приблизительно оценить, сколько инфекционных вирусных частиц было успешно создано в библиотеке. Для этого разведениями библиотеки трансформируют культуры кишечной палочки известных концентраций. Затем культуры высеивают на чашки с агаром и инкубируют в течение ночи. Количество вирусных бляшек подсчитывается и может быть использовано для расчета общего количества инфекционных вирусных частиц в библиотеке. Большинство вирусных векторов также несут маркер, позволяющий отличить клоны, содержащие вставку, от клонов, не имеющих вставки. Это позволяет исследователям также определить процент инфекционных вирусных частиц, действительно несущих фрагмент библиотеки. [11]
Подобный метод можно использовать для титрования геномных библиотек, созданных с использованием невирусных векторов, таких как плазмиды и BAC . Тестовое лигирование библиотеки можно использовать для трансформации E.coli. Затем трансформацию распределяют по чашкам с агаром и инкубируют в течение ночи. Титр трансформации определяют путем подсчета количества колоний, присутствующих на чашках. Эти векторы обычно имеют селектируемый маркер , позволяющий отличать клоны, содержащие вставку, от клонов, которые ее не содержат. Выполняя этот тест, исследователи также могут определить эффективность лигирования и при необходимости внести коррективы, чтобы гарантировать получение желаемого количества клонов для библиотеки. [12]

Чтобы изолировать из библиотеки клоны, содержащие интересующие области, библиотеку необходимо сначала проверить . Одним из методов скрининга является гибридизация . Каждая трансформированная клетка-хозяин библиотеки будет содержать только один вектор с одной вставкой ДНК. Всю библиотеку можно поместить на фильтр поверх носителя . Фильтр и колонии готовят к гибридизации, а затем метят зондом . [13] Целевая ДНК-вставка, представляющая интерес, может быть идентифицирована путем обнаружения, например авторадиографии , благодаря гибридизации с зондом, как показано ниже.
Другой метод скрининга – полимеразная цепная реакция (ПЦР). Некоторые библиотеки хранятся в виде пулов клонов, и скрининг с помощью ПЦР является эффективным способом идентификации пулов, содержащих определенные клоны. [2]
Размер генома различается у разных организмов, и вектор клонирования необходимо выбирать соответствующим образом. Для большого генома следует выбирать вектор с большой емкостью, чтобы относительно небольшого количества клонов было достаточно для покрытия всего генома. Однако зачастую бывает сложнее охарактеризовать вставку , содержащуюся в векторе более высокой емкости. [3]
Ниже приведена таблица нескольких типов векторов, обычно используемых в геномных библиотеках, а также размер вставки, который обычно соответствует каждому из них.
Плазмида представляет собой двухцепочечную кольцевую молекулу ДНК, обычно используемую для молекулярного клонирования . Плазмиды обычно имеют длину от 2 до 4 тысяч оснований (кб) и способны нести вставки длиной до 15 кб. Плазмиды содержат точку начала репликации , позволяющую им реплицироваться внутри бактерии независимо от хромосомы хозяина . Плазмиды обычно несут ген устойчивости к антибиотикам , который позволяет отбирать бактериальные клетки, содержащие плазмиду. Многие плазмиды также несут репортерный ген , который позволяет исследователям отличать клоны, содержащие вставку, от тех, которые ее не содержат. [3]
Фаг λ представляет собой двухцепочечный ДНК-вирус , инфицирующий E. coli . Х-хромосома имеет длину 48,5 КБ и может нести вставки длиной до 25 КБ. Эти вставки заменяют несущественные вирусные последовательности в λ-хромосоме, в то время как гены, необходимые для образования вирусных частиц и инфекции, остаются интактными. ДНК-вставка реплицируется с вирусной ДНК; таким образом, вместе они упаковываются в вирусные частицы. Эти частицы очень эффективны при инфицировании и размножении, что приводит к более высокому производству рекомбинантных λ-хромосом. [3] Однако из-за меньшего размера вставки библиотекам, созданным с использованием фага λ, может потребоваться множество клонов для полного покрытия генома. [14]
Космидные векторы представляют собой плазмиды, которые содержат небольшой участок ДНК бактериофага λ, называемый последовательностью cos. Эта последовательность позволяет упаковать космиду в частицы бактериофага λ. Эти частицы, содержащие линеаризованную космиду, вводятся в клетку-хозяина путем трансдукции . Попав внутрь хозяина, космиды циркулируют с помощью ДНК-лигазы хозяина и затем функционируют как плазмиды. Космиды способны переносить вставки размером до 40 КБ. [2]
Векторы бактериофага P1 могут содержать вставки размером 70–100 КБ. Они начинаются с линейных молекул ДНК, упакованных в частицы бактериофага P1. Эти частицы вводятся в штамм E. coli, экспрессирующий рекомбиназу Cre . Линейный вектор P1 становится кольцевым в результате рекомбинации между двумя сайтами loxP в векторе. Векторы P1 обычно содержат ген устойчивости к антибиотикам и положительный маркер селекции, позволяющий отличать клоны, содержащие вставку, от клонов, которые ее не содержат. Векторы P1 также содержат репликон плазмиды P1 , который гарантирует присутствие только одной копии вектора в клетке. Однако существует второй репликон P1, называемый литическим репликоном P1, который контролируется индуцибельным промотором . Этот промотор позволяет амплифицировать более одной копии вектора на клетку перед экстракцией ДНК . [2]

Искусственные хромосомы P1 (PAC) имеют характеристики как векторов P1, так и бактериальных искусственных хромосом (BAC). Подобно векторам P1, они содержат плазмиду и литический репликон, как описано выше. В отличие от векторов P1, их не нужно упаковывать в частицы бактериофага для трансдукции. Вместо этого они вводятся в E. coli в виде кольцевых молекул ДНК посредством электропорации , как и BAC. [2] Также как и BAC, их относительно сложнее приготовить из-за единственного источника репликации. [14]
Бактериальные искусственные хромосомы (BAC) представляют собой кольцевые молекулы ДНК, обычно длиной около 7 КБ, которые способны содержать вставки размером до 300 КБ. Векторы BAC содержат репликон, полученный из F-фактора E. coli , что гарантирует сохранение одной копии на клетку. [4] После того как вставка лигируется в BAC, BAC вводится в штаммы E. coli с дефицитом рекомбинации посредством электропорации. Большинство векторов BAC содержат ген устойчивости к антибиотикам, а также положительный маркер селекции. [2] На рисунке справа изображен вектор BAC, разрезаемый ферментом рестрикции с последующей вставкой чужеродной ДНК, которая повторно отжигается лигазой. В целом, это очень стабильный вектор, но его может быть сложно подготовить из-за единственного источника репликации, как и PAC. [14]
Искусственные дрожжевые хромосомы (YAC) представляют собой линейные молекулы ДНК, содержащие необходимые функции аутентичной дрожжевой хромосомы, включая теломеры , центромеры и точку начала репликации . Большие вставки ДНК можно лигировать в середину YAC так, чтобы по обе стороны от вставки было «плечо» YAC. Рекомбинантный YAC вводят в дрожжи путем трансформации; селектируемые маркеры , присутствующие в YAC, позволяют идентифицировать успешные трансформанты. YAC могут содержать вставки размером до 2000 КБ, но большинство библиотек YAC содержат вставки размером 250–400 КБ. Теоретически не существует верхнего предела размера вставки, которую может вместить YAC. Предельный размер определяется качеством подготовки ДНК, используемой для вставок. [2] Самым сложным аспектом использования YAC является тот факт, что они склонны к перестановке . [14]
Выбор вектора требует, чтобы созданная библиотека была репрезентативной для всего генома. Любая вставка генома, полученная из фермента рестрикции, должна иметь равные шансы оказаться в библиотеке по сравнению с любой другой вставкой. Кроме того, рекомбинантные молекулы должны содержать достаточно большие вставки, обеспечивающие удобство обращения с размером библиотеки. [14] Это, в частности, определяется количеством клонов, которые необходимо иметь в библиотеке. Количество клонов для получения выборки всех генов определяется размером генома организма, а также средним размером вставки. Это представлено формулой (также известной как формула Карбона и Кларка): [15]
где,
– необходимое количество рекомбинантов [16]
— желаемая вероятность того, что любой фрагмент генома хотя бы один раз встретится в созданной библиотеке.
представляет собой дробную долю генома в одном рекомбинантном
можно дополнительно показать:
где,
это размер вставки
размер генома
Таким образом, увеличение размера вставки (по выбору вектора) позволит использовать меньше клонов, необходимых для представления генома. Пропорция размера вставки по отношению к размеру генома представляет собой долю соответствующего генома в одном клоне. [14] Вот уравнение со всеми рассмотренными частями:
Приведенную выше формулу можно использовать для определения уровня уверенности 99% в том, что все последовательности в геноме представлены с использованием вектора с размером вставки в двадцать тысяч пар оснований (например, фагового лямбда-вектора). Размер генома организма в этом примере составляет три миллиарда пар оснований.
клоны
Таким образом, требуется примерно 688 060 клонов, чтобы гарантировать 99% вероятность того, что данная последовательность ДНК из этого генома, состоящего из трех миллиардов пар оснований, будет присутствовать в библиотеке с использованием вектора с размером вставки в двадцать тысяч пар оснований.
После создания библиотеки геном организма можно секвенировать , чтобы выяснить, как гены влияют на организм, или сравнить похожие организмы на уровне генома. Вышеупомянутые полногеномные исследования ассоциаций могут идентифицировать гены-кандидаты, обусловленные многими функциональными признаками. Гены можно выделить с помощью геномных библиотек и использовать на линиях клеток человека или моделях животных для дальнейших исследований. [17] Кроме того, создание высокоточных клонов с точным представлением генома и отсутствием проблем со стабильностью могло бы стать хорошим промежуточным продуктом для секвенирования дробовика или изучения полных генов в функциональном анализе. [10]

Одним из основных применений геномных библиотек является иерархическое секвенирование методом дробовика , которое также называется секвенированием сверху вниз, на основе карт или секвенированием по клонам. Эта стратегия была разработана в 1980-х годах для секвенирования целых геномов, прежде чем стали доступны высокопроизводительные методы секвенирования. Отдельные клоны из геномных библиотек можно разделить на более мелкие фрагменты, обычно размером от 500 до 1000 пар оснований, которые более удобны для секвенирования. [4] После секвенирования клона из геномной библиотеки эту последовательность можно использовать для скрининга библиотеки на наличие других клонов, содержащих вставки, которые перекрываются с секвенированным клоном. Любые новые перекрывающиеся клоны затем можно секвенировать, образуя контиг . Этот метод, называемый « прогулкой по хромосоме» , можно использовать для секвенирования целых хромосом. [2]
Полногеномное секвенирование — это еще один метод секвенирования генома, который не требует библиотеки векторов высокой емкости. Скорее, он использует компьютерные алгоритмы для сборки коротких считываний последовательностей, чтобы охватить весь геном. По этой причине геномные библиотеки часто используются в сочетании с полногеномным секвенированием. Карта высокого разрешения может быть создана путем секвенирования обоих концов вставок из нескольких клонов в геномной библиотеке. На этой карте представлены последовательности на известных расстояниях друг от друга, которые можно использовать для облегчения сборки считываний последовательностей, полученных с помощью дробовика. [4] Последовательность генома человека, объявленная завершенной в 2003 году, была собрана с использованием как библиотеки BAC, так и методом дробового секвенирования. [18] [19]
Полногеномные исследования ассоциаций представляют собой общие приложения для поиска конкретных генов-мишеней и полиморфизмов внутри человеческой расы. Фактически, международный проект HapMap был создан в результате партнерства ученых и агентств из нескольких стран для каталогизации и использования этих данных. [20] Целью этого проекта является сравнение генетических последовательностей разных людей для выяснения сходств и различий внутри хромосомных областей. [20] Ученые из всех участвующих стран каталогизируют эти атрибуты, используя данные о популяциях африканского, азиатского и европейского происхождения. Такие полногеномные оценки могут привести к дальнейшей диагностике и медикаментозной терапии, а также помогут будущим командам сосредоточиться на разработке терапии с учетом генетических особенностей. Эти концепции уже используются в генной инженерии . [20] Например, исследовательская группа фактически создала челночный вектор PAC, который создает библиотеку, представляющую двойной охват человеческого генома. [17] Это может послужить невероятным ресурсом для выявления генов или наборов генов, вызывающих заболевания. Более того, эти исследования могут служить мощным способом изучения регуляции транскрипции, как это было замечено при изучении бакуловирусов. [21] В целом, достижения в области создания геномных библиотек и секвенирования ДНК позволили эффективно обнаруживать различные молекулярные мишени. [5] Усвоение этих особенностей с помощью таких эффективных методов может ускорить использование новых кандидатов на лекарства.
Клуг, Каммингс, Спенсер, Палладино (2010). Основы генетики . Пирсон. стр. 355–264. ISBN 978-0-321-61869-6.{{cite book}}: CS1 maint: multiple names: authors list (link)