
Ленточные диаграммы , также известные как диаграммы Ричардсона , представляют собой трехмерное схематическое представление структуры белка и являются одним из наиболее распространенных методов изображения белков, используемых сегодня. Лента изображает общий ход и организацию основной цепи белка в 3D и служит визуальной основой для подвешивания деталей всей атомной структуры, таких как шарики атомов кислорода, прикрепленных к активному центру миоглобина на рисунке рядом. Ленточные диаграммы создаются путем интерполяции плавной кривой, проходящей через основную цепь полипептида . α-спирали показаны в виде скрученных лент или толстых трубок, β-листы — в виде стрелок, а неповторяющиеся витки или петли — в виде линий или тонких трубок. Направление полипептидной цепи показано локально стрелками и в целом может быть указано цветовой шкалой по длине ленты. [1]
Ленточные диаграммы просты, но эффективны и выражают визуальные основы молекулярной структуры (скручивание, сгибание и разворачивание). Этот метод успешно отображает общую организацию белковых структур, отражая их трехмерную природу и позволяя лучше понять эти сложные объекты как экспертам-структурным биологам, так и другим ученым, студентам [2] и широкой публике.

Первые ленточные диаграммы, нарисованные от руки Джейн С. Ричардсон в 1980 году (под влиянием более ранних отдельных иллюстраций) [3] , были первыми схематическими схемами трехмерной структуры белка, которые создавались систематически. [3] [4] Они были созданы, чтобы проиллюстрировать классификацию белковых структур для статьи в журнале « Advances in Protein Chemistry» [5] (теперь доступной в аннотированной форме онлайн на сайте Anatax). Эти рисунки обводились ручкой на кальке поверх распечатки Cα- следа координат атомов и заштриховывались цветным карандашом или пастелью; [6] они сохранили позиции, сгладили магистральный путь и внесли небольшие локальные изменения, чтобы устранить неоднозначность внешнего вида. [4] Помимо рисунка ленты триозоизомеразы справа, на других нарисованных от руки примерах изображены преальбумин, флаводоксин и супероксиддисмутаза Cu,Zn.
В 1982 году Артур М. Леск и его коллеги впервые включили автоматическое создание ленточных диаграмм с помощью вычислительной реализации, которая использует файлы банка данных белков в качестве входных данных. [7] Этот концептуально простой алгоритм подгоняет кривые B-сплайна кубического полинома к пептидным плоскостям. Большинство современных графических систем предоставляют в качестве базового примитива рисования либо B-сплайны, либо сплайны Эрмита . Через каждую направляющую точку Cα проходит один тип реализации сплайна, создавая точную, но прерывистую кривую. Как нарисованные от руки, так и большинство компьютерных лент (например, показанных здесь) сглаживаются примерно по четырем последовательным направляющим точкам (обычно средней точке пептида), чтобы получить более визуально приятное и понятное изображение. Чтобы придать правильный радиус спиральным спиралям, сохраняя при этом гладкие β-нити, сплайны можно модифицировать путем смещения, пропорционального локальной кривизне, как это впервые было разработано Майком Карсоном для его программы Ribbons [8] и позже адаптировано с помощью других программ молекулярной графики, таких как как программа Mage с открытым исходным кодом для кинематической графики [9] , которая создавала изображение ленты в правом верхнем углу (другие примеры: тример 1XK8 и ДНК-полимераза).
С момента своего появления и до сих пор ленточные диаграммы являются единственным наиболее распространенным представлением структуры белка и частым выбором изображения обложки журнала или учебника.
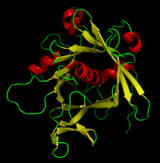
Одной из популярных программ, используемых для рисования ленточных диаграмм, является Molscript. Molscript использует сплайны Эрмита для создания координат витков, витков, прядей и спиралей. Кривая проходит через все свои контрольные точки ( атомы Cα ), руководствуясь векторами направления. Программа была построена на основе традиционной молекулярной графики Артуром М. Леском , Карлом Хардманом и Джоном Пристлом. [10] Jmol — это программа просмотра на основе Java с открытым исходным кодом для просмотра молекулярных структур в Интернете; он включает упрощенную «мультяшную» версию лент. Другие графические программы, такие как DeepView (пример: urease) и MolMol (пример: домен SH2), также создают ленточные изображения. KiNG [11] является преемником Mage на основе Java (примеры: вид сверху и вид сбоку α-гемолизина).
UCSF Chimera — это мощная программа молекулярного моделирования, которая также включает в себя такие визуализации , как ленты, особенно примечательные возможностью комбинировать их с контурными формами из данных криоэлектронной микроскопии . [12] PyMOL , автор Уоррен ДеЛано , [13] — популярная и гибкая программа молекулярной графики (на основе Python ), которая работает в интерактивном режиме, а также создает 2D-изображения презентационного качества для ленточных диаграмм и многих других представлений.
