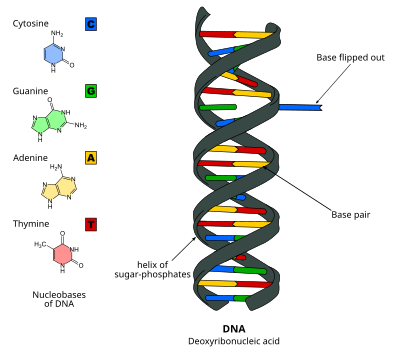
Переворот основания ДНК , или переворот нуклеотида , представляет собой механизм, при котором одно нуклеотидное основание, или нуклеиновое основание , поворачивается за пределы двойной спирали нуклеиновой кислоты. [1] Это происходит, когда ферменту , обрабатывающему нуклеиновую кислоту, требуется доступ к основанию для выполнения работы над ним, например, его вырезания для замены другим основанием во время репарации ДНК . Впервые это наблюдалось в 1994 году с помощью рентгеновской кристаллографии в ферменте метилтрансферазе , катализирующем метилирование цитозинового основания в ДНК. С тех пор было показано, что оно используется различными ферментами во многих биологических процессах, таких как метилирование ДНК , различные механизмы репарации ДНК и репликация ДНК . Это также может происходить в двойных спиралях РНК [2] или в промежуточных соединениях ДНК:РНК, образующихся во время транскрипции РНК .
Переворачивание оснований ДНК происходит путем разрыва водородных связей между основаниями и отсоединения основания от его соседей. Это может происходить посредством активного процесса, когда фермент связывается с ДНК, а затем облегчает вращение основания, или пассивного процесса, когда основание спонтанно выворачивается, и это состояние распознается и связывается ферментом. Его можно обнаружить с помощью рентгеновской кристаллографии , ЯМР-спектроскопии , флуоресцентной спектроскопии или гибридизационных зондов .
Переворачивание оснований впервые наблюдалось в 1994 году, когда исследователи Климасаускас, Кумар, Робертс и Ченг использовали рентгеновскую кристаллографию для наблюдения за промежуточным этапом химической реакции метилтрансферазы, связанной с ДНК . [3] Метилтрансфераза, которую они использовали, была C5-цитозинметилтрансферазой из Haemophilus haemolyticus (M. HhaI). Этот фермент распознает определенную последовательность ДНК (5'-GCGC-3') и метилирует первое основание цитозина последовательности в его положении C5. [3] После кристаллизации комплекса M. HhaI-ДНК они увидели, что целевое основание цитозина было полностью повернуто из двойной спирали и было расположено в активном центре M. HhaI. Оно удерживалось на месте многочисленными взаимодействиями между M. HhaI и ДНК. [3]
Авторы предположили, что переворот основания — это механизм, используемый многими другими ферментами, такими как геликазы , ферменты рекомбинации , РНК-полимеразы , ДНК-полимеразы и топоизомеразы типа II . [3] За годы, прошедшие после этого открытия, было проведено много исследований, и было обнаружено, что переворот основания — это механизм, используемый во многих биологических процессах, которые предполагают авторы. [4] [5] [6]

Нуклеотиды ДНК удерживаются вместе водородными связями , которые относительно слабы и могут быть легко разорваны. Переворот основания происходит в течение миллисекунд [7] путем разрыва водородных связей между основаниями и отсоединения основания от его соседей. [8] Основание поворачивается из двойной спирали на 180 градусов, [9] как правило, через большую бороздку , [10] и в активный центр фермента. Это открытие приводит к небольшим конформационным изменениям в остове ДНК [11] , которые быстро стабилизируются за счет усиления взаимодействий фермент-ДНК. [12] Исследования, изучающие профили свободной энергии переворота основания, показали, что барьер свободной энергии для переворота может быть снижен на 17 ккал/моль для M.HhaI в закрытой конформации . [10]
Существует два механизма переворачивания оснований ДНК: активный и пассивный. [13] В активном механизме фермент связывается с ДНК, а затем активно вращает основание, в то время как в пассивном механизме поврежденное основание сначала спонтанно вращается, а затем распознается и связывается ферментом. [8] Исследования продемонстрировали оба механизма: урацил-ДНК-гликозилаза следует пассивному механизму [8] , а транспозаза Tn10 следует активному механизму. [14]
Более того, исследования показали, что переворачивание оснований ДНК используется многими различными ферментами в различных биологических процессах, таких как метилирование ДНК , различные механизмы репарации ДНК , транскрипция РНК и репликация ДНК . [4] [5] [6]

ДНК может иметь мутации , которые приводят к повреждению основания в цепи ДНК. Чтобы обеспечить генетическую целостность ДНК, ферменты должны восстанавливать любые повреждения. Существует много типов репарации ДНК . Репарация эксцизии оснований использует переворачивание оснований для переворачивания поврежденного основания из двойной спирали [5] в карман специфичности гликозилазы , которая гидролизует гликозидную связь и удаляет основание. [15] ДНК-гликозилазы взаимодействуют с ДНК, переворачивая основания для определения несоответствия. Пример репарации эксцизии оснований происходит, когда основание цитозина дезаминируется и становится основанием урацила. Это вызывает ошибочную пару U:G, которая обнаруживается урацил-ДНК-гликозилазой . Основание урацила переворачивается в активный карман гликозилазы, где оно удаляется из цепи ДНК. [16] Переворачивание оснований используется для исправления мутаций, таких как 8-оксогуанин (oxoG) [17] и димеры тимина, созданные УФ-излучением. [15] [18]
Репликация ДНК и транскрипция РНК используют переворачивание оснований. [5] ДНК-полимераза — это фермент, который осуществляет репликацию. Ее можно представить как руку, которая захватывает шаблон одноцепочечной ДНК. [15] Когда шаблон проходит через область ладони полимеразы, основания шаблона переворачиваются из спирали и от сайта связывания dNTP . [19] Во время транскрипции РНК-полимераза катализирует синтез РНК. Во время фазы инициации два основания в элементе -10 переворачиваются из спирали и попадают в два кармана в РНК-полимеразе. Эти новые взаимодействия стабилизируют элемент -10 и способствуют разделению или расплавлению цепей ДНК. [15] [20]
Переворачивание оснований происходит на последних стадиях рекомбинации . [21] RecA — это белок, который способствует инвазии нитей [15] во время гомологичной рекомбинации . Переворачивание оснований было предложено как механизм, с помощью которого RecA может позволить одной нити распознавать гомологию в дуплексной ДНК. [22] Другие исследования показывают, что он также участвует в рекомбинации V(D)J . [23]
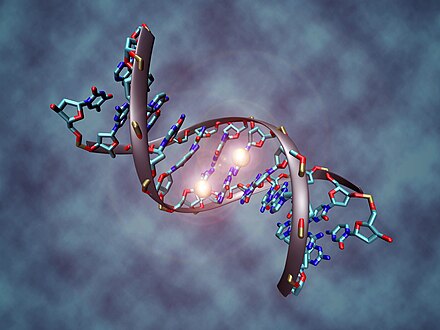
Метилирование ДНК — это процесс, в котором метильная группа добавляется либо к цитозину , либо к аденину . [24] Этот процесс вызывает активацию или инактивацию экспрессии генов , тем самым приводя к регуляции генов в эукариотических клетках. Известно также, что процесс метилирования ДНК участвует в формировании определенных типов рака . [25] [26] [27] Для того чтобы произошла эта химическая модификация, необходимо, чтобы целевое основание вывернулось из двойной спирали ДНК, чтобы позволить метилтрансферазам катализировать реакцию. [5]
Рестрикционные эндонуклеазы, также известные как рестриктазы, являются ферментами, которые расщепляют сахарофосфатный остов ДНК в определенных последовательностях нуклеотидов , которые обычно имеют длину от четырех до шести нуклеотидов. [28] Исследования, проведенные Хортоном и коллегами, показали, что механизм, с помощью которого эти ферменты расщепляют ДНК, включает в себя переворачивание оснований, а также изгиб ДНК и расширение малой бороздки . [29] В 2006 году Хортон и коллеги представили доказательства рентгеновской кристаллографии , показывающие, что рестрикционная эндонуклеаза HinP1I использует переворачивание оснований для распознавания своей целевой последовательности. Известно, что этот фермент расщепляет ДНК в палиндромной тетрануклеотидной последовательности G↓CGC.
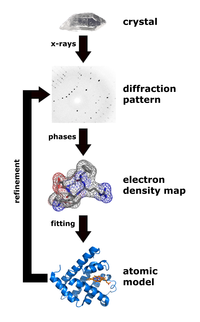
Рентгеновская кристаллография — это метод, который измеряет углы и интенсивности кристаллических атомов для определения атомной и молекулярной структуры интересующего кристалла. Затем кристаллографы могут создать трехмерную картину, на которой можно определить положения атомов , химические связи , а также другие важные характеристики. [30] Климасаукас и его коллеги использовали этот метод для наблюдения явления переворота первой базы, в котором их экспериментальная процедура включала несколько этапов: [3]
Во время очистки метилтрансфераза Haemophilus haemolyticus была сверхэкспрессирована и очищена с использованием этапа обратной экстракции высокой соли для селективной солюбилизации M.HhaI, за которым последовала быстрая жидкостная хроматография белка ( FPLC ), как это ранее делали Кумар и коллеги. [31] Авторы использовали анионообменную колонку Mono-Q для удаления небольшого количества белковых материалов и нежелательной ДНК перед этапом кристаллизации. После успешной очистки M.HhaI образец затем выращивали с использованием метода, который смешивает раствор, содержащий комплекс, при температуре 16 °C и метода диффузии паров в висячей капле для получения кристаллов. Затем авторы смогли собрать рентгеновские данные в соответствии с методом, использованным Ченгом и коллегами в 1993 году. [32] Этот метод включал измерение интенсивности дифракции на детекторе FAST, где время экспозиции для вращения на 0,1° составляло 5 или 10 секунд. Для определения и уточнения структуры Климасаукас и его коллеги использовали молекулярную замену уточненной структуры апо, описанной Ченгом и его коллегами в 1993 году [32] , где для решения функций вращения и трансляции использовались поисковые модели X-PLOR , MERLOT и TRNSUM. [33] [34] Эта часть исследования включает использование разнообразного программного обеспечения и компьютерных алгоритмов для решения структур и характеристик интересующего кристалла.
Спектроскопия ЯМР — это метод, который использовался на протяжении многих лет для изучения важных динамических аспектов переворота оснований. Этот метод позволяет исследователям определять физические и химические свойства атомов и других молекул, используя магнитные свойства атомных ядер . [35] Кроме того, ЯМР может предоставить разнообразную информацию, включая структуру, состояния реакции , химическое окружение молекул и динамику. [36] [37] Во время эксперимента по обнаружению переворота оснований ДНК исследователи использовали спектроскопию ЯМР для исследования переворота оснований метилтрансферазы HhaI, вызванного ферментом. Для выполнения этого эксперимента два остатка 5-фторцитозина были включены в целевое и исходное положение с субстратом ДНК, чтобы можно было выполнить анализ химического сдвига 19 F. После оценки анализа химического сдвига 19 F был сделан вывод о том, что комплексы ДНК существуют с несколькими формами целевого 5-фторцитозина вдоль пути переворота оснований. [38]
Флуоресцентная спектроскопия — это метод, который используется для анализа образца с использованием флуоресцентного зонда. Сами по себе нуклеотиды ДНК не являются хорошими кандидатами для этого метода, поскольку они нелегко повторно испускают свет при возбуждении светом. [39] Для обнаружения переворота оснований необходим флуоресцентный маркер. 2-аминопурин — это основание, которое структурно похоже на аденин , но очень флуоресцентно при перевороте из дуплекса ДНК. [40] Он обычно используется для обнаружения переворота оснований и имеет возбуждение при 305‑320 нм и испускание при 370 нм, так что он хорошо отделен от возбуждений белков и ДНК. Другие флуоресцентные зонды, используемые для изучения переворота оснований ДНК, — это 6MAP (4-амино-6-метил-7(8H)-птеридон) [41] и Pyrrolo-C (3-[β-D-2-рибофуранозил]-6-метилпирроло[2,3-d]пиримидин-2(3H)-он). [42] [43] Флуоресцентная спектроскопия с временным разрешением также используется для получения более подробной картины степени переворота оснований, а также конформационной динамики, происходящей во время переворота оснований. [44]
Гибридизационные зонды могут использоваться для обнаружения переворота оснований. Этот метод использует молекулу, которая имеет комплементарную последовательность к последовательности, которую вы хотели бы обнаружить, так что она связывается с одной цепочкой ДНК или РНК. Несколько гибридизационных зондов использовались для обнаружения переворота оснований. Перманганат калия используется для обнаружения остатков тимина , которые были перевернуты цитозин-C5 и аденин-N6 метилтрансферазами . [45] Хлороацетальдегид используется для обнаружения остатков цитозина , перевернутых ДНК-цитозин-5 метилтрансферазой HhaI (M. HhaI). [46]
{{cite book}}: |first=имеет общее название ( помощь ){{cite book}}: CS1 maint: несколько имен: список авторов ( ссылка ){{cite book}}: |first=имеет общее название ( помощь )