
Системная биология — это вычислительный и математический анализ и моделирование сложных биологических систем . Это основанная на биологии междисциплинарная область исследований, которая фокусируется на сложных взаимодействиях в биологических системах, используя целостный подход ( холизм вместо более традиционного редукционизма ) к биологическим исследованиям. [1]
В частности, с 2000 года эта концепция широко использовалась в биологии в различных контекстах. Проект «Геном человека» является примером прикладного системного мышления в биологии, который привел к новым, совместным способам работы над проблемами в области биологической генетики. [2] Одной из целей системной биологии является моделирование и обнаружение возникающих свойств , свойств клеток , тканей и организмов, функционирующих как система , теоретическое описание которой возможно только с использованием методов системной биологии. [1] [3] Они обычно включают метаболические сети или сети клеточной сигнализации . [1] [4]
Системную биологию можно рассматривать с разных сторон.
Как область изучения, в частности, изучение взаимодействий между компонентами биологических систем и того, как эти взаимодействия приводят к функционированию и поведению этой системы (например, ферменты и метаболиты в метаболическом пути или сердечные сокращения). [5] [6] [7]
Как парадигма , системная биология обычно определяется в противовес так называемой редукционистской парадигме ( биологической организации ), хотя она согласуется с научным методом . Различие между двумя парадигмами упоминается в следующих цитатах: « редукционистский подход успешно идентифицировал большинство компонентов и многие взаимодействия, но, к сожалению, не предлагает убедительных концепций или методов для понимания того, как возникают системные свойства... плюрализм причин и следствий в биологических сетях лучше всего решать, наблюдая с помощью количественных измерений за несколькими компонентами одновременно и строгой интеграцией данных с математическими моделями». (Зауэр и др. ) [8] «Системная биология... заключается в том, чтобы собирать, а не разбирать, интегрировать, а не редуцировать. Она требует, чтобы мы разработали способы мышления об интеграции, которые были бы такими же строгими, как наши редукционистские программы, но другими. ... Это означает изменение нашей философии в полном смысле этого слова». ( Денис Нобл ) [7]
Как ряд рабочих протоколов, используемых для проведения исследований, а именно цикл, состоящий из теории, аналитического или вычислительного моделирования для предложения конкретных проверяемых гипотез о биологической системе, экспериментальной проверки и последующего использования вновь полученного количественного описания клеток или клеточных процессов для уточнения вычислительной модели или теории. [9] Поскольку целью является модель взаимодействий в системе, экспериментальные методы, которые больше всего подходят для системной биологии, — это те, которые являются общесистемными и пытаются быть как можно более полными. Поэтому транскриптомика , метаболомика , протеомика и высокопроизводительные методы используются для сбора количественных данных для построения и проверки моделей. [10]
Как приложение теории динамических систем к молекулярной биологии . Действительно, фокус на динамике изучаемых систем является основным концептуальным отличием системной биологии от биоинформатики . [11]
Как социально-научное явление, определяемое стратегией достижения интеграции сложных данных о взаимодействиях в биологических системах из различных экспериментальных источников с использованием междисциплинарных инструментов и персонала. [12]
Хотя концепция системного взгляда на клеточную функцию была хорошо понята, по крайней мере, с 1930-х годов, [13] технологические ограничения затрудняли проведение общесистемных измерений. Появление технологии микрочипов в 1990-х годах открыло совершенно новую визу для изучения клеток на системном уровне. В 2000 году в Сиэтле был основан Институт системной биологии в попытке привлечь людей «вычислительного» типа, которых, как считалось, не привлекала академическая обстановка университета. У института не было четкого определения того, что на самом деле представляла собой эта область: грубо говоря, объединение людей из разных областей для использования компьютеров для целостного изучения биологии новыми способами. [14] Кафедра системной биологии в Гарвардской медицинской школе была открыта в 2003 году. [15] В 2006 году было предсказано, что шумиха, вызванная «очень модной» новой концепцией, заставит все крупные университеты нуждаться в кафедре системной биологии, таким образом, будут доступны карьеры для выпускников с минимальными способностями в компьютерном программировании и биологии. [14] В 2006 году Национальный научный фонд выдвинул задачу построить математическую модель всей клетки. [ необходима цитата ] В 2012 году первая цельноклеточная модель Mycoplasma genitalium была получена в лаборатории Covert в Стэнфордском университете. Цельноклеточная модель способна предсказать жизнеспособность клеток M. genitalium в ответ на генетические мутации. [16]
Ранним предшественником системной биологии как отдельной дисциплины, возможно, был системный теоретик Михайло Месарович в 1966 году на международном симпозиуме в Технологическом институте Кейса в Кливленде , штат Огайо, под названием «Теория систем и биология ». Месарович предсказал, что, возможно, в будущем появится такая вещь, как «системная биология». [17] [18] Другими ранними предшественниками, которые сосредоточились на представлении о том, что биология должна анализироваться как система, а не как простая совокупность частей, были анализ метаболического контроля , разработанный Хенриком Качером и Джимом Бернсом [19], позже тщательно пересмотренный, [20] и Рейнхартом Генрихом и Томом Рапопортом , [21] и теория биохимических систем , разработанная Майклом Саважо [22] [23] [24]
По словам Роберта Розена в 1960-х годах, целостная биология к началу 20-го века устарела, поскольку популярнее стала более эмпирическая наука, в которой доминировала молекулярная химия. [18] Вторя ему сорок лет спустя, в 2006 году Клинг пишет, что успех молекулярной биологии на протяжении 20-го века подавил целостные вычислительные методы. [14] К 2011 году Национальные институты здравоохранения выделили гранты на поддержку более десяти центров системной биологии в Соединенных Штатах, [25] но к 2012 году Хантер пишет, что системной биологии еще предстоит пройти определенный путь, чтобы раскрыть свой полный потенциал. Тем не менее, сторонники надеялись, что в будущем она может оказаться более полезной. [26]
Важной вехой в развитии системной биологии стал международный проект Physiome . [ необходима ссылка ]
Согласно интерпретации системной биологии как использования больших наборов данных с использованием междисциплинарных инструментов, типичным применением является метаболомика , которая представляет собой полный набор всех метаболических продуктов, метаболитов , в системе на уровне организма, клетки или ткани. [28]
Элементы, которые могут быть компьютерной базой данных, включают: феномику , организменные вариации фенотипа , поскольку он меняется в течение жизни; геномику , последовательность дезоксирибонуклеиновой кислоты (ДНК) организма, включая внутриорганизменные специфичные вариации клеток (например, вариации длины теломер ); эпигеномику / эпигенетику , организменные и соответствующие клеточно-специфичные факторы регуляции транскриптомики, эмпирически не закодированные в геномной последовательности (например, метилирование ДНК , ацетилирование и деацетилирование гистонов и т. д.); транскриптомику , измерения экспрессии генов в организме, ткани или целых клетках с помощью микрочипов ДНК или последовательного анализа экспрессии генов ; интерферомику , факторы коррекции транскриптов на уровне организма, ткани или клеток (например, интерференция РНК ), протеомику , измерения белков и пептидов на уровне организма, ткани или клеток с помощью двумерного гель-электрофореза , масс-спектрометрии или многомерных методов идентификации белков (продвинутые системы ВЭЖХ в сочетании с масс-спектрометрией ). Поддисциплины включают фосфопротеомику , гликопротеомику и другие методы обнаружения химически модифицированных белков; гликомику , измерения углеводов на уровне организмов, тканей или клеток ; липидомику , измерения липидов на уровне организмов, тканей или клеток . [ необходима ссылка ]
Молекулярные взаимодействия внутри клетки также изучаются, это называется интерактомикой . [29] Дисциплина в этой области изучения — белок-белковые взаимодействия , хотя интерактомика включает взаимодействия других молекул. [ требуется ссылка ] Нейроэлектродинамика , где вычислительная функция компьютера или мозга как динамической системы изучается вместе с ее (био)физическими механизмами; [30] и флюксомика , измерения скоростей метаболических реакций в биологической системе (клетке, ткани или организме). [28]
При подходе к проблеме системной биологии существует два основных подхода. Это подход сверху вниз и подход снизу вверх. Подход сверху вниз учитывает как можно больше системы и в значительной степени опирается на экспериментальные результаты. Метод РНК-Seq является примером экспериментального подхода сверху вниз. Наоборот, подход снизу вверх используется для создания подробных моделей, а также включает экспериментальные данные. Примером подхода снизу вверх является использование моделей цепей для описания простой генной сети. [31]
Различные технологии, используемые для захвата динамических изменений в мРНК, белках и посттрансляционных модификациях. Механобиология , силы и физические свойства на всех уровнях, их взаимодействие с другими регуляторными механизмами; [32] биосемиотика , анализ системы знаковых отношений организма или других биосистем; Физиомика , систематическое изучение физиома в биологии.
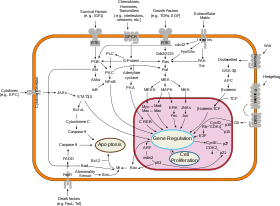
Системная биология рака является примером подхода системной биологии, который можно различить по конкретному объекту исследования ( опухолеобразование и лечение рака ). Он работает с конкретными данными (образцы пациентов, высокопроизводительные данные с особым вниманием к характеристике генома рака в образцах опухолей пациентов) и инструментами (иммортализованные линии раковых клеток , мышиные модели опухолеобразования, модели ксенотрансплантатов , высокопроизводительные методы секвенирования , высокопроизводительные скрининги на основе siRNA, подавляющие гены , вычислительное моделирование последствий соматических мутаций и нестабильности генома ). [33] Долгосрочная цель системной биологии рака — способность лучше диагностировать рак, классифицировать его и лучше предсказывать результат предлагаемого лечения, что является основой для персонализированной онкологической медицины и виртуального онкологического пациента в более отдаленной перспективе. Значительные усилия в области вычислительной системной биологии рака были предприняты для создания реалистичных многомасштабных моделей in silico различных опухолей. [34]
Подход системной биологии часто включает разработку механистических моделей, таких как реконструкция динамических систем из количественных свойств их элементарных строительных блоков. [35] [36] [37] [38] Например, клеточная сеть может быть смоделирована математически с использованием методов, взятых из химической кинетики [39] и теории управления . Из-за большого количества параметров, переменных и ограничений в клеточных сетях часто используются численные и вычислительные методы (например, анализ баланса потоков ). [37] [39]
Другие аспекты компьютерной науки, информатики и статистики также используются в системной биологии. К ним относятся новые формы вычислительных моделей, такие как использование исчислений процессов для моделирования биологических процессов (примечательные подходы включают стохастическое π-исчисление , BioAmbients, Beta Binders, BioPEPA и Brane calculus) и моделирование на основе ограничений ; интеграция информации из литературы с использованием методов извлечения информации и интеллектуального анализа текста ; [40] разработка онлайн-баз данных и репозиториев для обмена данными и моделями, подходы к интеграции баз данных и совместимости программного обеспечения через слабое связывание программного обеспечения, веб-сайтов и баз данных или коммерческих костюмов; сетевые подходы для анализа наборов геномных данных высокой размерности. Например, анализ сетей с весовой корреляцией часто используется для идентификации кластеров (называемых модулями), моделирования взаимосвязи между кластерами, вычисления нечетких мер членства в кластере (модуле), идентификации внутримодульных концентраторов и для изучения сохранения кластеров в других наборах данных; Методы анализа данных омики на основе путей, например, подходы к идентификации и оценке путей с дифференциальной активностью их генов, белков или метаболитов. [41] Большая часть анализа наборов геномных данных также включает выявление корреляций. Кроме того, поскольку большая часть информации поступает из разных областей, необходима разработка синтаксически и семантически обоснованных способов представления биологических моделей. [42]

Исследователи начинают с выбора биологического пути и построения диаграммы всех белковых, генных и/или метаболических путей. После определения всех взаимодействий кинетика действия масс или законы скорости ферментативной кинетики используются для описания скорости реакций в системе. Используя сохранение массы, можно построить дифференциальные уравнения для биологической системы. Можно провести эксперименты или подобрать параметры, чтобы определить значения параметров для использования в дифференциальных уравнениях . [44] Эти значения параметров будут различными кинетическими константами, необходимыми для полного описания модели. Эта модель определяет поведение видов в биологических системах и привносит новое понимание в конкретные действия системы. Иногда невозможно собрать все скорости реакции системы. Неизвестные скорости реакции определяются путем моделирования модели известных параметров и целевого поведения, которое обеспечивает возможные значения параметров. [45] [43]
Использование методов реконструкции и анализа на основе ограничений (COBRA) стало популярным среди системных биологов для моделирования и прогнозирования метаболических фенотипов с использованием моделей в масштабе генома. Одним из методов является подход анализа баланса потока (FBA), с помощью которого можно изучать биохимические сети и анализировать поток метаболитов через определенную метаболическую сеть, оптимизируя целевую функцию интереса (например, максимизируя производство биомассы для прогнозирования роста). [46]

{{cite journal}}: Цитировать журнал требует |journal=( помощь ){{cite journal}}: Цитировать журнал требует |journal=( помощь ){{cite journal}}: Цитировать журнал требует |journal=( помощь )