В биоинформатике сборка последовательности означает выравнивание и слияние фрагментов из более длинной последовательности ДНК с целью реконструкции исходной последовательности. [1] Это необходимо, поскольку технология секвенирования ДНК не может «считывать» целые геномы за один раз, а скорее считывает небольшие фрагменты размером от 20 до 30 000 оснований, в зависимости от используемой технологии. [1] Обычно короткие фрагменты (считывания) получаются в результате дробового секвенирования геномной ДНК или транскриптов генов ( EST ). [1]
Проблему сборки последовательности можно сравнить с тем, как если бы вы взяли много копий книги, пропустили каждую из них через измельчитель с другим резаком и собрали текст книги обратно, просто взглянув на измельченные части. Помимо очевидной сложности этой задачи, есть некоторые дополнительные практические проблемы: в оригинале может быть много повторяющихся абзацев, а некоторые клочки могут быть изменены во время измельчения, чтобы иметь опечатки. Также могут быть добавлены отрывки из другой книги, а некоторые клочки могут быть совершенно неузнаваемыми.
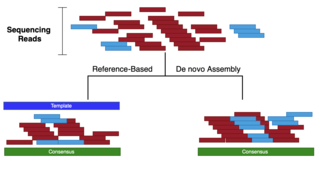
Существует три подхода к сбору данных секвенирования:
Сборка с использованием ссылок представляет собой комбинацию других типов. Этот тип применяется к длинным чтениям для имитации преимуществ коротких чтений (т. е. качества вызова). Логика, лежащая в его основе, заключается в группировке чтений по меньшим окнам в пределах ссылки. Затем чтения в каждой группе будут уменьшены в размере с использованием подхода k-mere для выбора наивысшего качества и наиболее вероятного смежного (contig). Затем контиги будут объединены вместе для создания каркаса. Окончательное согласие достигается путем закрытия любых пробелов в каркасе.
Первые ассемблеры последовательностей начали появляться в конце 1980-х и начале 1990-х годов как варианты более простых программ выравнивания последовательностей для объединения огромных количеств фрагментов, сгенерированных автоматизированными инструментами секвенирования, называемыми ДНК-секвенаторами . [2] По мере того, как секвенируемые организмы росли в размерах и сложности (от небольших вирусов и плазмид до бактерий и, наконец, эукариот ), программы сборки, используемые в этих геномных проектах, нуждались во все более сложных стратегиях для обработки:
Столкнувшись с проблемой сборки первых крупных эукариотических геномов — плодовой мушки Drosophila melanogaster в 2000 году и человеческого генома всего год спустя, — ученые разработали ассемблеры, такие как Celera Assembler [4] и Arachne [5], способные обрабатывать геномы от 130 миллионов (например, плодовая мушка D. melanogaster ) до 3 миллиардов (например, человеческий геном) пар оснований. После этих усилий несколько других групп, в основном в основных центрах секвенирования генома, построили крупномасштабные ассемблеры, и была запущена работа с открытым исходным кодом, известная как AMOS [6], чтобы объединить все инновации в технологии сборки генома в рамках открытой исходной структуры.

Экспрессированная последовательность тега или сборка EST была ранней стратегией, датируемой серединой 1990-х и серединой 2000-х годов, для сборки отдельных генов, а не целых геномов. [7] Проблема отличается от сборки генома несколькими способами. Входные последовательности для сборки EST являются фрагментами транскрибированной мРНК клетки и представляют собой лишь подмножество всего генома. [7] Ряд алгоритмических проблем различаются между сборкой генома и EST. Например, геномы часто имеют большое количество повторяющихся последовательностей, сосредоточенных в межгенных областях. Транскрибированные гены содержат гораздо меньше повторов, что делает сборку несколько проще. С другой стороны, некоторые гены экспрессируются (транскрибируются) в очень больших количествах (например, гены домашнего хозяйства ), что означает, что в отличие от полногеномного дробовика секвенирования, чтения неравномерно выбираются по всему геному.
Сборка EST значительно усложняется такими особенностями, как (цис-) альтернативный сплайсинг , транс-сплайсинг , однонуклеотидный полиморфизм и посттранскрипционная модификация . Начиная с 2008 года, когда был изобретен RNA-Seq , секвенирование EST было заменено этой гораздо более эффективной технологией, описанной в разделе «сборка транскриптома de novo» .
С точки зрения сложности и временных требований сборки de-novo на порядок медленнее и требуют больше памяти, чем сборки карт. Это в основном связано с тем, что алгоритм сборки должен сравнивать каждое чтение с каждым другим чтением (операция, имеющая наивную временную сложность O( n 2 )). Текущие сборщики генома de-novo могут использовать различные типы графовых алгоритмов, такие как: [8]
Ссылаясь на сравнение с измельченными книгами, проведенное во введении: в то время как для картографических сборок в качестве шаблона можно было бы использовать очень похожую книгу (возможно, с измененными именами главных героев и несколькими местами), сборки de-novo представляют собой более сложную задачу, поскольку заранее неизвестно, станет ли это научной книгой, романом, каталогом или даже несколькими книгами. Кроме того, каждый клочок будет сравниваться с каждым другим клочком.
Обработка повторов в сборке de-novo требует построения графа, представляющего соседние повторы. Такая информация может быть получена путем чтения длинного фрагмента, охватывающего повторы полностью или только два его конца . С другой стороны, в сборке отображения части с несколькими совпадениями или без совпадений обычно оставляются для изучения другой техникой сборки. [3]
Сложность сборки последовательностей обусловлена двумя основными факторами: числом фрагментов и их длиной. Хотя большее количество и более длинные фрагменты позволяют лучше идентифицировать перекрытия последовательностей, они также создают проблемы, поскольку базовые алгоритмы демонстрируют квадратичное или даже экспоненциальное поведение сложности как для числа фрагментов, так и для их длины. И хотя более короткие последовательности быстрее выравниваются, они также усложняют фазу компоновки сборки, поскольку более короткие чтения сложнее использовать с повторами или почти идентичными повторами.
На заре секвенирования ДНК ученые могли получить лишь несколько коротких последовательностей (несколько десятков оснований) после недель работы в лабораториях. Следовательно, эти последовательности можно было выровнять вручную за несколько минут.
В 1975 году был изобретен метод дидезокситерминации (также известный как секвенирование по Сэнгеру ), и вскоре после 2000 года технология была усовершенствована до такой степени, что полностью автоматизированные машины могли штамповать последовательности в высокопараллельном режиме 24 часа в сутки. Крупные геномные центры по всему миру размещали полные фермы этих секвенирующих машин, что, в свою очередь, привело к необходимости оптимизации ассемблеров для последовательностей из проектов по дробовому секвенированию всего генома , где считывания
С технологией Сэнгера бактериальные проекты с 20 000–200 000 прочтений можно было легко собрать на одном компьютере. Более крупные проекты, такие как человеческий геном с примерно 35 миллионами прочтений, требовали больших вычислительных ферм и распределенных вычислений.
К 2004/2005 году пиросеквенирование было выведено на коммерческую основу компанией 454 Life Sciences . [9] Этот новый метод секвенирования генерировал считывания, намного более короткие, чем считывания по Сэнгеру: изначально около 100 оснований, теперь 400-500 оснований. [9] Его гораздо более высокая пропускная способность и более низкая стоимость (по сравнению с секвенированием по Сэнгеру) подтолкнули принятие этой технологии геномными центрами, что, в свою очередь, подтолкнуло разработку ассемблеров последовательностей, которые могли бы эффективно обрабатывать наборы считываний. Огромный объем данных в сочетании с технологически специфичными шаблонами ошибок в считываниях задержали разработку ассемблеров; в начале 2004 года был доступен только ассемблер Newbler от 454. Выпущенная в середине 2007 года гибридная версия ассемблера MIRA от Chevreux et al. [10] был первым свободно доступным ассемблером, который мог собирать 454 прочтения, а также смеси 454 прочтений и прочтений Сэнгера. Сборка последовательностей из разных технологий секвенирования впоследствии была названа гибридной сборкой . [10]
С 2006 года доступна технология Illumina (ранее Solexa), которая может генерировать около 100 миллионов прочтений за один запуск на одной машине для секвенирования. Сравните это с 35 миллионами прочтений проекта человеческого генома, на которые потребовалось несколько лет для производства на сотнях машин для секвенирования. [11] Первоначально Illumina была ограничена длиной всего в 36 оснований, что делало ее менее подходящей для сборки de novo (например, сборки транскриптома de novo ), но более новые итерации технологии достигают длины прочтений более 100 оснований с обоих концов клона 3-400 п.н. [11] Анонсированный в конце 2007 года ассемблер SHARCGS [12] Домом и др. был первым опубликованным ассемблером, который использовался для сборки с прочтениями Solexa. За ним быстро последовал ряд других.
Позже были выпущены новые технологии, такие как SOLiD от Applied Biosystems , Ion Torrent и SMRT , и новые технологии (например, секвенирование нанопор ) продолжают появляться. Несмотря на более высокий уровень ошибок этих технологий, они важны для сборки, поскольку их более длинная длина считывания помогает решить проблему повторения. [11] Невозможно собрать через идеальный повтор, который длиннее максимальной длины считывания; однако, по мере того, как считывания становятся длиннее, вероятность идеального повторения такого размера становится малой. Это дает более длинным считываниям секвенирования преимущество в сборке повторов, даже если они имеют низкую точность (~85%). [11]
Большинство сборщиков последовательностей имеют некоторые встроенные алгоритмы для контроля качества, такие как Phred . [13] Однако такие меры не оценивают полноту сборки с точки зрения содержания генов. Некоторые инструменты оценивают качество сборки постфактум.
Например, BUSCO (Benchmarking Universal Single-Copy Orthologs) является мерой полноты генов в геноме, наборе генов или транскриптоме , используя тот факт, что многие гены присутствуют только как гены с одной копией в большинстве геномов. [14] Первоначальные наборы BUSCO представляли 3023 гена для позвоночных , 2675 для членистоногих , 843 для метазоа , 1438 для грибов и 429 для эукариот . В этой таблице показан пример для геномов человека и плодовой мушки: [14]
Различные организмы имеют в своем геноме отдельную область более высокой сложности. Следовательно, необходимы различные вычислительные подходы. Некоторые из наиболее часто используемых алгоритмов:
Учитывая набор фрагментов последовательности, цель состоит в том, чтобы найти более длинную последовательность, содержащую все фрагменты (см. рисунок в разделе « Типы сборки последовательностей» ):
Результат может оказаться не оптимальным решением проблемы.
В целом, сборка результатов секвенирования в каркас состоит из трех этапов:
Список de-novo ассемблеров см. в разделе De novo sequence assemblers . Список картирующих выравнивателей см. в разделе List of sequence alignment software § Short-read sequence alignment .
Некоторые из распространенных инструментов, используемых на различных этапах сборки, перечислены в следующей таблице: