В биологии трансляция — это процесс в живых клетках , в котором белки производятся с использованием молекул РНК в качестве шаблонов. Сгенерированный белок представляет собой последовательность аминокислот . Эта последовательность определяется последовательностью нуклеотидов в РНК. Нуклеотиды считаются тремя за раз. Каждая такая тройка приводит к добавлению одной определенной аминокислоты к генерируемому белку. Соответствие от тройки нуклеотидов к аминокислоте называется генетическим кодом . Трансляция выполняется большим комплексом функциональных РНК и белков, называемых рибосомами . Весь процесс называется экспрессией гена .
При трансляции информационная РНК (мРНК) декодируется в рибосоме, вне ядра, для получения определенной аминокислотной цепи или полипептида . Полипептид позже сворачивается в активный белок и выполняет свои функции в клетке. Рибосома облегчает декодирование, вызывая связывание комплементарных последовательностей антикодонов транспортной РНК (тРНК) с кодонами мРНК . ТРНК переносят определенные аминокислоты, которые объединяются в полипептид, когда мРНК проходит через нее и «считывается» рибосомой.
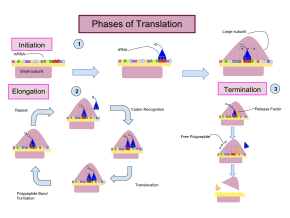
Перевод осуществляется в три этапа:
У прокариот (бактерий и архей) трансляция происходит в цитозоле, где большая и малая субъединицы рибосомы связываются с мРНК. У эукариот трансляция происходит в цитоплазме или через мембрану эндоплазматического ретикулума в процессе, называемом котрансляционной транслокацией . При котрансляционной транслокации весь комплекс рибосома/мРНК связывается с внешней мембраной шероховатого эндоплазматического ретикулума (ЭР), и новый белок синтезируется и высвобождается в ЭР; вновь созданный полипептид может храниться внутри ЭР для будущего транспорта везикул и секреции за пределы клетки или немедленно секретироваться.
Многие типы транскрибируемой РНК, такие как тРНК, рибосомальная РНК и малая ядерная РНК, не транслируются в белки.
Некоторые антибиотики действуют путем ингибирования трансляции. К ним относятся анизомицин , циклогексимид , хлорамфеникол , тетрациклин , стрептомицин , эритромицин и пуромицин . Прокариотические рибосомы имеют структуру, отличную от структуры эукариотических рибосом, и поэтому антибиотики могут специфически воздействовать на бактериальные инфекции без какого-либо вреда для клеток эукариотического хозяина .
Основной процесс производства белка заключается в добавлении одной аминокислоты за раз к концу белка. Эта операция выполняется рибосомой . [ 1] Рибосома состоит из двух субъединиц, малой субъединицы и большой субъединицы. Эти субъединицы объединяются перед трансляцией мРНК в белок, чтобы обеспечить место для осуществления трансляции и производства полипептида. [2] Выбор типа добавляемой аминокислоты определяется молекулой матричной РНК (мРНК). Каждая добавляемая аминокислота сопоставляется с трехнуклеотидной подпоследовательностью мРНК. Для каждого такого возможного триплета принимается соответствующая аминокислота. Последовательные аминокислоты, добавляемые в цепь, сопоставляются с последовательными триплетами нуклеотидов в мРНК. Таким образом, последовательность нуклеотидов в шаблонной цепи мРНК определяет последовательность аминокислот в сгенерированной аминокислотной цепи. [3] Добавление аминокислоты происходит на С-конце пептида; Таким образом, говорят, что трансляция направлена от амина к карбоксилу. [4]
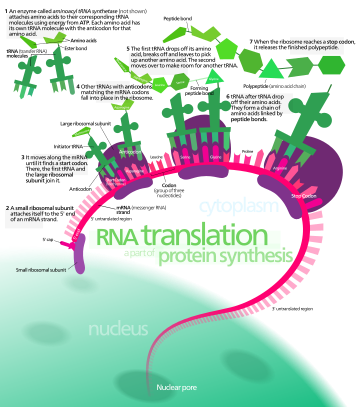
мРНК переносит генетическую информацию, закодированную в виде последовательности рибонуклеотидов от хромосом к рибосомам. Рибонуклеотиды «считываются» трансляционным аппаратом в последовательности триплетов нуклеотидов , называемых кодонами. Каждый из этих триплетов кодирует определенную аминокислоту . [ необходима цитата ]
Молекулы рибосомы транслируют этот код в определенную последовательность аминокислот. Рибосома — это многосубъединичная структура, содержащая рибосомальную РНК (рРНК) и белки. Это «фабрика», где аминокислоты собираются в белки.
Транспортные РНК (тРНК) — это небольшие некодирующие цепи РНК (74–93 нуклеотида), которые транспортируют аминокислоты в рибосому. Репертуар генов тРНК сильно различается между видами: некоторые бактерии имеют от 20 до 30 генов, в то время как сложные эукариоты могут иметь тысячи. [5] тРНК имеют участок для прикрепления аминокислот и участок, называемый антикодоном. Антикодон — это триплет РНК, комплементарный триплету мРНК, который кодирует их грузовую аминокислоту .
Аминоацил-тРНК-синтетазы ( ферменты ) катализируют связывание между определенными тРНК и аминокислотами , которые требуются для их антикодоновых последовательностей. Продуктом этой реакции является аминоацил -тРНК . Аминокислота присоединяется своей карбоксильной группой к 3' ОН тРНК с помощью эфирной связи . Когда с тРНК связана аминокислота, тРНК называется «заряженной». У бактерий эта аминоацил-тРНК переносится в рибосому с помощью EF-Tu , где кодоны мРНК сопоставляются посредством комплементарного спаривания оснований со специфическими антикодонами тРНК. Аминоацил-тРНК-синтетазы, которые неправильно спаривают тРНК с неправильными аминокислотами, могут производить неправильно заряженные аминоацил-тРНК, что может привести к неподходящим аминокислотам в соответствующем положении в белке. Такая «неправильная трансляция» [6] генетического кода естественным образом происходит на низких уровнях у большинства организмов, но определенные клеточные среды вызывают увеличение допустимого декодирования мРНК, иногда на пользу клетке.
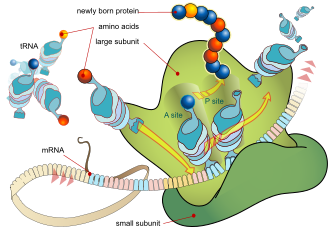
Рибосома имеет два сайта связывания для тРНК. Это аминоацильный сайт (сокращенно A) и пептидильный сайт/сайт выхода (сокращенно P/E). Что касается мРНК, три сайта ориентированы от 5' до 3' EPA, поскольку рибосомы движутся к 3' концу мРНК. A-сайт связывает входящую тРНК с комплементарным кодоном на мРНК. P/E-сайт удерживает тРНК с растущей полипептидной цепью. Когда аминоацил-тРНК изначально связывается с соответствующим ей кодоном на мРНК, она находится на сайте A. Затем образуется пептидная связь между аминокислотой тРНК на сайте A и аминокислотой заряженной тРНК на сайте P/E. Растущая полипептидная цепь переносится на тРНК на сайте A. Происходит транслокация, перемещающая тРНК на сайт P/E, теперь без аминокислоты; тРНК, которая была в сайте А, теперь заряженная полипептидной цепью, перемещается в сайт P/E, а незаряженная тРНК уходит, а другая аминоацил-тРНК входит в сайт А, чтобы повторить процесс. [7]
После добавления новой аминокислоты к цепи и после высвобождения тРНК из рибосомы в цитозоль энергия, вырабатываемая гидролизом ГТФ, связанного с транслоказой EF -G (у бактерий ) и a/eEF-2 (у эукариот и архей ), перемещает рибосому на один кодон вниз к 3'-концу . Энергия, необходимая для трансляции белков, значительна. Для белка, содержащего n аминокислот, количество высокоэнергетических фосфатных связей, необходимых для его трансляции, составляет 4 n -1. [8] Скорость трансляции варьируется; она значительно выше в прокариотических клетках (до 17–21 аминокислотных остатков в секунду), чем в эукариотических клетках (до 6–9 аминокислотных остатков в секунду). [9]
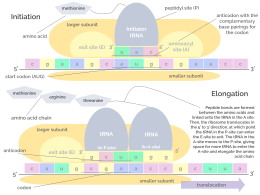
Инициация включает связывание малой субъединицы рибосомы с 5'-концом мРНК с помощью факторов инициации (IF). У бактерий и меньшинства архей инициация синтеза белка включает распознавание богатой пуринами последовательности инициации на мРНК, называемой последовательностью Шайна-Дальгарно . Последовательность Шайна-Дальгарно связывается с комплементарной богатой пиримидином последовательностью на 3'-конце части 16S рРНК 30S рибосомальной субъединицы. Связывание этих комплементарных последовательностей гарантирует, что 30S рибосомальная субъединица связана с мРНК и выровнена таким образом, что кодон инициации находится в 30S части P-сайта. После того, как мРНК и 30S субъединица связаны должным образом, фактор инициации переносит комплекс инициатор тРНК-аминокислота, f-Met -тРНК, в 30S P-сайт. Фаза инициации завершается, когда субъединица 50S присоединяется к субъединице 30S, образуя активную рибосому 70S. [10] Терминация полипептида происходит, когда сайт A рибосомы занят стоп-кодоном (UAA, UAG или UGA) на мРНК, создавая первичную структуру белка. тРНК обычно не может распознавать стоп-кодоны или связываться с ними. Вместо этого стоп-кодон индуцирует связывание белка фактора высвобождения [11] (RF1 и RF2), который вызывает разборку всего комплекса рибосома/мРНК путем гидролиза полипептидной цепи из пептидилтрансферазного центра [1] рибосомы. [12] Лекарственные средства или особые мотивы последовательности на мРНК могут изменять структуру рибосомы таким образом, что почти родственные тРНК связываются со стоп-кодоном вместо факторов высвобождения. В таких случаях «трансляционного считывания» трансляция продолжается до тех пор, пока рибосома не встретит следующий стоп-кодон. [13]
Несмотря на то, что рибосомы обычно считаются точными и процессуальными машинами, процесс трансляции подвержен ошибкам, которые могут привести либо к синтезу ошибочных белков, либо к преждевременному отказу от трансляции, либо потому, что тРНК соединяется с неправильным кодоном, либо потому, что тРНК соединяется с неправильной аминокислотой. [14] Частота ошибок при синтезе белков оценивается в пределах от 1 из 10 5 до 1 из 10 3 неправильно включенных аминокислот, в зависимости от экспериментальных условий. [15] Частота преждевременного отказа от трансляции, напротив, оценивается в величину порядка 10 −4 событий на транслируемый кодон. [16] [17]
Процесс трансляции строго регулируется как в эукариотических, так и в прокариотических организмах. Регулирование трансляции может влиять на глобальную скорость синтеза белка, которая тесно связана с метаболическим и пролиферативным состоянием клетки.
Чтобы глубже изучить этот сложный процесс, ученые обычно используют метод, известный как профилирование рибосом. [18] Этот метод позволяет исследователям сделать снимок транслатома, показывающий, какие части мРНК транслируются в белки рибосомами в данный момент времени. Профилирование рибосом дает ценную информацию о динамике трансляции, раскрывая сложное взаимодействие между последовательностью генов, структурой мРНК и регуляцией трансляции. Например, исследования, использующие этот метод, показали, что генетические различия и их последующее выражение в виде мРНК также могут влиять на скорость трансляции специфичным для РНК образом. [19]
Расширяя эту концепцию, более недавней разработкой является профилирование рибосом отдельных клеток, метод, который позволяет нам изучать процесс трансляции с разрешением отдельных клеток. [20] Это особенно важно, поскольку клетки, даже одного типа, могут демонстрировать значительную изменчивость в синтезе белка. Профилирование рибосом отдельных клеток имеет потенциал пролить свет на гетерогенную природу клеток, что приводит к более тонкому пониманию того, как регуляция трансляции может влиять на поведение клеток, метаболическое состояние и реакцию на различные стимулы или условия.
Трансляционный контроль имеет решающее значение для развития и выживания рака . Раковые клетки должны часто регулировать фазу трансляции экспрессии генов, хотя не до конца понятно, почему трансляция нацелена на такие этапы, как транскрипция. Хотя раковые клетки часто имеют генетически измененные факторы трансляции, гораздо чаще раковые клетки изменяют уровни существующих факторов трансляции. [21] Несколько основных онкогенных сигнальных путей, включая пути RAS–MAPK , PI3K/AKT/mTOR , MYC и WNT–β-катенин , в конечном итоге перепрограммируют геном через трансляцию. [22] Раковые клетки также контролируют трансляцию, чтобы адаптироваться к клеточному стрессу. Во время стресса клетка транслирует мРНК, которые могут смягчить стресс и способствовать выживанию. Примером этого является экспрессия AMPK при различных видах рака; ее активация запускает каскад, который в конечном итоге может позволить раку избежать апоптоза (запрограммированной гибели клеток), вызванного лишением питания. Будущие методы лечения рака могут включать нарушение механизма трансляции клетки, чтобы противостоять последующим эффектам рака. [21]
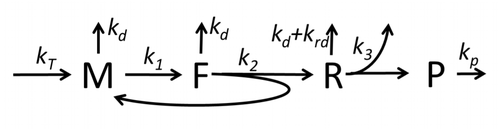
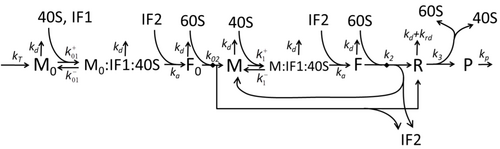
Описание процесса транскрипции-трансляции, в котором упоминаются только самые основные «элементарные» процессы, состоит из:
Процесс построения аминокислот для создания белка при трансляции долгое время является предметом различных физических моделей, начиная с первых подробных кинетических моделей, таких как [24] или других, учитывающих стохастические аспекты трансляции и использующих компьютерное моделирование. За последние четыре десятилетия было разработано и проанализировано множество моделей синтеза белка, основанных на химической кинетике. [25] [26] Помимо химической кинетики, для моделирования подробной кинетики синтеза белка или некоторых его стадий применялись различные формализмы моделирования, такие как полностью асимметричный простой процесс исключения , [26] вероятностные булевы сети , сети Петри и алгебра max-plus . Была разработана базовая модель синтеза белка, которая учитывает все восемь «элементарных» процессов, [23] следуя парадигме , что «полезные модели просты и расширяемы». [27] Простейшая модель M0 представлена кинетическим механизмом реакции (рисунок M0). Он был обобщен, включив связывание 40S, 60S и факторов инициации (IF) (рисунок M1'). Он был расширен далее, включив влияние микроРНК на синтез белка. [28] Большинство моделей в этой иерархии могут быть решены аналитически. Эти решения использовались для извлечения «кинетических сигнатур» различных специфических механизмов регуляции синтеза.
Перевод также возможен вручную (для коротких последовательностей) или с помощью компьютера (после предварительного соответствующего программирования, см. раздел ниже); это позволяет биологам и химикам изобразить химическую структуру закодированного белка на бумаге.
Сначала преобразуйте каждую основу ДНК-матрицы в ее комплемент РНК (обратите внимание, что комплемент A теперь U), как показано ниже. Обратите внимание, что нить-матрица ДНК — это та, против которой полимеризуется РНК; другая нить ДНК будет такой же, как РНК, но с тимином вместо урацила.
ДНК -> РНК А -> У Т -> А С -> Г Г -> С А=Т-> А=У
Затем разделите РНК на триплеты (группы из трех оснований). Обратите внимание, что есть 3 "окна" трансляции или рамки считывания , в зависимости от того, где вы начинаете считывать код. Наконец, используйте таблицу в Genetic code , чтобы перевести вышеизложенное в структурную формулу , используемую в химии.
Это даст первичную структуру белка. Однако белки имеют тенденцию к сворачиванию , что частично зависит от гидрофильных и гидрофобных сегментов вдоль цепи. Вторичную структуру часто все еще можно угадать, но правильную третичную структуру часто очень трудно определить.
В то время как другие аспекты, такие как трехмерная структура белка , называемая третичной структурой , могут быть предсказаны только с использованием сложных алгоритмов , последовательность аминокислот, называемая первичной структурой, может быть определена исключительно из последовательности нуклеиновой кислоты с помощью таблицы трансляции .
Такой подход может не обеспечить правильный аминокислотный состав белка, особенно если в белок включены нетрадиционные аминокислоты , такие как селеноцистеин , который кодируется обычным стоп-кодоном в сочетании с последующей шпилькой (последовательность вставки селеноцистеина, или SECIS).
Существует множество компьютерных программ, способных транслировать последовательность ДНК/РНК в последовательность белка. Обычно это выполняется с использованием стандартного генетического кода, однако немногие программы могут обрабатывать все «особые» случаи, такие как использование альтернативных инициирующих кодонов, которые биологически значимы. Например, редкий альтернативный стартовый кодон CTG кодирует метионин при использовании в качестве стартового кодона и лейцин во всех других позициях.
Пример: Сжатая таблица перевода для стандартного генетического кода (со страницы таксономии NCBI). [29]
AAs = FFLLSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG Начинается = ---M---------------M----------------M----------------------------- База1 = TTTTTTTTTTTTTTTTTTCCCCCCCAAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGGG База2 = TTTTCCCCAAAAGGGGTTTTTCCCCAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG База3 = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG
Строка "Starts" указывает на три стартовых кодона, UUG, CUG и очень распространенный AUG. Она также указывает на первый аминокислотный остаток, если его интерпретировать как старт: в данном случае это все метионин.
Даже при работе с обычными эукариотическими последовательностями, такими как геном дрожжей , часто желательно иметь возможность использовать альтернативные таблицы трансляции, а именно для трансляции митохондриальных генов. В настоящее время следующие таблицы трансляции определены NCBI Taxonomy Group для трансляции последовательностей в GenBank : [29]
{{cite journal}}: CS1 maint: несколько имен: список авторов ( ссылка )