
Рождение генов de novo — это процесс, посредством которого новые гены развиваются из некодирующей ДНК . [1] [3] Гены de novo представляют собой подмножество новых генов и могут кодировать белки или действовать как гены РНК. [4] Процессы, управляющие рождением генов de novo, изучены недостаточно, хотя существует несколько моделей, описывающих возможные механизмы, посредством которых может происходить рождение генов de novo .
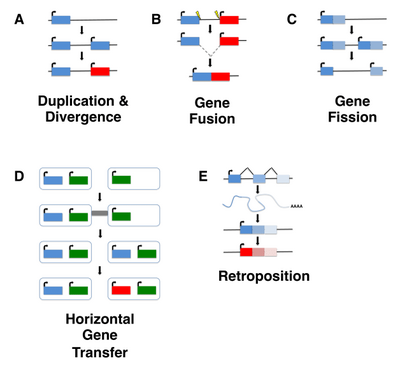
Хотя рождение генов de novo могло произойти в любой момент эволюционной истории организма, древние события рождения генов de novo трудно обнаружить. Большинство исследований генов de novo на сегодняшний день были сосредоточены на молодых генах, как правило, таксономически ограниченных генах (TRG), которые присутствуют в одном виде или линии, включая так называемые гены-сироты , определяемые как гены, которые не имеют какого-либо идентифицируемого гомолога. Однако важно отметить, что не все гены-сироты возникают de novo , а вместо этого могут появляться посредством довольно хорошо охарактеризованных механизмов, таких как дупликация генов (включая ретропозицию) или горизонтальный перенос генов с последующей дивергенцией последовательностей или делением/слиянием генов . [5] [6]
Хотя рождение генов de novo когда-то считалось крайне маловероятным явлением [7] , в настоящее время описано несколько недвусмысленных примеров [8], и некоторые исследователи предполагают, что рождение генов de novo может играть важную роль в эволюционных инновациях, морфологической спецификации и адаптации [9] [10], вероятно, благодаря их низкому уровню плейотропии .
Еще в 1930-х годах Дж. Б. С. Холдейн и другие предположили, что копии существующих генов могут приводить к появлению новых генов с новыми функциями. [6] В 1970 году Сусуму Оно опубликовал основополагающий текст «Эволюция путем дупликации генов» . [11] В течение некоторого времени впоследствии консенсусное мнение заключалось в том, что практически все гены произошли от предковых генов, [12] а Франсуа Жакоб в своем знаменитом эссе 1977 года заметил, что «вероятность того, что функциональный белок появится de novo путем случайной ассоциации аминокислот, практически равна нулю». [7]
Однако в том же году Пьер-Поль Грассе ввел термин « overprinting » для описания появления генов посредством экспрессии альтернативных открытых рамок считывания (ORF), которые перекрывают уже существующие гены. [13] Эти новые ORF могут быть вне рамки или антисмысловыми по отношению к уже существующему гену. Они также могут быть в рамке с существующей ORF, создавая укороченную версию исходного гена, или представлять собой 3'-расширения существующей ORF в соседнюю ORF. Первые два типа overprinting можно рассматривать как особый подтип рождения гена de novo ; хотя и перекрывается с ранее кодирующей областью генома, первичная аминокислотная последовательность нового белка является полностью новой и получена из рамки, которая ранее не содержала ген. Первые примеры этого явления у бактериофагов были описаны в серии исследований с 1976 по 1978 год, [14] [15] [16] и с тех пор было выявлено множество других примеров у вирусов, бактерий и нескольких видов эукариот. [17] [18] [19] [20] [21] [22]
Феномен экзонизации также представляет собой особый случай рождения гена de novo , в котором, например, часто повторяющиеся интронные последовательности приобретают сайты сплайсинга посредством мутации, что приводит к экзонам de novo . Впервые это было описано в 1994 году в контексте последовательностей Alu , обнаруженных в кодирующих областях мРНК приматов. [23] Интересно, что такие экзоны de novo часто встречаются в второстепенных вариантах сплайсинга, что может позволить эволюционное «тестирование» новых последовательностей, сохраняя при этом функциональность основных вариантов сплайсинга. [24]
Тем не менее, некоторые считали, что большинство или все эукариотические белки были построены из ограниченного пула экзонов «стартового типа». [25] Используя данные о последовательностях, доступные в то время, обзор 1991 года оценил количество уникальных, предковых эукариотических экзонов как < 60 000, [25] в то время как в 1992 году была опубликована работа, в которой оценивалось, что подавляющее большинство белков принадлежало не более чем к 1000 семействам. [26] Примерно в то же время, однако, была опубликована последовательность хромосомы III почкующихся дрожжей Saccharomyces cerevisiae , [27] что стало первым случаем, когда была секвенирована целая хромосома любого эукариотического организма. Секвенирование всего ядерного генома дрожжей было завершено к началу 1996 года благодаря масштабным совместным международным усилиям. [28] В своем обзоре проекта генома дрожжей Бернар Дюжон отметил, что неожиданное обилие генов, не имеющих известных гомологов, было, пожалуй, самым поразительным открытием всего проекта. [28]
В 2006 и 2007 годах ряд исследований предоставил, возможно, первые задокументированные примеры рождения генов de novo , которые не включали наложение. [29] [30] [31] Эти исследования проводились с использованием транскриптомов добавочных желез Drosophila yakuba и Drosophila erecta , и они идентифицировали 20 предполагаемых генов, ограниченных линией, которые, по-видимому, вряд ли были результатом дупликации генов. [31] Левин и его коллеги идентифицировали и подтвердили пять генов-кандидатов de novo, специфичных для Drosophila melanogaster и/или близкородственной Drosophila simulans, с помощью строгого подхода, сочетающего биоинформатические и экспериментальные методы. [30]
После этих первоначальных исследований многие группы выявили конкретные случаи событий рождения генов de novo в различных организмах. [32] Первый ген de novo , идентифицированный у дрожжей, ген BSC4, был идентифицирован у S. cerevisiae в 2008 году. Этот ген демонстрирует доказательства очищающего отбора, экспрессируется как на уровне мРНК, так и на уровне белка, и при удалении является синтетически летальным с двумя другими генами дрожжей, все из которых указывают на функциональную роль продукта гена BSC4 . [33] Исторически одним из аргументов против понятия широко распространенного рождения генов de novo является эволюционная сложность сворачивания белка. Интересно, что позже было показано, что Bsc4 принимает частично свернутое состояние, которое сочетает свойства нативного и ненативного сворачивания белка. [34] У растений первым геном de novo , который был функционально охарактеризован, был QQS , ген Arabidopsis thaliana, идентифицированный в 2009 году, который регулирует метаболизм углерода и азота. [35] Первый функционально охарактеризованный ген de novo , идентифицированный у мышей, некодирующий ген РНК, также был описан в 2009 году. [36] У приматов информационный анализ 2008 года подсчитал, что 15/270 генов-сирот приматов были сформированы de novo . [37] В отчете 2009 года были идентифицированы первые три гена de novo человека, один из которых является терапевтической мишенью при хроническом лимфолейкозе. [38] С этого времени множество исследований на уровне генома выявили большое количество генов-сирот во многих организмах, хотя степень, в которой они возникли de novo , и степень, в которой их можно считать функциональными, остаются предметом споров.
Существует два основных подхода к систематической идентификации новых генов: геномная филостратиграфия [39] и методы, основанные на синтении . [40] Оба подхода широко используются по отдельности или в качестве дополнительных.
Геномная филостратиграфия включает в себя изучение каждого гена в фокусном или референтном виде и вывод о наличии или отсутствии предковых гомологов с помощью алгоритмов выравнивания последовательностей BLAST [41] или связанных с ними инструментов. Каждому гену в фокусном виде может быть назначен возраст (он же «уровень сохранения» или «геномный филострат»), который основан на предопределенной филогении, причем возраст соответствует наиболее отдаленному родственному виду, в котором обнаружен гомолог. [39] Когда ген не имеет какого-либо обнаруживаемого гомолога за пределами его собственного генома или близких родственников, говорят, что это новый, таксономически ограниченный или сиротский ген.
Филостратиграфия ограничена набором близкородственных геномов, которые доступны, и результаты зависят от критериев поиска BLAST. [42] Кроме того, часто бывает трудно определить на основе отсутствия наблюдаемого сходства последовательностей, возник ли новый ген de novo или отделился от предкового гена до неузнаваемости, например, после события дупликации. На это указало исследование, в котором моделировалась эволюция генов одинакового возраста и было обнаружено, что далекие ортологи могут быть необнаружимы для быстро эволюционирующих генов. [43] С другой стороны, при учете изменений в скорости эволюции в молодых регионах генов филостратиграфический подход был более точным при назначении возраста генов в моделируемых данных. [44] Последующие исследования с использованием моделируемой эволюции показали, что филостратиграфия не смогла обнаружить ортолога в наиболее отдаленно родственных видах для 13,9% генов D. melanogaster и 11,4% генов S. cerevisiae . [45] [46] Однако повторный анализ исследований, в которых использовалась филостратиграфия на дрожжах, плодовых мушках и людях, показал, что даже при учете таких ошибок и исключении из анализа трудно стратифицируемых генов качественные выводы не изменились. [47] Влияние филостратиграфической ошибки на исследования, изучающие различные особенности генов de novo, остается спорным.
Подходы, основанные на синтении, используют порядок и относительное расположение генов (или других признаков) для идентификации потенциальных предков кандидатов на гены de novo . [10] [42] Синтенические выравнивания закрепляются консервативными «маркерами». Гены являются наиболее распространенным маркером при определении синтенных блоков, хотя также используются k-меры и экзоны. [48] [40] Подтверждение того, что синтенная область не имеет кодирующего потенциала у видов внешней группы, позволяет с большей уверенностью утверждать о происхождении de novo . [42] Самым сильным возможным доказательством возникновения de novo является вывод о конкретной «разрешающей» мутации(ях), которая создала кодирующий потенциал, как правило, посредством анализа более мелких областей последовательности, называемых микросинтеническими областями, у близкородственных видов.
Одной из проблем применения методов, основанных на синтении, является то, что синтению может быть трудно обнаружить в более длительных временных масштабах. Для решения этой проблемы были созданы различные методы оптимизации, такие как использование экзонов, сгруппированных независимо от их конкретного порядка, для определения синтенных блоков [40] или алгоритмов, которые используют хорошо сохранившиеся геномные регионы для расширения микросинтенных блоков. [49] Существуют также трудности, связанные с применением подходов, основанных на синтении, к фрагментированным геномным сборкам [50] или в линиях с высокой скоростью хромосомных перестроек, как это часто бывает у насекомых. [51] Подходы, основанные на синтении, могут применяться к общегеномным исследованиям генов de novo [37] [38] [52] [53] [54] [55] [56] [57] и представляют собой перспективную область разработки алгоритмов для датирования рождения генов. Некоторые использовали подходы, основанные на синтении, в сочетании с поиском сходства в попытке разработать стандартизированные, строгие конвейеры [58] , которые можно было бы применить к любой группе геномов, чтобы попытаться устранить расхождения в различных списках генов de novo , которые были созданы.
Даже когда эволюционное происхождение конкретной кодирующей последовательности установлено, все еще отсутствует консенсус относительно того, что представляет собой подлинное событие рождения гена de novo . Одной из причин этого является отсутствие согласия относительно того, должна ли вся последовательность иметь негенное происхождение. Для генов de novo , кодирующих белок , было предложено разделить гены de novo на подтипы на основе доли рассматриваемой ORF, которая была получена из ранее некодирующей последовательности. [42] Кроме того, для того, чтобы произошло рождение гена de novo , рассматриваемая последовательность должна быть геном, что привело к вопросу о том, что представляет собой ген, при этом некоторые модели устанавливают строгую дихотомию между генными и негенными последовательностями, а другие предлагают более подвижный континуум. [59]
Все определения генов связаны с понятием функции, поскольку общепризнанно, что настоящий ген должен кодировать функциональный продукт, будь то РНК или белок. Однако существуют различные взгляды на то, что составляет функцию, в зависимости от того, оценивается ли данная последовательность с использованием генетических, биохимических или эволюционных подходов. [42] [60] [61] [62] Неоднозначность понятия «функция» особенно проблематична для области рождения генов de novo , где объекты исследования часто быстро развиваются. [62] Чтобы решить эти проблемы, Питтсбургская модель функции деконструирует «функцию» на пять значений, чтобы описать различные свойства, которые приобретает локус, подвергающийся рождению гена de novo : Экспрессия, Возможности, Взаимодействия, Физиологические импликации и Эволюционные импликации. [62]
Общепринято, что подлинный ген de novo экспрессируется по крайней мере в некотором контексте, [5] что позволяет отбору работать, и многие исследования используют доказательства экспрессии в качестве критерия включения при определении генов de novo . Экспрессия последовательностей на уровне мРНК может быть подтверждена индивидуально с помощью таких методов, как количественная ПЦР , или глобально с помощью секвенирования РНК (RNA-seq) . Аналогичным образом, экспрессия на уровне белка может быть определена с высокой достоверностью для отдельных белков с помощью таких методов, как масс-спектрометрия или вестерн-блоттинг , в то время как профилирование рибосом (Ribo-seq) обеспечивает глобальный обзор трансляции в данном образце. В идеале, чтобы подтвердить, что ген возник de novo , также должно быть продемонстрировано отсутствие экспрессии синтенной области видов внешней группы. [63]
Генетические подходы к обнаружению определенного фенотипа или изменения в приспособленности при нарушении определенной последовательности полезны для выведения функции. [61] Другие экспериментальные подходы, включая скрининг белок-белковых и/или генетических взаимодействий, также могут использоваться для подтверждения биологического эффекта для конкретного de novo ORF.
Эволюционные подходы могут быть использованы для вывода о существовании молекулярной функции из вычислительно полученных сигнатур отбора. В случае TRG одной из общих сигнатур отбора является отношение несинонимичных к синонимичным заменам ( отношение dN/dS ), рассчитанное для разных видов из одного и того же таксона. Аналогично, в случае видоспецифичных генов данные полиморфизма могут быть использованы для расчета отношения pN/pS для разных штаммов или популяций фокального вида. Учитывая, что молодые видоспецифичные гены de novo по определению не обладают глубокой консервацией, обнаружение статистически значимых отклонений от 1 может быть затруднено без нереалистично большого количества секвенированных штаммов/популяций. Пример этого можно увидеть в Mus musculus , где три очень молодых гена de novo не имеют сигнатур отбора, несмотря на хорошо продемонстрированные физиологические роли. [64] По этой причине подходы pN/pS часто применяются к группам генов-кандидатов, позволяя исследователям делать вывод, что по крайней мере некоторые из них эволюционно консервативны, не имея возможности указать, какие именно. Вместо этого использовались другие признаки отбора, такие как степень расхождения нуклеотидов в синтенных областях, сохранение границ ORF или для генов, кодирующих белки, оценка кодирования, основанная на частотах гексамеров нуклеотидов. [65] [66]
Оценки частоты и количества генов de novo в различных линиях сильно различаются и сильно зависят от методологии. Исследования могут идентифицировать гены de novo только с помощью методов филостратиграфии/BLAST или могут использовать комбинацию вычислительных методов и могут или не могут оценивать экспериментальные доказательства экспрессии и/или биологической роли. [10] Кроме того, анализы в масштабе генома могут рассматривать все или большинство ORF в геноме, [59] или вместо этого могут ограничивать свой анализ ранее аннотированными генами.
Линия D. melanogaster является иллюстрацией этих различных подходов. Раннее исследование с использованием комбинации поисков BLAST, выполненных на последовательностях кДНК, вместе с ручным поиском и информацией о синтении, выявило 72 новых гена, специфичных для D. melanogaster , и 59 новых генов, специфичных для трех из четырех видов в комплексе видов D. melanogaster . В этом отчете было обнаружено, что только 2/72 (~2,8%) новых генов, специфичных для D. melanogaster , и 7/59 (~11,9%) новых генов, специфичных для комплекса видов, были получены de novo [56] , а остальные возникли путем дупликации/ретропозиции. Аналогичным образом, анализ 195 молодых (<35 миллионов лет) генов D. melanogaster, идентифицированных с помощью синтенных выравниваний, показал, что только 16 возникли de novo . [54] Напротив, анализ, сосредоточенный на транскриптомных данных из семенников шести штаммов D. melanogaster, идентифицировал 106 фиксированных и 142 сегрегирующих гена de novo . [55] Для многих из них были идентифицированы предковые ORF, но не были выражены. Более новое исследование показало, что до 39 % сиротских генов в кладе Drosophila могли возникнуть de novo , поскольку они перекрываются с некодирующими областями генома. [67] Подчеркивая различия между межвидовыми и внутривидовыми сравнениями, исследование в естественных популяциях Saccharomyces paradoxus показало, что количество идентифицированных полипептидов de novo более чем удвоилось при рассмотрении внутривидового разнообразия. [68] У приматов одно раннее исследование идентифицировало 270 сиротских генов (уникальных для людей, шимпанзе и макак), из которых 15, как считалось, возникли de novo . [37] Более поздние отчеты выявили гораздо больше генов de novo только у людей, что подтверждается транскрипционными и протеомными доказательствами. [57] [69] Исследования других линий/организмов также пришли к другим выводам относительно количества генов de novo , присутствующих в каждом организме, а также конкретных наборов идентифицированных генов. Пример этих крупномасштабных исследований описан в таблице ниже.
Вообще говоря, остается спорным, представляют ли дупликация и дивергенция или рождение генов de novo доминирующий механизм появления новых генов, [54] [56] [59] [70] [71] [72] отчасти потому, что гены de novo, вероятно, как появляются, так и теряются чаще, чем другие молодые гены. В исследовании происхождения генов-сирот в трех различных эукариотических линиях авторы обнаружили, что в среднем только около 30% генов-сирот можно объяснить дивергенцией последовательностей. [72]
Важно различать частоту рождения генов de novo и количество генов de novo в данной линии. Если рождение генов de novo происходит часто, можно было бы ожидать, что геномы будут иметь тенденцию к росту содержания генов с течением времени; однако содержание генов в геномах обычно относительно стабильно. [10] Это подразумевает, что частый процесс гибели генов должен уравновешивать рождение генов de novo , и действительно, гены de novo отличаются своей быстрой заменой по сравнению с устоявшимися генами. В поддержку этого представления недавно появившиеся гены Drosophila с гораздо большей вероятностью будут потеряны, в первую очередь через псевдогенизацию , причем самые молодые сироты теряются с самой высокой скоростью; [73] это несмотря на тот факт, что было показано, что некоторые сироты Drosophila быстро становятся необходимыми. [54] Похожая тенденция частой потери среди молодых семейств генов наблюдалась в роде нематод Pristionchus . [74] Аналогичным образом, анализ пяти транскриптомов млекопитающих показал, что большинство ORF у мышей были либо очень старыми, либо видоспецифичными, что подразумевает частое рождение и гибель de novo транскриптов. [71] Сходную тенденцию можно было бы продемонстрировать с помощью дальнейшего анализа шести транскриптомов приматов. [69] В популяциях диких S. paradoxus de novo ORF появляются и исчезают с аналогичной скоростью. [68] Тем не менее, сохраняется положительная корреляция между количеством видоспецифичных генов в геноме и эволюционным расстоянием от его самого недавнего предка. [75] [67] Быстрое появление и исчезновение генов de novo было также обнаружено на популяционном уровне путем анализа девяти естественных популяций трехиглой колюшки. [76] Помимо рождения и гибели генов de novo на уровне ORF, мутационные и другие процессы также подвергают геномы постоянному «транскрипционному обороту». В одном исследовании на мышах было обнаружено, что, хотя все области генома предка были транскрибированы в какой-то момент по крайней мере у одного потомка, часть генома, находящаяся под активной транскрипцией в данном штамме или подвиде, подвержена быстрым изменениям. [77] Транскрипционный оборот некодирующих РНК-генов особенно быстр по сравнению с кодирующими генами. [78]
Недавно возникшие de novo гены отличаются от устоявшихся генов несколькими способами. Сообщалось, что у широкого спектра видов молодые и/или таксономически ограниченные гены короче по длине, чем устоявшиеся гены, более положительно заряжены, быстрее развиваются [88] и менее выражены. [37] [59] [73] [74] [89] [90 ] [91] [92] [93] [94] [95] [96] [71] [69] [67] [76] [ чрезмерное цитирование ] Хотя эти тенденции могут быть результатом смещения обнаружения гомологии, повторный анализ нескольких исследований, которые учитывали это смещение, показал, что полученные качественные выводы не были затронуты. [47] Другая особенность включает тенденцию молодых генов иметь свои гидрофобные аминокислоты, более сгруппированные рядом друг с другом вдоль первичной последовательности. [97] [98]
Экспрессия молодых генов также оказалась более специфичной для тканей или состояний, чем у существующих генов. [29] [31] [ 37] [55] [57] [59] [94] [99] [100] [101] [67] [76] В частности, относительно высокая экспрессия генов de novo наблюдалась в мужских репродуктивных тканях у дрозофилы , колюшки, мышей и людей, а также в человеческом мозге. [57] [102] [67] [76] У животных с адаптивной иммунной системой более высокая экспрессия в мозге и яичках может быть функцией иммунопривилегированной природы этих тканей. Анализ на мышах обнаружил специфическую экспрессию межгенных транскриптов в тимусе и селезенке (в дополнение к мозгу и яичкам). Было высказано предположение, что у позвоночных транскрипты de novo должны сначала экспрессироваться в тканях, лишенных иммунных клеток, прежде чем они смогут экспрессироваться в тканях, имеющих иммунный надзор. [101]
Что касается эволюции последовательностей, исследования анализа dN/dS часто указывают на то, что гены de novo развиваются с более высокой скоростью по сравнению с другими генами. [103] [88] Что касается эволюции экспрессии и структурной эволюции, количественные исследования в различных эволюционных возрастах или филостратиграфических ветвях очень немногочисленны.
Также интересно сравнить особенности недавно возникших de novo генов с пулом негенных ORF, из которых они возникают. Теоретическое моделирование показало, что такие различия являются продуктом как отбора по особенностям, которые увеличивают вероятность функционализации, так и нейтральных эволюционных сил, которые влияют на оборот аллелей. [104] Эксперименты на S. cerevisiae показали, что предсказанные трансмембранные домены были тесно связаны с полезными эффектами приспособленности, когда молодые ORF были сверхэкспрессированы, но не когда установленные (старые) ORF были сверхэкспрессированы. [105] Эксперименты на E. coli показали, что случайные пептиды имели тенденцию оказывать более благоприятные эффекты, когда они были обогащены аминокислотами, которые были маленькими, и это способствовало внутреннему структурному беспорядку. [106]
Особенности генов de novo могут зависеть от вида или изучаемой линии. Это, по-видимому, отчасти является результатом изменения содержания GC в геномах и того, что молодые гены имеют больше сходства с негенными последовательностями из генома, в котором они возникли, чем устоявшиеся гены. [107] Особенности в полученном белке, такие как процент трансмембранных остатков и относительная частота различных предсказанных вторичных структурных особенностей, показывают сильную зависимость GC в генах-сиротах, тогда как в более древних генах эти особенности лишь слабо зависят от содержания GC. [107]
Связь между возрастом гена и количеством прогнозируемого внутреннего структурного расстройства (ISD) в кодируемых белках была предметом значительных дебатов. Утверждалось, что ISD также является признаком, зависящим от линии, примером чего является тот факт, что в организмах с относительно высоким содержанием GC, от D. melanogaster до паразита Leishmania major , молодые гены имеют высокий ISD, [108] [109] тогда как в геноме с низким содержанием GC, таком как почкующиеся дрожжи, несколько исследований показали, что молодые гены имеют низкий ISD. [59] [89] [96] [107] Однако исследование, которое исключило молодые гены с сомнительными доказательствами функциональности, определяемыми в бинарных терминах как находящиеся под отбором для сохранения генов, обнаружило, что оставшиеся молодые гены дрожжей имеют высокий ISD, что позволяет предположить, что результат для дрожжей может быть обусловлен загрязнением набора молодых генов ORF, которые не соответствуют этому определению, и, следовательно, с большей вероятностью будут иметь свойства, отражающие содержание GC и другие негенные особенности генома. [110] Помимо самых молодых сирот, это исследование показало, что ISD имеет тенденцию уменьшаться с увеличением возраста гена, и что это в первую очередь связано с аминокислотным составом, а не с содержанием GC. [110] В более коротких временных масштабах использование генов de novo , которые имеют наибольшую валидацию, позволяет предположить, что молодые гены более неупорядочены у Lachancea , но менее неупорядочены у Saccharomyces . [96] Внутреннее структурное расстройство и склонность к агрегации не показали существенных различий с возрастом в некоторых исследованиях млекопитающих [71] и приматов, [69], но обнаружили в других исследованиях млекопитающих. [110] Одно крупное исследование всей базы данных доменов белка Pfam показало обогащение более молодого домена белка аминокислотами, способствующими расстройству, среди животных, но обогащение на основе доступности аминокислот в растениях. [98]
Исследование генов de novo у A. thaliana показало, что они оба гиперметилированы и в целом лишены модификаций гистонов . [53] В соответствии с моделью протогена или загрязнением не-генами, уровни метилирования генов de novo были промежуточными между установленными генами и межгенными областями. Паттерны метилирования этих генов de novo стабильно наследуются, а уровни метилирования были самыми высокими и наиболее похожими на установленные гены в генах de novo с подтвержденной способностью кодировать белок. [53] У патогенного грибка Magnaporthe oryzae менее консервативные гены, как правило, имеют паттерны метилирования , связанные с низким уровнем транскрипции. [111] Исследование на дрожжах также показало, что гены de novo обогащаются в горячих точках рекомбинации , которые, как правило, являются областями, свободными от нуклеосом. [96]
У Pristionchus pacificus гены-сироты с подтвержденной экспрессией демонстрируют состояния хроматина, которые отличаются от состояний аналогично экспрессируемых установленных генов. [95] Стартовые сайты генов-сирот имеют эпигенетические сигнатуры, характерные для энхансеров, в отличие от консервативных генов, которые демонстрируют классические промоторы. [95] Многие неэкспрессируемые гены-сироты украшены репрессивными модификациями гистонов, в то время как отсутствие таких модификаций облегчает транскрипцию экспрессируемого подмножества генов-сирот, что подтверждает идею о том, что открытый хроматин способствует формированию новых генов. [95]
Белки de novo обычно демонстрируют менее четко определенные вторичные и трехмерные структуры, часто лишенные жесткой укладки, но имеющие обширные неупорядоченные области. [103] [110] Количественный анализ эволюции вторичных структурных элементов и третичных структур с течением времени все еще отсутствует. Поскольку структура обычно более консервативна, чем последовательность, сравнение структур между ортологами может обеспечить более глубокое понимание возникновения и эволюции генов de novo и помочь подтвердить, что эти гены являются истинными генами de novo . [112] Тем не менее, до сих пор только очень немногие белки de novo были структурно и функционально охарактеризованы, особенно из-за проблем с очисткой белков и последующей стабильностью. Прогресс был достигнут с использованием различных меток очистки, типов клеток и шаперонов. [113]
«Антифризный гликопротеин» (AFGP) у арктической трески предотвращает замерзание их крови в арктических водах. [84] [83] Было показано, что Bsc4, короткий несущественный de novo белок у дрожжей, [33] строится в основном из β-слоев и имеет гидрофобное ядро. [34] Он связан с восстановлением ДНК в условиях дефицита питательных веществ. [114] Белок de novo у Drosophila Goddard был впервые охарактеризован в 2017 году. Самцы мух Drosophila melanogaster с нокдауном не могли производить сперму. [80] Недавно удалось показать, что этот недостаток был вызван неудачей индивидуализации удлиненных сперматид. Используя вычислительные филогеномные и структурные прогнозы, экспериментальный структурный анализ и клеточные биологические анализы, было высказано предположение, что половина структуры Годдарда неупорядочена, а другая половина состоит из альфа-спиральных аминокислот. Эти анализы также показали, что ортологи Годдарда показывают схожие результаты. Таким образом, структура Годдарда, по-видимому, в основном сохранилась с момента ее появления. [81]
С развитием таких технологий, как РНК-секвенирование и Рибо-секвенирование, теперь известно, что эукариотические геномы повсеместно транскрибируются [115] [116] [117] [118] и транслируются. [119] Многие ОРС, которые либо не аннотированы, либо аннотированы как длинные некодирующие РНК (lncRNA), транслируются на каком-то уровне, либо в условиях, либо тканеспецифическим образом. [59] [119] [120] [121] [122] [123] Хотя эти события трансляции происходят нечасто, они подвергают негенную последовательность отбору. Эта всеобъемлющая экспрессия составляет основу нескольких моделей, описывающих рождение генов de novo .
Было высказано предположение, что эпигенетический ландшафт генов de novo на ранних стадиях формирования может быть особенно изменчивым между популяциями, что приводит к изменчивой экспрессии генов, тем самым позволяя молодым генам исследовать «ландшафт экспрессии». [124] Ген QQS у A. thaliana является одним из примеров этого явления; его экспрессия отрицательно регулируется метилированием ДНК, которое, хотя и наследуется в течение нескольких поколений, широко варьируется по своим уровням как среди естественных образцов, так и в пределах диких популяций. [124] Эпигенетика также в значительной степени отвечает за пермиссивную транскрипционную среду в семенниках, в частности, посредством включения в нуклеосомы неканонических вариантов гистонов, которые заменяются гистонподобными протаминами во время сперматогенеза. [125]
Анализ разнообразия потенциала сворачивания показывает, что большинство аминокислотных последовательностей, кодируемых межгенными ОРС S. cerevisiae, предположительно, являются складчатыми. [126] Что еще более важно, эти аминокислотные последовательности с потенциалом сворачивания могут служить элементарными строительными блоками для генов de novo или интегрироваться в уже существующие гены. [126]
Для рождения гена, кодирующего белок de novo , негенная последовательность должна быть транскрибирована и приобрести ORF перед тем, как стать транслируемой. Эти события могут происходить в любом порядке, и есть доказательства, подтверждающие как модель «сначала ORF», так и модель «сначала транскрипция». [5] [127] Анализ генов de novo , которые разделяются у D. melanogaster, показал, что последовательности, которые транскрибируются, имеют схожий кодирующий потенциал с ортологичными последовательностями из линий, не имеющих доказательств транскрипции. [55] Это открытие подтверждает идею о том, что многие ORF могут существовать до транскрипции. Ген антифриза гликопротеина AFGP , который появился de novo у арктических тресковых, представляет собой более определенный пример, в котором было показано, что появление ORF de novo предшествует промоторной области. [83] Кроме того, предположительно негенные ORF, достаточно длинные, чтобы кодировать функциональные пептиды, многочисленны в эукариотических геномах и, как ожидается, будут встречаться с высокой частотой случайно. [55] [59] Прослеживая историю эволюции последовательностей ORF и активацию транскрипции генов de novo человека , исследование показало, что некоторые ORF были готовы придать биологическое значение при их рождении. [127] В то же время транскрипция эукариотических геномов гораздо более обширна, чем считалось ранее, и есть задокументированные примеры геномных областей, которые были транскрибированы до появления ORF, ставшей геном de novo . [79] Доля генов de novo , которые кодируют белок, неизвестна, но появление «сначала транскрипция» привело некоторых к предположению, что гены de novo , кодирующие белок , могут сначала существовать как промежуточные звенья генов РНК. Случай бифункциональных РНК, которые и транслируются, и функционируют как гены РНК, показывает, что такой механизм правдоподобен. [128]
Эти два события могут происходить одновременно, когда перестройка хромосом является событием, ускоряющим рождение гена. [129]
Было описано несколько теоретических моделей и возможных механизмов рождения генов de novo . Модели, как правило, не являются взаимоисключающими, и возможно, что множественные механизмы могут привести к появлению генов de novo . [42] Примером является ген белка антифриза типа III, который происходит от старого гена синтазы сиаловой кислоты ( SAS ) у антарктической рыбы зоарцид.
Раннее исследование случая рождения генов de novo , которое идентифицировало пять генов de novo у D. melanogaster , отметило преимущественную экспрессию этих генов в семенниках, [30] и несколько дополнительных генов de novo были идентифицированы с использованием транскриптомных данных, полученных из семенников и мужских добавочных желез D. yakuba и D. erecta . [29] [31] Это согласуется с другими исследованиями, которые показали, что существует быстрая эволюция генов, связанных с воспроизводством, в ряде линий, [130] [131] [132] предполагая, что половой отбор может играть ключевую роль в адаптивной эволюции и рождении генов de novo . Последующий крупномасштабный анализ шести штаммов D. melanogaster идентифицировал 248 генов de novo , экспрессируемых в семенниках , из которых ~57% не были фиксированными. [55] Недавнее исследование двенадцати видов Drosophila дополнительно идентифицировало более высокую долю генов de novo с экспрессией, смещенной в семенники, по сравнению с аннотированным протеомом. [67] Было высказано предположение, что большое количество генов de novo с экспрессией, специфичной для самцов, выявленных у Drosophila, вероятно, связано с тем, что такие гены предпочтительно сохраняются по сравнению с другими генами de novo по причинам, которые не совсем ясны. [73] Интересно, что два предполагаемых гена de novo у Drosophila ( Goddard и Saturn ) оказались необходимыми для нормальной мужской фертильности. [80] [81] Генетический скрининг более 40 предполагаемых генов de novo с экспрессией, обогащенной семенниками, у Drosophila melanogaster показал, что один из генов de novo, atlas , необходим для правильной конденсации хроматина на последних стадиях сперматогенеза у самцов. atlas произошел в результате слияния гена, кодирующего белок, который возник у основания рода Drosophila , и консервативной некодирующей РНК. [133] Сравнительный анализ транскриптомов яичек и придаточных желез, соматической ткани самцов, которая важна для фертильности, D. melanogaster показывает, что гены de novo вносят больший вклад в транскриптомную сложность яичек по сравнению с придаточными железами. [134] Одноклеточное РНК-секвенирование D. melanogasterяички показали, что паттерн экспрессии генов de novo был смещен в сторону раннего сперматогенеза. [135]
У людей исследование, в котором были идентифицированы 60 генов de novo , специфичных для человека , показало, что их средняя экспрессия, измеренная с помощью РНК-секвенирования, была самой высокой в яичках. [57] Другое исследование, изучающее гены, специфичные для млекопитающих, в более общем плане, также обнаружило обогащенную экспрессию в яичках. [136] Транскрипция в яичках млекопитающих считается особенно беспорядочной, отчасти из-за повышенной экспрессии транскрипционного аппарата [137] [138] и открытой хроматиновой среды. [139] Наряду с иммунопривилегированной природой яичек, эта беспорядочная транскрипция, как полагают, создает идеальные условия для экспрессии негенных последовательностей, необходимых для рождения генов de novo . Экспрессия, специфичная для яичек, по-видимому, является общей чертой всех новых генов, поскольку анализ Drosophila и видов позвоночных показал, что молодые гены демонстрировали экспрессию, смещенную в сторону яичек, независимо от механизма их происхождения. [99]
Преадаптационная модель рождения генов de novo использует математическое моделирование, чтобы показать, что когда последовательности, которые обычно скрыты, подвергаются слабому или экранированному отбору, полученный пул «криптических» последовательностей (т. е. протогенов) может быть очищен от «очевидно вредных» вариантов, таких как те, которые склонны приводить к агрегации белков, и, таким образом, обогащен потенциальными адаптациями относительно полностью невыраженного и неочищенного набора последовательностей. [140] Это выявление и очистка криптических вредных негенных последовательностей является побочным продуктом всепроникающей транскрипции и трансляции межгенных последовательностей и, как ожидается, будет способствовать рождению функциональных генов, кодирующих белок de novo . [122] Это происходит потому, что при устранении наиболее вредных вариантов то, что остается, в процессе устранения, с большей вероятностью будет адаптивным, чем ожидается от случайных последовательностей. Используя эволюционное определение функции (то есть, что ген по определению находится под очищающим отбором против потери), модель преадаптации предполагает, что «рождение гена — это внезапный переход к функциональности» [110] , который происходит, как только ORF приобретает чистый полезный эффект. Чтобы избежать вреда, новорожденные гены должны демонстрировать преувеличенные версии генных особенностей, связанных с избеганием вреда. Это контрастирует с моделью протогена, которая ожидает, что новорожденные гены будут иметь особенности, промежуточные между старыми генами и не-генами. [110]
Математика модели преадаптации предполагает, что распределение эффектов приспособленности является бимодальным, с новыми последовательностями мутаций, имеющими тенденцию что-то ломать или ремонтировать, но редко где-то посередине. [140] [141] Следуя этой логике, популяции могут либо развить локальные решения, в которых отбор работает на каждом отдельном локусе и поддерживается относительно высокая частота ошибок, либо глобальное решение с низкой частотой ошибок, которое допускает накопление вредных криптических последовательностей. [140] Считается, что рождение генов de novo благоприятно в популяциях, которые развивают локальные решения, поскольку относительно высокая частота ошибок приведет к пулу криптических вариаций, которые «преадаптированы» посредством очистки вредных последовательностей. Локальные решения более вероятны в популяциях с высоким эффективным размером популяции .
В поддержку модели преадаптации анализ ISD у мышей и дрожжей показал, что молодые гены имеют более высокий ISD, чем старые гены, в то время как случайные негенные последовательности, как правило, показывают самые низкие уровни ISD. [110] Хотя наблюдаемая тенденция могла быть частично результатом подмножества молодых генов, полученных путем оверпринтинга, [142] более высокий ISD в молодых генах также наблюдается среди перекрывающихся пар вирусных генов. [143] Что касается других предсказанных структурных особенностей, таких как содержание β-цепи и склонность к агрегации, пептиды, кодируемые протогенами, похожи на негенные последовательности и категорически отличаются от канонических генов. [144]
Эта протогенная модель согласуется с преадаптационной моделью относительно важности всеобъемлющей экспрессии и относится к набору всеобъемлюще экспрессируемых последовательностей, которые не соответствуют всем определениям гена, как к «протогенам». [59] В отличие от преадаптационной модели, протогенная модель предполагает, что новорожденные гены имеют промежуточные характеристики между старыми генами и негенами. [110] В частности, эта модель предусматривает более постепенный процесс отбора из негенного в генное состояние, отвергая бинарную классификацию гена и негена.
В расширении модели протогена было высказано предположение, что по мере того, как протогены становятся более похожими на гены, их потенциал для адаптивных изменений уступает место выбранным эффектам; таким образом, прогнозируемое влияние мутаций на приспособленность зависит от эволюционного статуса ORF. [105] Это представление подтверждается тем фактом, что сверхэкспрессия устоявшихся ORF в S. cerevisiae имеет тенденцию быть менее полезной (и более вредной), чем сверхэкспрессия появляющихся ORF. [105]
Несколько особенностей ORF коррелируют с возрастом ORF, как определено филостратиграфическим анализом, причем молодые ORF имеют свойства, промежуточные между старыми ORF и не-генами; это было принято как доказательство в пользу модели прото-гена, в которой состояние прото-гена является континуумом. [59] Это доказательство подверглось критике, поскольку те же очевидные тенденции ожидаются и в модели, в которой идентичность как гена является бинарным. В этой модели, когда каждая возрастная группа содержит различное соотношение генов и не-генов, парадокс Симпсона может генерировать корреляции в неправильном направлении. [110]
Модель «расти медленно и линять» описывает потенциальный механизм рождения генов de novo , в частности, генов, кодирующих белки. В этом сценарии существующие ORF, кодирующие белки, расширяются на своих концах, особенно на своих 3'-концах, что приводит к созданию новых N- и C-концевых доменов. [145] [146] [147] [148] [149] Новые C-концевые домены могут сначала развиваться под слабым отбором посредством случайной экспрессии через сквозную трансляцию, как в модели преадаптации, и только позже становятся конститутивно экспрессируемыми через мутацию, которая нарушает стоп-кодон. [140] [146] Гены, испытывающие высокую трансляционную сквозную трансляцию, как правило, имеют внутренне неупорядоченные C-концы. [150] Кроме того, существующие гены часто находятся близко к повторяющимся последовательностям, которые кодируют неупорядоченные домены. Эти новые неупорядоченные домены могут изначально давать некоторую неспецифическую связывающую способность, которая постепенно совершенствуется отбором. Последовательности, кодирующие эти новые домены, могут иногда отделяться от их родительской ORF, приводя или способствуя созданию гена de novo . [146] Интересно, что анализ 32 геномов насекомых показал, что новые домены (т. е. те, которые уникальны для насекомых), как правило, развиваются довольно нейтрально, только несколько участков находятся под положительным отбором, в то время как их белки-хозяева остаются под очищающим отбором, что предполагает, что новые функциональные домены возникают постепенно и несколько стохастически. [151]
Эволюционная модель выхода из адаптивного конфликта (EAC) предлагает возможный способ исправления новой дупликации генов: конфликт из-за контрастной функции в пределах одного гена приводит к фиксации новой дупликации. [152] [153]
Модель «барьера плейотропии» предполагает, что вновь эволюционировавшие гены, включая гены de novo и гены, связанные с дупликацией, могут способствовать эволюционным инновациям или эволюции определенных функций из-за их низкого (или нулевого) плейотропного эффекта при столкновении с новой селективной силой, основываясь на наблюдениях за данными о генетических заболеваниях человека.
Помимо своей значимости для области эволюционной биологии, рождение генов de novo имеет последствия для здоровья человека. Было высказано предположение, что новые гены, включая гены de novo , могут играть огромную роль в видоспецифичных признаках; [6] [10] [32] [154] однако, многие видоспецифичные гены не имеют функциональной аннотации. [136] Тем не менее, есть данные, позволяющие предположить, что специфичные для человека гены de novo участвуют в таких заболеваниях, как рак. NYCM , ген de novo , уникальный для людей и шимпанзе, регулирует патогенез нейробластом в мышиных моделях, [155] а специфичный для приматов PART1 , ген lncRNA, был идентифицирован как супрессор опухолей и онкоген в различных контекстах. [37] [156] [157] Несколько других генов de novo , специфичных для человека или приматов , включая PBOV1 , [158] GR6 , [159] [160] MYEOV , [161] ELFN1-AS1 , [162] и CLLU1 , [38] также связаны с раком. Некоторые даже предложили рассматривать специфически экспрессируемые в опухолях эволюционно новые гены как свой собственный класс генетических элементов, отмечая, что многие такие гены находятся под положительным отбором и могут быть неофункционализированы в контексте опухолей. [162]
Специфическая экспрессия многих генов de novo в человеческом мозге [57] также поднимает интригующую возможность того, что гены de novo влияют на когнитивные черты человека. Одним из таких примеров является FLJ33706 , ген de novo , который был идентифицирован в GWAS и анализах сцепления для никотиновой зависимости и показывает повышенную экспрессию в мозге пациентов с болезнью Альцгеймера. [163] Вообще говоря, экспрессия молодых, специфичных для приматов генов обогащена в плодном человеческом мозге по сравнению с экспрессией аналогичных молодых генов в мозге мыши. [164] Большинство этих молодых генов, некоторые из которых возникли de novo , экспрессируются в неокортексе, который, как считается, отвечает за многие аспекты специфичного для человека познания. Многие из этих молодых генов демонстрируют сигнатуры положительного отбора, а функциональные аннотации указывают на то, что они участвуют в различных молекулярных процессах, но обогащены факторами транскрипции. [164]
Помимо своей роли в процессах рака, человеческие гены, возникшие de novo, были вовлечены в поддержание плюрипотентности [165] и в иммунную функцию. [37] [136] [166] Преимущественная экспрессия генов de novo в яичках также предполагает их роль в воспроизводстве. Учитывая, что функция многих человеческих генов de novo остается неохарактеризованной, кажется вероятным, что понимание их вклада в здоровье и развитие человека будет продолжать расти.
Примечание: Для целей этой таблицы гены определяются как гены-сироты (когда они специфичны для вида) или TRG (когда они ограничены близкородственной группой видов), когда механизм возникновения не был исследован, и как гены de novo , когда было выведено возникновение de novo , независимо от метода вывода. Обозначение генов de novo как «кандидатов» или «протогенов» отражает язык, используемый авторами соответствующих исследований.
![]() Эта статья была адаптирована из следующего источника по лицензии CC BY 4.0 (2019) (отчеты рецензента): Стивен Бранден Ван Осс; Энн-Руксандра Карвунис (23 мая 2019 г.). "De novo gene birth". PLOS Genetics . 15 (5): e1008160. doi : 10.1371/JOURNAL.PGEN.1008160 . ISSN 1553-7390. PMC 6542195. PMID 31120894. Wikidata Q86320144 .
Эта статья была адаптирована из следующего источника по лицензии CC BY 4.0 (2019) (отчеты рецензента): Стивен Бранден Ван Осс; Энн-Руксандра Карвунис (23 мая 2019 г.). "De novo gene birth". PLOS Genetics . 15 (5): e1008160. doi : 10.1371/JOURNAL.PGEN.1008160 . ISSN 1553-7390. PMC 6542195. PMID 31120894. Wikidata Q86320144 .