Молекулярная эволюция описывает, как наследуемые ДНК и/или РНК изменяются в течение эволюционного времени, и последствия этого для белков и других компонентов клеток и организмов . Молекулярная эволюция является основой филогенетических подходов к описанию древа жизни . Молекулярная эволюция пересекается с популяционной генетикой , особенно в более коротких временных масштабах. Темы молекулярной эволюции включают происхождение новых генов, генетическую природу сложных признаков , генетическую основу адаптации и видообразования , эволюцию развития , а также закономерности и процессы, лежащие в основе геномных изменений в ходе эволюции.
История молекулярной эволюции начинается в начале 20-го века со сравнительной биохимии и использования методов «отпечатков пальцев», таких как иммунные анализы, гель-электрофорез и бумажная хроматография в 1950-х годах для изучения гомологичных белков . [1] [2] Появление секвенирования белков позволило молекулярным биологам создавать филогении на основе сравнения последовательностей и использовать различия между гомологичными последовательностями в качестве молекулярных часов для оценки времени с момента появления самого последнего общего предка . [3] [1] Удивительно большое количество молекулярных расхождений внутри видов и между ними вдохновило нейтральную теорию молекулярной эволюции в конце 1960-х годов. [4] [5] [6] Нейтральная теория также предоставила теоретическую основу для молекулярных часов , хотя это не является необходимым для действительности часов. После 1970-х годов секвенирование нуклеиновых кислот позволило молекулярной эволюции выйти за рамки белков и изучить высококонсервативные последовательности рибосомной РНК , что стало основой переосмысления ранней истории жизни . [1] Общество молекулярной биологии и эволюции было основано в 1982 году.
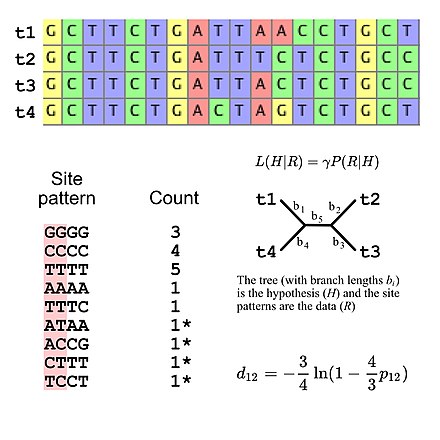
Молекулярная филогенетика использует последовательности ДНК , РНК или белков для решения вопросов в систематике , то есть об их правильной научной классификации с точки зрения эволюционной истории . Результат молекулярного филогенетического анализа выражается в филогенетическом дереве . Филогенетический вывод проводится с использованием данных секвенирования ДНК . Это выравнивается для определения того, какие сайты являются гомологичными . Модель замещения описывает, какие шаблоны, как ожидается, будут общими или редкими. Затем сложный вычислительный вывод используется для создания одного или нескольких правдоподобных деревьев.
Некоторые филогенетические методы учитывают различия между участками и ветвями дерева . Различные гены, например, гемоглобин против цитохрома c , обычно развиваются с разной скоростью . [7] Эти скорости относительно постоянны с течением времени (например, гемоглобин не развивается с той же скоростью, что и цитохром c, но гемоглобины от людей, мышей и т. д. имеют сопоставимые скорости эволюции), хотя быстрая эволюция вдоль одной ветви может указывать на усиление направленного отбора на этой ветви. [8] Очищающий отбор приводит к тому, что функционально важные регионы развиваются медленнее, а замены аминокислот, включающие похожие аминокислоты, происходят чаще, чем разнородные замены. [7]


Дупликация генов может производить несколько гомологичных белков (паралогов) в пределах одного вида. Филогенетический анализ белков показал, как белки развиваются и меняют свою структуру и функцию с течением времени. [9] [10]
Например, рибонуклеотидредуктаза (РНР) развила множество структурных и функциональных вариантов. РНР класса I используют субъединицу ферритина и различаются по металлу, который они используют в качестве кофакторов. В РНР класса II тиильный радикал генерируется с использованием кофактора аденозилкобаламина , и эти ферменты не требуют дополнительных субъединиц (в отличие от класса I, которым это требуется). В РНР класса III тиильный радикал генерируется с использованием S-аденозилметионина, связанного с кластером [ 4Fe-4S ]. То есть в пределах одного семейства белков могут развиваться многочисленные структурные и функциональные механизмы. [11]
В исследовании, подтверждающем концепцию, Бхаттачарья и его коллеги превратили миоглобин , неферментативный белок хранения кислорода, в высокоэффективную элиминазу Кемпа, используя всего три мутации . Это показывает, что для радикального изменения функции белка требуется всего несколько мутаций. [12] Направленная эволюция — это попытка конструировать белки с использованием методов, вдохновленных молекулярной эволюцией.
Изменение в одном локусе начинается с новой мутации , которая может закрепиться благодаря некоторому сочетанию естественного отбора , генетического дрейфа и генной конверсии .

Мутации — это постоянные, передаваемые изменения генетического материала ( ДНК или РНК ) клетки или вируса . Мутации возникают из-за ошибок в репликации ДНК во время деления клеток и под воздействием радиации , химикатов, других факторов окружающей среды, вирусов или мобильных элементов . Когда точечные мутации только одной пары оснований ДНК попадают в область, кодирующую белок , они характеризуются тем, являются ли они синонимичными (не изменяют последовательность аминокислот) или несинонимичными. Другие типы мутаций изменяют более крупные сегменты ДНК и могут вызывать дупликации, вставки, делеции, инверсии и транслокации. [13]
Распределение скоростей для различных видов мутаций называется «спектром мутаций» (см. Приложение B из [14] ). Мутации разных типов происходят с очень разной скоростью. Скорости точечных мутаций для большинства организмов очень низкие, примерно от 10−9 до 10−8 на сайт на поколение, [15] хотя некоторые вирусы имеют более высокие скорости мутаций, порядка 10−6 на сайт на поколение. [16] Транзиции (A ↔ G или C ↔ T) встречаются чаще, чем трансверсии ( пурин (аденин или гуанин)) ↔ пиримидин (цитозин или тимин, или в РНК, урацил)). [17] Возможно, наиболее распространенным типом мутации у людей является изменение длины короткого тандемного повтора (например, повторы CAG, лежащие в основе различных мутаций, связанных с заболеваниями). Такие мутации STR могут происходить со скоростью порядка 10−3 на поколение. [18]
Различные частоты различных типов мутаций могут играть важную роль в эволюции посредством смещения при введении вариации (смещение прибытия), способствуя параллелизму, тенденциям и различиям в навигации адаптивных ландшафтов. [19] [20] Смещение мутаций вносит систематический или предсказуемый вклад в параллельную эволюцию . [14] С 1960-х годов считалось , что геномное содержание GC отражает мутационные тенденции. [21] [22] Мутационные смещения также способствуют смещению использования кодонов . [23] Хотя такие гипотезы часто связаны с нейтральностью, недавние теоретические и эмпирические результаты установили, что мутационные тенденции могут влиять как на нейтральную, так и на адаптивную эволюцию посредством смещения при введении вариации (смещение прибытия).
Отбор может происходить, когда аллель придает большую приспособленность , т. е. большую способность выживать или размножаться, среднему индивидууму, чем его носитель. Селекционистский подход подчеркивает, например, что предубеждения в использовании кодонов обусловлены, по крайней мере, частично, способностью даже слабого отбора формировать молекулярную эволюцию. [24]
Отбор может также работать на уровне генов за счет приспособленности организма, что приводит к внутригеномному конфликту . Это происходит потому, что может быть селективное преимущество для эгоистичных генетических элементов, несмотря на стоимость хозяина. Примерами таких эгоистичных элементов являются транспонируемые элементы , мейотические драйверы и эгоистичные митохондрии .
Отбор можно обнаружить с помощью соотношения Ka/Ks , теста Макдональда-Крейтмана . Быстрая адаптивная эволюция часто обнаруживается для генов, вовлеченных во внутригеномный конфликт , половую антагонистическую коэволюцию и иммунную систему .
Генетический дрейф — это изменение частот аллелей от одного поколения к другому из-за стохастических эффектов случайной выборки в конечных популяциях. Эти эффекты могут накапливаться до тех пор, пока мутация не зафиксируется в популяции . Для нейтральных мутаций скорость фиксации на поколение равна скорости мутации на репликацию. Таким образом, относительно постоянная скорость мутации создает постоянную скорость изменения на поколение (молекулярные часы).
Слабо вредные мутации с коэффициентом отбора меньше порогового значения 1/ эффективный размер популяции также могут быть исправлены. Многие геномные особенности были приписаны накоплению почти нейтральных вредных мутаций в результате малых эффективных размеров популяции. [25] При меньшем эффективном размере популяции большее разнообразие мутаций будет вести себя так, как будто они нейтральны из-за неэффективности отбора.
Конверсия генов происходит во время рекомбинации, когда повреждение нуклеотидов восстанавливается с использованием гомологичной геномной области в качестве шаблона. Это может быть смещенный процесс, то есть один аллель может иметь более высокую вероятность быть донором, чем другой в событии конверсии генов. В частности, конверсия генов со смещением GC имеет тенденцию увеличивать содержание GC в геномах, особенно в областях с более высокими скоростями рекомбинации. [26] Также имеются доказательства смещения GC в процессе репарации несоответствий. [27] Считается, что это может быть адаптацией к высокой скорости дезаминирования метилцитозина, что может привести к переходам C→T.
Динамика смещенной генной конверсии напоминает динамику естественного отбора, в том смысле, что частота предпочтительного аллеля будет экспоненциально увеличиваться, если он редок.
Размер генома зависит от количества повторяющейся ДНК, а также от количества генов в организме. Некоторые организмы, такие как большинство бактерий, Drosophila и Arabidopsis, имеют особенно компактные геномы с небольшим количеством повторяющегося содержимого или некодирующей ДНК. Другие организмы, такие как млекопитающие или кукуруза, имеют большое количество повторяющейся ДНК, длинные интроны и значительные интервалы между генами. Парадокс C-значения относится к отсутствию корреляции между «сложностью» организма и размером генома. Объяснения так называемого парадокса двояки. Во-первых, повторяющиеся генетические элементы могут составлять большие части генома для многих организмов, тем самым раздувая содержание ДНК гаплоидного генома. Повторяющиеся генетические элементы часто происходят от мобильных элементов .
Во-вторых, количество генов не обязательно указывает на количество стадий развития или типов тканей в организме. Организм с небольшим количеством стадий развития или типов тканей может иметь большое количество генов, которые влияют на фенотипы, не связанные с развитием, раздувая содержание генов относительно семейств генов развития.
Нейтральные объяснения размера генома предполагают, что когда размеры популяции малы, многие мутации становятся почти нейтральными. Следовательно, в небольших популяциях повторяющееся содержимое и другая «мусорная» ДНК могут накапливаться, не ставя организм в невыгодное конкурентное положение. Существует мало доказательств, позволяющих предположить, что размер генома находится под сильным широко распространенным отбором у многоклеточных эукариот. Размер генома, независимо от содержания генов, плохо коррелирует с большинством физиологических признаков, и многие эукариоты, включая млекопитающих, несут в себе очень большое количество повторяющейся ДНК.
Однако птицы , вероятно, испытали сильный отбор на уменьшенный размер генома в ответ на изменение энергетических потребностей для полета. Птицы, в отличие от людей, производят ядросодержащие эритроциты, а более крупные ядра приводят к более низким уровням транспорта кислорода. Метаболизм птиц намного выше, чем у млекопитающих, в основном из-за полета, и потребности в кислороде высоки. Следовательно, у большинства птиц небольшие, компактные геномы с небольшим количеством повторяющихся элементов. Косвенные доказательства свидетельствуют о том, что нептичьи тероподовые динозавры, предки современных птиц [28], также имели уменьшенный размер генома, что согласуется с эндотермией и высокими энергетическими потребностями для скорости бега. Многие бактерии также испытали отбор на небольшой размер генома, поскольку время репликации и потребление энергии так тесно коррелируют с приспособленностью.
Муравей Myrmecia pilosula имеет только одну пару хромосом [29] , тогда как папоротник-гадюка Ophioglossum reticulatum имеет до 1260 хромосом. [30] Количество хромосом в геноме организма не обязательно коррелирует с количеством ДНК в его геноме. Общегеномное количество рекомбинации напрямую контролируется количеством хромосом, с одним кроссинговером на хромосому или на плечо хромосомы, в зависимости от вида. [31]
Изменения в числе хромосом могут играть ключевую роль в видообразовании , поскольку разные числа хромосом могут служить барьером для размножения у гибридов. Человеческая хромосома 2 была создана путем слияния двух хромосом шимпанзе и все еще содержит центральные теломеры , а также рудиментарную вторую центромеру . Полиплоидия , особенно аллополиплоидия, которая часто встречается у растений, также может приводить к репродуктивной несовместимости с родительскими видами. Голубые бабочки Agrodiatus имеют различные числа хромосом в диапазоне от n=10 до n=134 и, кроме того, имеют один из самых высоких показателей видообразования, выявленных на сегодняшний день. [32]
Геномы инфузорий содержат каждый ген в отдельных хромосомах.
В дополнение к ядерному геному , эндосимбионтные органеллы содержат свой собственный генетический материал. Митохондриальная и хлоропластная ДНК различается в разных таксонах, но мембраносвязанные белки , особенно компоненты цепи переноса электронов , чаще всего кодируются в органелле. Хлоропласты и митохондрии наследуются по материнской линии у большинства видов, поскольку органеллы должны пройти через яйцо . В редком случае известно, что некоторые виды мидий наследуют митохондрии от отца к сыну.
Новые гены возникают в результате нескольких различных генетических механизмов, включая дупликацию генов , рождение генов de novo , ретротранспозицию , образование химерных генов , включение некодирующей последовательности в существующий ген и усечение гена.
Дупликация генов изначально приводит к избыточности. Однако дублированные последовательности генов могут мутировать, чтобы развить новые функции или специализироваться так, чтобы новый ген выполнял подмножество исходных предковых функций. Ретротранспозиция дублирует гены, копируя мРНК в ДНК и вставляя ее в геном. Ретрогены обычно вставляются в новые геномные области, не имеют интронов и иногда развивают новые паттерны экспрессии и функции.
Химерные гены образуются, когда дупликация, делеция или неполная ретротранспозиция объединяют части двух различных кодирующих последовательностей, чтобы создать новую последовательность гена. Химеры часто вызывают регуляторные изменения и могут перетасовывать домены белков, чтобы создать новые адаптивные функции.
Рождение генов de novo может привести к появлению генов, кодирующих белок, и некодирующих генов из ранее нефункциональной ДНК. [33] Например, Левин и его коллеги сообщили о происхождении пяти новых генов в геноме D. melanogaster . [34] [35] Подобное происхождение генов de novo было также показано у других организмов, таких как дрожжи, [36] рис [37] и люди. [38] Гены de novo могут развиваться из ложных транскриптов, которые уже экспрессируются на низких уровнях. [39]
Конструктивная нейтральная эволюция (CNE) объясняет, что сложные системы могут возникать и распространяться в популяции посредством нейтральных переходов с принципами избыточной емкости, пресупрессии и храпового механизма, [40] [41] [42] и она применялась в различных областях, от происхождения сплайсосомы до сложной взаимозависимости микробных сообществ . [43] [44] [45]
Общество молекулярной биологии и эволюции издает журналы «Molecular Biology and Evolution» и «Genome Biology and Evolution» и проводит ежегодную международную встречу. Другие журналы, посвященные молекулярной эволюции, включают Journal of Molecular Evolution и Molecular Phylogenetics and Evolution . Исследования в области молекулярной эволюции также публикуются в журналах genetics , molecular biology , genomics , systemics и evolutionary biology .
В этой связи неважно, был ли отбор незначительным или самоотменяющимся.
Категория: молекулярная эволюция (Кимура 1968)