Транскриптом — совокупность всех транскриптов РНК , включая кодирующие и некодирующие , в индивидууме или популяции клеток . Этот термин также иногда может использоваться для обозначения всех РНК или только мРНК , в зависимости от конкретного эксперимента. Термин «транскриптом» представляет собой сочетание слов «транскрипт» и «геном» ; это связано с процессом производства транскриптов во время биологического процесса транскрипции .
Ранние этапы аннотаций транскриптома начались с библиотек кДНК , опубликованных в 1980-х годах. Впоследствии появление высокопроизводительных технологий привело к более быстрым и эффективным способам получения данных о транскриптоме. Для изучения транскриптома используются два биологических метода, а именно микрочип ДНК , метод, основанный на гибридизации, и RNA-seq , подход, основанный на последовательностях. [1] РНК-секвенирование является предпочтительным методом и доминирующим методом транскриптомики с 2010-х годов. Транскриптомика отдельных клеток позволяет отслеживать изменения транскриптов с течением времени внутри отдельных клеток.
Данные, полученные из транскриптома, используются в исследованиях, чтобы получить представление о таких процессах, как клеточная дифференциация , канцерогенез , регуляция транскрипции и открытие биомаркеров , среди других. Данные, полученные с помощью транскриптома, также находят применение при установлении филогенетических связей в процессе эволюции и при экстракорпоральном оплодотворении . Транскриптом тесно связан с другими биологическими областями исследований, основанными на -оме ; он дополняет протеом и метаболом и включает транслатом , экзом , мейом и танатотранскриптом , которые можно рассматривать как некоторые поля, изучающие определенные типы транскриптов РНК. Между транскриптомом и другими -омами существуют количественные и консервативные отношения, и данные транскриптомики можно эффективно использовать для прогнозирования других молекулярных видов, таких как метаболиты. [2] Существует множество общедоступных баз данных транскриптомов.
Слово «транскриптом» представляет собой сочетание слов «транскрипт» и «геном» . Он появился вместе с другими неологизмами , образованными с помощью суффиксов -ome и -omics для обозначения всех исследований, проводимых в полногеномном масштабе в области наук о жизни и технологий. Таким образом, транскриптом и транскриптомика были одними из первых слов, появившихся наряду с геномом и протеомом. [3] Первое исследование, представившее случай коллекции библиотеки кДНК мРНК шелкопряда, было опубликовано в 1979 году. [4] Первое плодотворное исследование, в котором упоминался и исследовался транскриптом организма, было опубликовано в 1997 году и описало 60 633 транскрипты экспрессируются в S. cerevisiae с использованием серийного анализа экспрессии генов (SAGE). [5] С появлением высокопроизводительных технологий и биоинформатики и последующим увеличением вычислительной мощности стало все более эффективно и легко характеризовать и анализировать огромные объемы данных. [3] Попытки охарактеризовать транскриптом стали более заметными с появлением автоматизированного секвенирования ДНК в 1980-х годах. [6] В 1990-е годы для идентификации генов и их фрагментов использовалось секвенирование экспрессированных последовательностей . [7] За этим последовали такие методы, как серийный анализ экспрессии генов (SAGE), анализ экспрессии генов (CAGE) и массово-параллельное сигнатурное секвенирование (MPSS).
Транскриптом включает в себя все транскрипты рибонуклеиновой кислоты (РНК), присутствующие в данном организме или экспериментальном образце. [8] РНК является основным носителем генетической информации, которая отвечает за процесс преобразования ДНК в фенотип организма. Ген может дать начало одноцепочечной информационной РНК (мРНК) посредством молекулярного процесса, известного как транскрипция ; эта мРНК комплементарна цепи ДНК, из которой она произошла. [6] Фермент РНК-полимераза II прикрепляется к цепи матрицы ДНК и катализирует добавление рибонуклеотидов к 3'-концу растущей последовательности транскрипта мРНК. [9]
Чтобы инициировать свою функцию, РНК-полимераза II должна распознать последовательность промотора , расположенную выше (5') гена. У эукариот этот процесс опосредован факторами транскрипции , в первую очередь фактором транскрипции II D (TFIID), который распознает ТАТА-бокс и помогает позиционировать РНК-полимеразу в соответствующем стартовом сайте. Чтобы завершить образование транскрипта РНК, обычно происходит терминация на расстоянии нескольких сотен нуклеотидов от терминирующей последовательности и происходит расщепление. [9] Этот процесс происходит в ядре клетки вместе с процессингом РНК , посредством которого молекулы мРНК блокируются , сплайсируются и полиаденилируются для повышения их стабильности перед последующим попаданием в цитоплазму. МРНК дает начало белкам в процессе трансляции , происходящей в рибосомах .
Почти все функциональные транскрипты происходят от известных генов. Единственным исключением является небольшое количество транскриптов, которые могут играть непосредственную роль в регуляции экспрессии генов вблизи суфлеров известных генов. (См. Энхансерная РНК .)
Гены занимают большую часть геномов прокариот, поэтому большая часть их геномов транскрибируется. Геномы многих эукариот очень велики, и известные гены могут занимать лишь часть генома. Например, у млекопитающих известные гены составляют лишь 40–50% генома. [10] Тем не менее, идентифицированные транскрипты часто соответствуют гораздо большей части генома, что позволяет предположить, что транскриптом содержит ложные транскрипты, которые не происходят от генов. Известно, что некоторые из этих транскриптов нефункциональны, поскольку они картируются с транскрибируемыми псевдогенами или дегенеративными транспозонами и вирусами. Другие соответствуют неопознанным участкам генома, которые могут быть мусорной ДНК.
Ложная транскрипция очень распространена у эукариот, особенно у тех, у кого большие геномы, которые могут содержать много мусорной ДНК . [11] [12] [13] [14] Некоторые учёные утверждают, что если транскрипт не был приписан известному гену, то по умолчанию должно быть предположение, что это мусорная РНК, пока не будет доказано, что она функциональна. [11] [15] Это будет означать, что большая часть транскриптома у видов с большими геномами, вероятно, представляет собой мусорную РНК. (См. Некодирующая РНК )
Транскриптом включает транскрипты генов, кодирующих белок (мРНК плюс интроны), а также транскрипты некодирующих генов (функциональные РНК плюс интроны).
В геноме человека все гены транскрибируются в РНК, потому что именно так определяется молекулярный ген. (См. Ген .) Транскриптом состоит из кодирующих областей мРНК плюс некодирующие НТО, интроны, некодирующие РНК и ложные нефункциональные транскрипты.
Несколько факторов затрудняют определение содержания транскриптома. К ним относятся, среди прочего, альтернативный сплайсинг , редактирование РНК и альтернативная транскрипция. [17] Кроме того, методы транскриптома способны фиксировать транскрипцию, происходящую в образце в определенный момент времени, хотя содержание транскриптома может меняться во время дифференцировки. [6] Основными целями транскриптомики являются следующие: «каталогизировать все виды транскриптов, включая мРНК, некодирующие РНК и малые РНК; определить транскрипционную структуру генов с точки зрения их стартовых сайтов, 5' и 3'. концов, паттернов сплайсинга и других посттранскрипционных модификаций, а также количественно оценить изменяющиеся уровни экспрессии каждого транскрипта во время развития и в различных условиях». [1]
Этот термин может применяться к общему набору транскриптов в данном организме или к определенному подмножеству транскриптов, присутствующих в определенном типе клеток. В отличие от генома , который примерно фиксирован для данной клеточной линии (без учета мутаций ), транскриптом может меняться в зависимости от внешних условий среды. Поскольку он включает в себя все транскрипты мРНК в клетке, транскриптом отражает гены , которые активно экспрессируются в любой момент времени, за исключением явлений деградации мРНК, таких как ослабление транскрипции . Исследование транскриптомики (которое включает в себя профилирование экспрессии , анализ вариантов сплайсинга и т. д.) изучает уровень экспрессии РНК в данной клеточной популяции, часто сосредотачиваясь на мРНК, но иногда включая и другие, такие как тРНК и мРНК.
Транскриптомика — это количественная наука, которая включает в себя присвоение списка строк («чтений») объекту («транскриптам» в геноме). Для расчета силы экспрессии подсчитывается плотность прочтений, соответствующих каждому объекту. [18] Первоначально транскриптомы анализировались и изучались с использованием библиотек экспрессируемых последовательностей меток , а также серийного и кэп-анализа экспрессии генов (SAGE).
В настоящее время два основных метода транскриптомики включают микрочипы ДНК и RNA-Seq . Оба метода требуют выделения РНК с помощью методов экстракции РНК с последующим ее отделением от других клеточных компонентов и обогащением мРНК. [19] [20]
Существует два основных метода определения последовательностей транскриптома. Один подход сопоставляет считывание последовательности с эталонным геномом либо самого организма (транскриптом которого изучается), либо близкородственного вида. Другой подход, сборка транскриптома de novo , использует программное обеспечение для вывода транскриптов непосредственно из считывания коротких последовательностей и используется в организмах с геномами, которые не секвенированы. [21]

Первые исследования транскриптома были основаны на методах микрочипов (также известных как ДНК-чипы). Микрочипы состоят из тонких стеклянных слоев с пятнами, на которых расположены олигонуклеотиды , известные как «зонды»; каждое пятно содержит известную последовательность ДНК. [22]
При проведении микрочипового анализа мРНК собирают из контрольного и экспериментального образца, последний обычно представляет заболевание. Интересующую РНК преобразуют в кДНК для повышения ее стабильности и маркируют флуорофорами двух цветов, обычно зеленого и красного, для двух групп. кДНК наносится на поверхность микрочипа, где она гибридизуется с олигонуклеотидами на чипе, а для сканирования используется лазер. Интенсивность флуоресценции в каждом пятне микрочипа соответствует уровню экспрессии генов, и по цвету выбранных флуорофоров можно определить, какой из образцов демонстрирует более высокие уровни интересующей мРНК. [7]
Один микрочип обычно содержит достаточно олигонуклеотидов, чтобы представить все известные гены; однако данные, полученные с помощью микрочипов, не дают информации о неизвестных генах. В 2010-е годы микрочипы были почти полностью заменены методами нового поколения, основанными на секвенировании ДНК.
Секвенирование РНК — это технология секвенирования нового поколения ; как таковой для него требуется лишь небольшое количество РНК и никаких предварительных знаний о геноме. [3] Он позволяет проводить как качественный, так и количественный анализ транскриптов РНК, первый позволяет обнаруживать новые транскрипты, а второй позволяет измерить относительное количество транскриптов в образце. [16]
Три основных этапа секвенирования транскриптомов любых биологических образцов включают очистку РНК, синтез библиотеки РНК или кДНК и секвенирование библиотеки. [16] Процесс очистки РНК различен для коротких и длинных РНК. [16] За этим этапом обычно следует оценка качества РНК, чтобы избежать таких загрязнений, как ДНК, или технических загрязнений, связанных с обработкой образцов. Качество РНК измеряют с помощью УФ-спектрометрии с пиком поглощения 260 нм. [23] Целостность РНК также можно проанализировать количественно, сравнивая соотношение и интенсивность 28S РНК и 18S РНК , указанные в показателе целостности РНК (RIN). [23] Поскольку мРНК представляет собой вид, представляющий интерес, и она составляет только 3% от ее общего содержания, образец РНК следует обработать для удаления рРНК и тРНК, а также тканеспецифичных транскриптов РНК. [23]
Этап подготовки библиотеки с целью получения коротких фрагментов кДНК начинается с фрагментации РНК до транскриптов длиной от 50 до 300 пар оснований . Фрагментация может быть ферментативной ( эндонуклеазы РНК ), химической (буфер на основе трисмагниевой соли, химический гидролиз ) или механической ( обработка ультразвуком , распыление). [24] Обратная транскрипция используется для преобразования РНК-матриц в кДНК, и для этого можно использовать три метода прайминга, включая олиго-DT, использование случайных праймеров или лигирование специальных адаптерных олигонуклеотидов.
Транскрипцию также можно изучать на уровне отдельных клеток с помощью одноклеточной транскриптомики . Секвенирование одноклеточной РНК (scRNA-seq) — это недавно разработанный метод, позволяющий анализировать транскриптом отдельных клеток, включая бактерии . [25] При транскриптомике отдельных клеток также принимаются во внимание субпопуляции типов клеток, составляющих интересующую ткань. [26] Этот подход позволяет определить, связаны ли изменения в экспериментальных образцах с фенотипическими клеточными изменениями, а не с пролиферацией, при которой определенный тип клеток может сверхэкспрессироваться в образце. [27] Кроме того, при оценке прогрессирования клеток посредством дифференцировки средние профили экспрессии способны упорядочить клетки только по времени, а не по стадии их развития, и, следовательно, не могут показать тенденции в уровнях экспрессии генов, специфичные для определенных стадий. [28] Транскриптомные методы единичных клеток использовались для характеристики популяций редких клеток, таких как циркулирующие опухолевые клетки , раковые стволовые клетки в солидных опухолях и эмбриональные стволовые клетки (ЭСК) в бластоцистах млекопитающих . [29]
Хотя не существует стандартизированных методов транскриптомики отдельных клеток, необходимо предпринять несколько шагов. Первый шаг включает изоляцию клеток, которую можно выполнить с использованием методов с низкой и высокой пропускной способностью. За этим следует этап qPCR, а затем одноклеточная RNAseq, где интересующая РНК преобразуется в кДНК. Новые разработки в области транскриптомики отдельных клеток позволяют сохранять тканевую и субклеточную локализацию посредством криодеструкции тонких срезов тканей и секвенирования транскриптома в каждом срезе. Другой метод позволяет визуализировать отдельные транскрипты под микроскопом, сохраняя при этом пространственную информацию каждой отдельной клетки, в которой они экспрессируются. [29]
Был создан и аннотирован ряд баз данных транскриптомов, специфичных для организма, чтобы помочь в идентификации генов, которые дифференциально экспрессируются в различных популяциях клеток.
РНК-секвенирование становится (2013 г.) методом выбора для измерения транскриптомов организмов, хотя более старый метод микрочипов ДНК все еще используется. [1] RNA-seq измеряет транскрипцию конкретного гена путем преобразования длинных РНК в библиотеку фрагментов кДНК . Затем фрагменты кДНК секвенируются с использованием технологии высокопроизводительного секвенирования и выравниваются с эталонным геномом или транскриптомом, который затем используется для создания профиля экспрессии генов. [1]
Транскриптомы стволовых и раковых клеток представляют особый интерес для исследователей, стремящихся понять процессы клеточной дифференцировки и канцерогенеза . Конвейер, использующий данные РНК-секвенирования или массива генов, может использоваться для отслеживания генетических изменений, происходящих в стволовых клетках и клетках- предшественниках , и требует как минимум трех независимых данных об экспрессии генов из первого типа клеток и зрелых клеток. [30]
Анализ транскриптомов ооцитов и эмбрионов человека используется для понимания молекулярных механизмов и сигнальных путей, контролирующих раннее эмбриональное развитие, и теоретически может стать мощным инструментом для правильного отбора эмбрионов при экстракорпоральном оплодотворении . [ нужна ссылка ] Анализ содержания транскриптома плаценты в первом триместре беременности при экстракорпоральном оплодотворении и переносе эмбрионов (IVT-ET) выявил различия в генетической экспрессии, которые связаны с более высокой частотой неблагоприятных перинатальных исходов. Такое понимание можно использовать для оптимизации практики. [31] Транскриптомный анализ также можно использовать для оптимизации криоконсервации ооцитов за счет снижения травм, связанных с этим процессом. [32]
Транскриптомика является новой и постоянно развивающейся областью поиска биомаркеров для использования при оценке безопасности лекарств или оценке химического риска . [33]
Транскриптомы также можно использовать для вывода о филогенетических отношениях между людьми или для обнаружения эволюционных закономерностей сохранения транскриптома. [34]
Анализ транскриптома использовался для выявления частоты антисмысловой транскрипции, их роли в экспрессии генов посредством взаимодействия с окружающими генами и их распространенности в различных хромосомах. [35] RNA-seq также использовался, чтобы показать, как изоформы РНК, транскрипты, происходящие от одного и того же гена, но с разными структурами, могут производить сложные фенотипы из ограниченных геномов. [21]
Транскриптомный анализ использовался для изучения процесса эволюции и разнообразия видов растений. В 2014 году был завершен проект «1000 геномов растений» , в рамках которого были секвенированы транскриптомы 1124 видов растений из семейств viridiplantae , glaucophyta и rhodophyta . В дальнейшем кодирующие белки последовательности сравнивали, чтобы сделать вывод о филогенетических отношениях между растениями и охарактеризовать время их диверсификации в процессе эволюции. [36] Исследования транскриптома использовались для характеристики и количественной оценки экспрессии генов в зрелой пыльце . Было обнаружено, что гены, участвующие в метаболизме клеточной стенки и цитоскелете, сверхэкспрессируются. Транскриптомные подходы также позволили отслеживать изменения в экспрессии генов на разных стадиях развития пыльцы, от микроспор до зрелых пыльцевых зерен; кроме того, такие стадии можно было бы сравнить у разных видов растений, включая арабидопсис , рис и табак . [37]
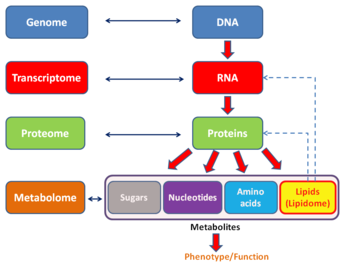
Подобно другим технологиям, основанным на -оме , анализ транскриптома позволяет использовать объективный подход при экспериментальной проверке гипотез. Этот подход также позволяет открыть новые медиаторы в сигнальных путях. [18] Как и в случае с другими технологиями, основанными на -омике, транскриптом можно анализировать в рамках подхода мультиомики . Он дополняет метаболомику , но, в отличие от протеомики, не может установить прямую связь между транскриптом и метаболитом .
Существует несколько полей -ome, которые можно рассматривать как подкатегории транскриптома. Экзом отличается от транскриптома тем, что он включает только те молекулы РНК, обнаруженные в определенной популяции клеток, и обычно включает количество или концентрацию каждой молекулы РНК в дополнение к молекулярным особенностям. Кроме того, транскритпома также отличается от транслатома , который представляет собой набор РНК, подвергающихся трансляции.
Термин мейом используется в функциональной геномике для описания мейотического транскриптома или набора транскриптов РНК, образующихся в процессе мейоза . [38] Мейоз является ключевой особенностью эукариот , размножающихся половым путем , и включает в себя спаривание гомологичных хромосом , синапсов и рекомбинацию. Поскольку мейоз у большинства организмов происходит в течение короткого периода времени, профилирование мейотических транскриптов затруднено из-за проблемы выделения (или обогащения) мейотических клеток ( мейоцитов ). Как и в случае с анализом транскриптома, мейом можно изучать на уровне всего генома с использованием крупномасштабных транскриптомных методов. [39] Мейом хорошо охарактеризован у млекопитающих и дрожжевых систем и несколько менее подробно охарактеризован у растений. [40]
Танатотранскриптом состоит из всех транскриптов РНК, которые продолжают экспрессироваться или начинают реэкспрессироваться во внутренних органах мертвого тела через 24–48 часов после смерти . Некоторые гены включают те, которые ингибируются после развития плода . Если танатотранскриптом связан с процессом запрограммированной гибели клеток ( апоптоз ), его можно назвать апоптотическим танатотранскриптомом. Анализы танатотранскриптома используются в судебной медицине . [41]
Картирование eQTL можно использовать для дополнения геномики транскриптомикой; генетические варианты на уровне ДНК и меры экспрессии генов на уровне РНК. [42]
Транскриптом можно рассматривать как подмножество протеома , то есть весь набор белков, экспрессируемых геномом.
Однако анализ относительных уровней экспрессии мРНК может быть осложнен тем фактом, что относительно небольшие изменения экспрессии мРНК могут вызывать большие изменения общего количества соответствующего белка, присутствующего в клетке. Один из методов анализа, известный как анализ обогащения набора генов , идентифицирует корегулируемые генные сети, а не отдельные гены, активность которых повышена или понижена в различных клеточных популяциях. [1]
Хотя исследования на микрочипах могут выявить относительное количество различных мРНК в клетке, уровни мРНК не прямо пропорциональны уровню экспрессии белков, которые они кодируют. [43] Количество белковых молекул, синтезируемых с использованием данной молекулы мРНК в качестве матрицы, сильно зависит от особенностей инициации трансляции последовательности мРНК; в частности, способность последовательности инициации трансляции является ключевым фактором, определяющим рекрутирование рибосом для трансляции белка .