
РНК -вирус — это вирус, характеризующийся геномом на основе рибонуклеиновой кислоты ( РНК ) . [1] Геном может быть одноцепочечным РНК ( ssRNA ) или двухцепочечным (dsRNA). [2] Известные заболевания человека, вызываемые РНК-вирусами, включают грипп , атипичную пневмонию ( SARS ) , ближневосточный респираторный синдром (MERS) , COVID-19 , вирус денге , гепатит C , гепатит E , лихорадку Западного Нила , болезнь, вызванную вирусом Эбола , бешенство , полиомиелит , эпидемический паротит и корь .
Все известные РНК-вирусы, то есть вирусы, которые используют гомологичную РНК-зависимую полимеразу для репликации, классифицируются Международным комитетом по таксономии вирусов (ICTV) в царство Riboviria . [3] Сюда входят РНК-вирусы, принадлежащие к Группе III , Группе IV или Группе V Балтиморской системы классификации , а также Группе VI. Вирусы Группы VI — это ретровирусы , вирусы с генетическим материалом РНК, которые используют промежуточные продукты ДНК в своем жизненном цикле , включая ВИЧ-1 и ВИЧ-2 , которые вызывают СПИД .
Большинство таких РНК-вирусов попадают в царство Orthornavirae , а остальные имеют еще не определенное позиционирование . [4] Царство не содержит все РНК-вирусы: Deltavirus , Avsunviroidae и Pospiviroidae — это таксоны РНК-вирусов, которые были ошибочно включены в 2019 году, [a] , но исправлены в 2020 году. [5]
РНК-вирусы могут быть далее классифицированы в соответствии со смыслом или полярностью их РНК на вирусы с отрицательным смыслом и с положительным смыслом , или амбисенс-РНК. Вирусная РНК с положительным смыслом похожа на мРНК и, таким образом, может быть немедленно транслирована клеткой-хозяином. Вирусная РНК с отрицательным смыслом комплементарна мРНК и, таким образом, должна быть преобразована в РНК с положительным смыслом с помощью РНК-зависимой РНК-полимеразы перед трансляцией. Очищенная РНК вируса с положительным смыслом может напрямую вызывать инфекцию, хотя она может быть менее заразной, чем целая вирусная частица. Напротив, очищенная РНК вируса с отрицательным смыслом сама по себе не заразна, так как ее необходимо транскрибировать в РНК с положительным смыслом; каждый вирион может быть транскрибирован в несколько РНК с положительным смыслом. Вирусы амбисенс -РНК напоминают вирусы с отрицательным смыслом РНК, за исключением того, что они транслируют гены со своих отрицательных и положительных цепей. [6]
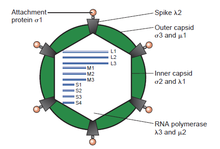
Вирусы с двухцепочечной (ds)РНК представляют собой разнообразную группу вирусов, которые сильно различаются по диапазону хозяев (люди, животные, растения, грибы , [b] и бактерии ), количеству сегментов генома (от одного до двенадцати) и организации вириона ( число триангуляции , слои капсида , шипы, башни и т. д.). Члены этой группы включают ротавирусы , которые являются наиболее распространенной причиной гастроэнтерита у маленьких детей, и пикобирнавирусы , которые являются наиболее распространенным вирусом в образцах фекалий как людей, так и животных с признаками диареи или без них. Вирус синего языка является экономически важным патогеном, который заражает крупный рогатый скот и овец. В последние годы был достигнут прогресс в определении структур с атомным и субнанометровым разрешением ряда ключевых вирусных белков и капсидов вирионов нескольких вирусов с двухцепочечной РНК, что подчеркивает существенные параллели в структуре и репликативных процессах многих из этих вирусов. [2] [ нужна страница ]
РНК-вирусы, как правило, имеют очень высокую скорость мутаций по сравнению с ДНК-вирусами , [8] потому что вирусные РНК-полимеразы не обладают способностью корректировать ДНК-полимеразы . [9] Генетическое разнообразие РНК-вирусов является одной из причин, по которой трудно создавать эффективные вакцины против них. [10] Ретровирусы также имеют высокую скорость мутаций, даже несмотря на то, что их промежуточная ДНК интегрируется в геном хозяина (и, таким образом, подвергается корректированию ДНК хозяина после интеграции), потому что ошибки во время обратной транскрипции встраиваются в обе цепи ДНК до интеграции. [11] Некоторые гены РНК-вируса важны для циклов репликации вируса, и мутации не допускаются. Например, область генома вируса гепатита С , которая кодирует основной белок, является высококонсервативной , [12] потому что она содержит структуру РНК, вовлеченную во внутренний сайт входа рибосомы . [13]
В среднем вирусы dsRNA показывают меньшую избыточность последовательностей по сравнению с вирусами ssRNA. Напротив, вирусы dsDNA содержат самые избыточные последовательности генома, тогда как вирусы ssDNA имеют наименьшее количество. [14] Было показано, что сложность последовательностей вирусов является ключевой характеристикой для точной безреферентной вирусной классификации. [14]
РНК-вирусы животных классифицируются ICTV. Существует три различных группы РНК-вирусов в зависимости от их генома и способа репликации:
Ретровирусы (группа VI) имеют одноцепочечный РНК-геном, но, в целом, не считаются РНК-вирусами, поскольку они используют промежуточные продукты ДНК для репликации. Обратная транскриптаза , вирусный фермент, который выделяется самим вирусом после того, как он становится непокрытым, преобразует вирусную РНК в комплементарную цепь ДНК, которая копируется для получения двухцепочечной молекулы вирусной ДНК. После того, как эта ДНК интегрируется в геном хозяина с помощью вирусного фермента интегразы , экспрессия закодированных генов может привести к образованию новых вирионов.
Многочисленные РНК-вирусы способны к генетической рекомбинации , когда в одной и той же клетке-хозяине присутствуют по крайней мере два вирусных генома . [15] Очень редко вирусная РНК может рекомбинировать с РНК-хозяином. [16] Рекомбинация РНК, по-видимому, является основной движущей силой в определении архитектуры генома и хода вирусной эволюции среди Picornaviridae ( (+)ssRNA ), например, полиовируса . [17] У Retroviridae ((+)ssRNA), например, ВИЧ , повреждения в геноме РНК, по-видимому, избегаются во время обратной транскрипции путем переключения цепей, формы рекомбинации. [18] [19] [20] Рекомбинация также происходит у Reoviridae (dsRNA), например, реовируса; Orthomyxoviridae ((-)ssRNA), например, вируса гриппа ; [20] и Coronaviridae ((+)ssRNA), например, SARS . [21] Рекомбинация в РНК-вирусах, по-видимому, является адаптацией для преодоления повреждения генома. [15] Рекомбинация может происходить нечасто между вирусами животных одного вида, но разных линий. Полученные рекомбинантные вирусы могут иногда вызывать вспышку инфекции у людей. [21]
Классификация основана в основном на типе генома (двуцепочечный, отрицательно- или положительно-одноцепочечный), а также на количестве и организации генов. В настоящее время известно 5 отрядов и 47 семейств РНК-вирусов. Существует также множество нераспределенных видов и родов.
Связанные с РНК-вирусами, но отличающиеся от них, — это вироиды и РНК-сателлитные вирусы . В настоящее время они не классифицируются как РНК-вирусы и описаны на их собственных страницах.
Исследование нескольких тысяч РНК-вирусов показало наличие по крайней мере пяти основных таксонов: группа левивирусов и их родственников; супергруппа пикорнавирусов; супергруппа альфавирусов плюс супергруппа флавивирусов; вирусы dsRNA; и вирусы с -ve-цепью. [22] Группа лентивирусов, по-видимому, является базовой для всех остальных РНК-вирусов. Следующее крупное подразделение лежит между пикорнасупрагруппой и остальными вирусами. Вирусы dsRNA, по-видимому, произошли от предка с +ve РНК, а вирусы РНК -ve - от вирусов dsRNA. Наиболее близким родственником вирусов с -ve-цепью РНК является Reoviridae .
Это самая большая группа РНК-вирусов [23] , которая была организована ICTV в типы Kitrinoviricota , Lenarviricota и Pisuviricota в царстве Orthornavirae и области Riboviria . [24]
Вирусы с положительной цепью РНК также можно классифицировать на основе РНК-зависимой РНК-полимеразы. Были признаны три группы: [25]
Было предложено подразделение альфа-подобной (Синдбис-подобной) супергруппы на основе нового домена, расположенного вблизи N-концов белков, участвующих в репликации вируса. [26] Предложены две группы: группа «альтовирусов» (альфавирусы, фуровирусы, вирус гепатита Е, гордеивирусы, тобамовирусы, тобравирусы, трикорнавирусы и, возможно, рубивирусы); и группа «типовирусов» (вирус хлоротической пятнистости листьев яблони, карлавирусы, потексвирусы и тимовирусы).
Супергруппу альфа-подобных вирусов можно разделить на три клады : рубиподобные, тобамоподобные и тимоподобные вирусы. [27]
Дополнительные исследования выявили пять групп вирусов с положительной цепью РНК, содержащих четыре, три, три, три и один порядок(а) соответственно. [28] Эти четырнадцать порядков содержат 31 семейство вирусов (включая 17 семейств вирусов растений) и 48 родов (включая 30 родов вирусов растений). Этот анализ предполагает, что альфавирусы и флавивирусы можно разделить на два семейства — Togaviridae и Flaviridae соответственно, — но предполагает, что другие таксономические отнесения, такие как пестивирусы, вирус гепатита С, рубивирусы, вирус гепатита Е и артеривирусы, могут быть неверными. Коронавирусы и торовирусы, по-видимому, являются отдельными семействами в отдельных порядках, а не отдельными родами одного и того же семейства, как классифицируется в настоящее время. Лютеовирусы, по-видимому, являются двумя семействами, а не одним, а вирус хлоротической пятнистости листьев яблони, по-видимому, является не клостеровирусом, а новым родом Potexviridae.
Эволюция пикорнавирусов, основанная на анализе их РНК-полимераз и геликаз , по-видимому, восходит к расхождению эукариот . [29] Их предполагаемые предки включают бактериальные ретроэлементы группы II , семейство протеаз HtrA и ДНК- бактериофаги .
Партитивирусы связаны с предком тотивирусом и, возможно, произошли от него. [30]
Гиповирусы и барнавирусы, по-видимому, имеют общее происхождение с линиями потивирусов и собемовирусов соответственно. [30]
Этот анализ также предполагает, что вирусы dsRNA не являются тесно связанными друг с другом, а вместо этого принадлежат к четырем дополнительным классам — Birnaviridae, Cystoviridae, Partitiviridae и Reoviridae — и одному дополнительному отряду (Totiviridae) одного из классов вирусов с положительной одноцепочечной РНК в том же подтипе, что и вирусы с положительной цепью РНК.
В одном исследовании было высказано предположение, что существует две большие клады: одна включает семейства Caliciviridae , Flaviviridae и Picornaviridae , а вторая включает семейства Alphatetraviridae , Birnaviridae , Cystoviridae , Nodaviridae и Permutotretraviridae . [31]
Эти вирусы имеют несколько типов генома, от одной молекулы РНК до восьми сегментов. Несмотря на их разнообразие, кажется, что они могли произойти от членистоногих и диверсифицироваться оттуда. [32]
Также известно несколько вирусов-сателлитов — вирусов, которым для завершения жизненного цикла требуется помощь другого вируса. Их таксономия еще не определена. Для вирусов-сателлитов с положительной полярностью одноцепочечной РНК, которые заражают растения, были предложены следующие четыре рода — Albetovirus , Aumaivirus , Papanivirus и Virtovirus . [33] Для вирусов-сателлитов с положительной полярностью одноцепочечной РНК, которые заражают членистоногих , было предложено семейство — Sarthroviridae , которое включает род Macronovirus .
В эту группу входят двенадцать семейств и ряд нераспределенных родов и видов. [9]
В этой группе выделяют три отряда и 34 семейства. Кроме того, имеется ряд неклассифицированных видов и родов.
Вирусы-сателлиты
Также был описан неклассифицированный вирус, подобный астровирусу/гепевирусу. [35]
За исключением вируса гепатита D , эта группа вирусов была помещена в один тип — Negarnaviricota . Этот тип был разделен на два подтипа — Haploviricotina и Polyploviricotina . Внутри подтипа Haploviricotina в настоящее время признаются четыре класса: Chunqiuviricetes , Milneviricetes , Monjiviricetes и Yunchangviricetes . В подтипе Polyploviricotina признаются два класса: Ellioviricetes и Insthoviricetes .
В настоящее время в этой группе признано шесть классов, семь порядков и двадцать четыре семейства. Ряд нераспределенных видов и родов еще предстоит классифицировать. [9]