
Секвенирование экзома , также известное как секвенирование всего экзома ( WES ), представляет собой геномный метод секвенирования всех кодирующих белки областей генов в геноме (известных как экзом ). [1] Он состоит из двух этапов: первый этап заключается в выборе только подмножества ДНК , кодирующего белки . Эти регионы известны как экзоны — у людей около 180 000 экзонов, что составляет около 1% человеческого генома , или приблизительно 30 миллионов пар оснований . Второй этап заключается в секвенировании экзонной ДНК с использованием любой высокопроизводительной технологии секвенирования ДНК . [2]
Целью этого подхода является выявление генетических вариантов, которые изменяют последовательности белков, и сделать это с гораздо меньшими затратами, чем секвенирование всего генома . Поскольку эти варианты могут быть ответственны как за менделевские , так и за распространенные полигенные заболевания, такие как болезнь Альцгеймера , секвенирование всего экзома применяется как в академических исследованиях, так и в качестве клинической диагностики.

Секвенирование экзома особенно эффективно при изучении редких менделевских заболеваний, поскольку это эффективный способ идентификации генетических вариантов во всех генах индивидуума. Чаще всего эти заболевания вызываются очень редкими генетическими вариантами, которые присутствуют только у небольшого числа индивидуумов; [3] в отличие от этого, такие методы, как массивы SNP, могут обнаружить только общие генетические варианты, которые являются общими для многих индивидуумов в более широкой популяции. [4] Кроме того, поскольку варианты, вызывающие серьезные заболевания, с гораздо большей вероятностью (но ни в коем случае не исключительно) находятся в последовательности, кодирующей белок, [5] [6] сосредоточение на этом 1% стоит гораздо меньше, чем секвенирование всего генома , но все равно обнаруживает высокий выход соответствующих вариантов.
В прошлом клинические генетические тесты выбирались на основе клинической картины пациента (т. е. фокусировались на одном гене или небольшом количестве, известных как связанные с определенным синдромом), или исследовали только определенные типы вариаций (например, сравнительная геномная гибридизация ), но обеспечивали окончательную генетический диагноз менее чем у половины всех пациентов. [7] Экзомное секвенирование в настоящее время все чаще используется для дополнения этих других тестов: как для поиска мутаций в генах, которые уже известны как вызывающие заболевание, так и для идентификации новых генов путем сравнения экзомов пациентов со схожими признаками. [ необходима цитата ]
Методы целевого обогащения позволяют выборочно захватывать интересующие геномные регионы из образца ДНК перед секвенированием. Несколько стратегий целевого обогащения были разработаны с момента первоначального описания метода прямого геномного отбора (DGS) в 2005 году. [8]
Хотя было описано много методов целевого захвата, только некоторые из них были расширены для захвата целых экзомов. [9] Первой стратегией обогащения мишени, примененной к секвенированию целых экзомов, был метод гибридного захвата на основе массива в 2007 году, но захват в растворе приобрел популярность в последние годы.
Микрочипы содержат одноцепочечные олигонуклеотиды с последовательностями из человеческого генома для укладки интересующей области, закрепленной на поверхности. Геномная ДНК разрезается для образования двухцепочечных фрагментов. Фрагменты подвергаются репарации концов для получения тупых концов, и добавляются адаптеры с универсальными праймирующими последовательностями. Эти фрагменты гибридизируются с олигонуклеотидами на микрочипе. Негибридизованные фрагменты смываются, а желаемые фрагменты элюируются. Затем фрагменты амплифицируются с помощью ПЦР . [10] [11]
Компания Roche NimbleGen первой взяла оригинальную технологию DGS [8] и адаптировала ее для секвенирования следующего поколения. Они разработали Sequence Capture Human Exome 2.1M Array для захвата ~180 000 кодирующих экзонов. [12] Этот метод экономит время и является экономически эффективным по сравнению с методами на основе ПЦР. Agilent Capture Array и сравнительный геномный гибридизационный массив — это другие методы, которые можно использовать для гибридного захвата целевых последовательностей. Ограничения этого метода включают необходимость в дорогостоящем оборудовании, а также относительно большом количестве ДНК. [13]
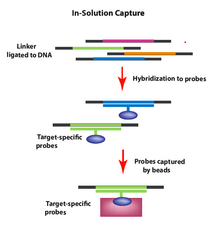
Для захвата интересующих геномных областей с помощью захвата в растворе синтезируется пул пользовательских олигонуклеотидов (зондов), которые гибридизуются в растворе с фрагментированным образцом геномной ДНК. Зонды (помеченные бусинами) селективно гибридизуются с интересующими геномными областями, после чего бусины (теперь включающие интересующие фрагменты ДНК) можно вытянуть и промыть, чтобы очистить от излишков материала. Затем бусины удаляются, а геномные фрагменты можно секвенировать, что позволяет проводить селективное секвенирование ДНК интересующих геномных областей (например, экзонов).
Этот метод был разработан для улучшения метода гибридизации захвата целевого обогащения. При захвате раствора (в отличие от гибридного захвата) существует избыток зондов для целевых областей интереса по сравнению с требуемым количеством шаблона. [13] Оптимальный размер цели составляет около 3,5 мегабаз и обеспечивает превосходное покрытие последовательностей целевых областей. Предпочтительный метод зависит от нескольких факторов, включая: количество пар оснований в интересующей области, требования к считываниям на цели, оборудование в доме и т. д. [14]
Существует множество платформ для секвенирования Next Generation Sequencing , которые появились после классических методологий секвенирования Сэнгера. Другие платформы включают секвенатор Roche 454 и системы Life Technologies SOLiD, Life Technologies Ion Torrent и Illumina Genome Analyzer II (несуществующий) и последующие серии инструментов Illumina MiSeq, HiSeq и NovaSeq, все из которых могут использоваться для массового параллельного секвенирования экзома. Эти системы NGS с «коротким считыванием» особенно хорошо подходят для анализа многих относительно коротких участков последовательности ДНК, как это обнаружено в экзонах человека.
Существует множество технологий, которые идентифицируют генетические варианты. Каждая технология имеет свои преимущества и недостатки с точки зрения технических и финансовых факторов. Две такие технологии — это микрочипы и секвенирование всего генома .
Микрочипы используют гибридизационные зонды для проверки распространенности известных последовательностей ДНК, поэтому их нельзя использовать для выявления неожиданных генетических изменений. [13] Напротив, высокопроизводительные технологии секвенирования, используемые при секвенировании экзома, напрямую предоставляют нуклеотидные последовательности ДНК в тысячах протестированных экзонных локусов. [15] Таким образом, WES устраняет некоторые из существующих ограничений гибридизационных генотипических массивов.
Хотя экзомное секвенирование обходится дороже, чем технологии, основанные на гибридизации, в расчете на один образец, его стоимость снижается из-за снижения стоимости и увеличения производительности полного геномного секвенирования . [ необходима ссылка ]
Секвенирование экзома способно идентифицировать только те варианты, которые находятся в кодирующей области генов, которые влияют на функцию белка. Оно не способно идентифицировать структурные и некодирующие варианты, связанные с заболеванием, которые можно найти с помощью других методов, таких как секвенирование всего генома . [2] Остается 99% генома человека, которые не охвачены секвенированием экзома, а секвенирование экзома позволяет секвенировать части генома по меньшей мере в 20 раз большем количестве образцов по сравнению с секвенированием всего генома. [2] Для перевода выявленных редких вариантов в клинику размер выборки и способность интерпретировать результаты для предоставления клинического диагноза указывают на то, что с текущими знаниями в области генетики есть сообщения об использовании секвенирования экзома для помощи в диагностике. [12] Стоимость секвенирования экзома, как правило, ниже, чем секвенирования всего генома. [16]
Статистический анализ большого количества данных, полученных с помощью подходов секвенирования, является сложной задачей. Даже при секвенировании только экзомов отдельных лиц генерируется большое количество данных и информации о последовательностях, что требует значительного объема анализа данных. Проблемы, связанные с анализом этих данных, включают изменения в программах, используемых для выравнивания и сборки прочтений последовательностей. [13] Различные технологии секвенирования также имеют разные показатели ошибок и генерируют разные длины прочтений, что может создавать проблемы при сравнении результатов с разных платформ секвенирования.
Ложноположительные и ложноотрицательные результаты связаны с подходами геномного ресеквенирования и являются критическими проблемами. Было разработано несколько стратегий для улучшения качества экзомных данных, таких как:
Редкие рецессивные расстройства могут не иметь однонуклеотидных полиморфизмов (SNP) в общедоступных базах данных, таких как dbSNP . Более распространенные рецессивные фенотипы с большей вероятностью будут иметь варианты, вызывающие заболевание, указанные в dbSNP. Например, наиболее распространенный вариант муковисцидоза имеет частоту аллелей около 3% в большинстве популяций. Отсеивание таких вариантов может ошибочно исключить такие гены из рассмотрения. Гены для рецессивных расстройств обычно легче идентифицировать, чем для доминантных расстройств, поскольку гены с меньшей вероятностью имеют более одного редкого несинонимичного варианта. [2] Система, которая отслеживает распространенные генетические варианты, опирается на dbSNP, который может не иметь точной информации об изменчивости аллелей. Использование списков распространенных вариаций из исследуемого экзома или человека, секвенированного по всему геному, было бы более надежным. Проблема в этом подходе заключается в том, что по мере увеличения числа секвенированных экзомов dbSNP также будет увеличивать количество необычных вариантов. Необходимо будет разработать пороговые значения для определения распространенных вариантов, которые вряд ли будут связаны с фенотипом заболевания. [15]
Генетическая гетерогенность и этническая принадлежность населения также являются серьезными ограничениями, поскольку они могут увеличить количество ложноположительных и ложноотрицательных результатов, что затруднит идентификацию генов-кандидатов. Конечно, можно снизить строгость порогов при наличии гетерогенности и этнической принадлежности, однако это также снизит способность обнаружения вариантов. Использование подхода, ориентированного на генотип , для идентификации генов-кандидатов также может предложить решение для преодоления этих ограничений.
В отличие от анализа распространенных вариантов, анализ редких вариантов в исследованиях по секвенированию всего экзома оценивает наборы вариантов, а не отдельные варианты. [17] [18] Функциональные аннотации предсказывают эффект или функцию редких вариантов и помогают расставить приоритеты среди редких функциональных вариантов. Включение этих аннотаций может эффективно повысить мощность генетического анализа ассоциации редких вариантов в исследованиях по секвенированию всего генома. [19] Были разработаны некоторые методы и инструменты для выполнения функционально-информированного анализа ассоциации редких вариантов путем включения функциональных аннотаций для расширения возможностей анализа в исследованиях по секвенированию всего экзома. [20] [21]
Новые технологии в геномике изменили подход исследователей как к фундаментальным, так и к трансляционным исследованиям. С такими подходами, как секвенирование экзома, можно значительно улучшить данные, полученные из индивидуальных геномов, что выдвинуло ряд вопросов о том, как обращаться с огромным объемом информации. Должны ли люди в этих исследованиях иметь доступ к своей информации о секвенировании? Должны ли эти данные быть предоставлены страховым компаниям? Эти данные могут привести к неожиданным результатам и усложнить клиническую полезность и пользу для пациентов. Эта область геномики по-прежнему остается сложной задачей, и исследователи ищут способы решения этих вопросов. [15]
Используя секвенирование экзома, исследования с фиксированной стоимостью могут секвенировать образцы на гораздо большую глубину, чем это можно было бы достичь с помощью секвенирования всего генома. Эта дополнительная глубина делает секвенирование экзома хорошо подходящим для нескольких приложений, которым требуются надежные варианты вызовов.
Текущие исследования ассоциаций сосредоточены на общих вариациях по всему геному, поскольку их легче всего идентифицировать с помощью наших текущих анализов. Однако было обнаружено, что болезнетворные варианты с большим эффектом лежат в экзомах в исследованиях генов-кандидатов, и из-за отрицательного отбора обнаруживаются в гораздо более низких частотах аллелей и могут оставаться нетипированными в текущих стандартных анализах генотипирования. Полногеномное секвенирование является потенциальным методом анализа новых вариантов по всему геному. Однако при сложных расстройствах (таких как аутизм) считается, что большое количество генов связано с риском заболевания. [1] [22] Эта гетерогенность основного риска означает, что для обнаружения генов требуются очень большие размеры выборки, и, таким образом, полногеномное секвенирование не особенно экономически эффективно. Эта проблема размера выборки смягчается разработкой новых передовых аналитических методов, которые эффективно картируют гены болезней, несмотря на то, что генетические мутации редки на уровне вариантов. [22] Кроме того, варианты в кодирующих областях были изучены гораздо более подробно, и их функциональные последствия гораздо легче вывести, что делает практическое применение вариантов в целевом регионе экзома более доступным.
Секвенирование экзома в редких вариантах генов остается очень активной и продолжающейся областью исследований, и появляется все больше доказательств того, что значительное бремя риска наблюдается по всем наборам генов. Секвенирование экзома было зарегистрировано для редких вариантов в гене KRT82 при аутоиммунном заболевании Alopecia Areata. [1]
При менделевских расстройствах большого эффекта полученные на сегодняшний день данные свидетельствуют о том, что в основе всего состояния лежит один или очень небольшое количество вариантов в кодирующих генах. Из-за тяжести этих расстройств предполагается, что несколько причинных вариантов чрезвычайно редки или новы в популяции и будут пропущены любым стандартным анализом генотипирования. Экзомное секвенирование обеспечивает высокий охват вариантов вызовов по кодирующим областям, что необходимо для отделения истинных вариантов от шума. Успешная модель открытия менделевских генов включает открытие вариантов de novo с использованием трио-секвенирования, где генотипируются родители и пробанд.
В исследовании, опубликованном в сентябре 2009 года, обсуждался эксперимент по проверке концепции, чтобы определить, возможно ли идентифицировать причинные генетические варианты с помощью экзомного секвенирования. Они секвенировали четырех человек с синдромом Фримена–Шелдона (FSS) (OMIM 193700), редким аутосомно-доминантным заболеванием, которое, как известно, вызывается мутацией в гене MYH3 . [2] Восемь человек HapMap также были секвенированы для удаления распространенных вариантов с целью идентификации причинного гена для FSS. После исключения распространенных вариантов авторам удалось идентифицировать MYH3 , что подтверждает, что экзомное секвенирование может использоваться для идентификации причинных вариантов редких заболеваний. [2] Это было первое опубликованное исследование, в котором экзомное секвенирование использовалось в качестве подхода к идентификации неизвестного причинного гена для редкого менделевского заболевания.
Впоследствии другая группа сообщила об успешной клинической диагностике предполагаемого пациента с синдромом Барттера турецкого происхождения. [12] Синдром Барттера — это заболевание почек, приводящее к потере соли. Секвенирование экзома выявило неожиданную хорошо сохранившуюся рецессивную мутацию в гене SLC26A3 , которая связана с врожденной хлоридной диареей (CLD). Этот молекулярный диагноз CLD был подтвержден направляющим клиницистом. Этот пример предоставил доказательство концепции использования секвенирования всего экзома в качестве клинического инструмента для оценки пациентов с недиагностированными генетическими заболеваниями. Этот отчет рассматривается как первое применение технологии секвенирования следующего поколения для молекулярной диагностики пациента.
Второй отчет был проведен по секвенированию экзома людей с менделевским расстройством, известным как синдром Миллера (MIM#263750), редким расстройством аутосомно-рецессивного наследования. Были изучены два брата и два неродственных человека с синдромом Миллера. Они рассмотрели варианты, которые могут быть патогенными, такие как несинонимичные мутации, акцепторные и донорные сайты сплайсинга и короткие кодирующие вставки или делеции. [3] Поскольку синдром Миллера является редким расстройством, ожидается, что причинный вариант ранее не был идентифицирован. Предыдущие исследования секвенирования экзома распространенных однонуклеотидных полиморфизмов (SNP) в публичных базах данных SNP использовались для дальнейшего исключения генов-кандидатов. После исключения этих генов авторы обнаружили мутации в DHODH , которые были общими для людей с синдромом Миллера. Каждый человек с синдромом Миллера был сложной гетерозиготой по мутациям DHODH , которые были унаследованы, поскольку каждый родитель пораженного человека был обнаружен как носитель. [3]
Это был первый случай, когда было показано, что секвенирование экзома идентифицирует новый ген, ответственный за редкое менделевское заболевание. Это захватывающее открытие демонстрирует, что секвенирование экзома имеет потенциал для обнаружения причинных генов в сложных заболеваниях, что ранее было невозможно из-за ограничений традиционных методов. Целевой захват и массовое параллельное секвенирование представляют собой экономически эффективную, воспроизводимую и надежную стратегию с высокой чувствительностью и специфичностью для обнаружения вариантов, вызывающих изменения в кодировании белков в отдельных геномах человека.
Секвенирование экзома может использоваться для диагностики генетической причины заболевания у пациента. Определение мутации(й) гена основного заболевания может иметь важные последствия для диагностических и терапевтических подходов, может служить руководством для прогнозирования естественного течения заболевания и позволяет тестировать членов семьи из группы риска. [2] [3] [12] [23] [24] [25] Существует много факторов, которые делают секвенирование экзома более эффективным, чем анализ одного гена, включая возможность идентифицировать мутации в генах, которые не были протестированы из-за нетипичной клинической картины [25] или возможность идентифицировать клинические случаи, когда мутации из разных генов способствуют разным фенотипам у одного и того же пациента. [3]
Диагностировав генетическую причину заболевания, эта информация может помочь в выборе соответствующего лечения. Впервые эта стратегия была успешно применена в клинике при лечении младенца с воспалительным заболеванием кишечника. [24] [26] Ранее использовался ряд традиционных диагностических методов, но результаты не могли объяснить симптомы младенца. Анализ данных секвенирования экзома выявил мутацию в гене XIAP . Знание функции этого гена помогло в лечении младенца, что привело к трансплантации костного мозга, которая излечила ребенка от болезни. [24]
Исследователи использовали секвенирование экзома для определения базовой мутации у пациента с синдромом Барттера и врожденной хлоридной диареей. [12] Группа Билгулара также использовала секвенирование экзома и определила базовую мутацию у пациента с тяжелыми пороками развития мозга, заявив: «[Эти результаты] подчеркивают использование секвенирования всего экзома для определения локусов заболевания в условиях, в которых традиционные методы оказались сложными... Наши результаты показывают, что эта технология будет особенно ценной для открытия генов в тех условиях, в которых картирование было затруднено гетерогенностью локуса и неопределенностью границ диагностической классификации, что указывает на светлое будущее для ее широкого применения в медицине». [23]
Исследователи из Университета Кейптауна (Южная Африка) использовали экзомное секвенирование для обнаружения генетической мутации CDH2 как основной причины генетического заболевания, известного как аритмогенная кардиомиопатия правого желудочка (АКПЖ), которая увеличивает риск сердечных заболеваний и остановки сердца. [1]
Несколько компаний предложили потребителям услуги по секвенированию экзома. Knome была первой компанией, предложившей потребителям услуги по секвенированию экзома [ когда? ] по цене в несколько тысяч долларов. [27] Позже 23andMe запустила пилотную программу WES, которая была анонсирована в сентябре 2011 года и была прекращена в 2012 году. Потребители могли получить данные экзома по цене 999 долларов. Компания предоставляла необработанные данные и не предлагала анализ. [27] [28] [29]
В ноябре 2012 года DNADTC, подразделение Gene by Gene , начало предлагать экзомы с покрытием 80X и начальной ценой 695 долларов США. [30] В настоящее время эта цена на веб-сайте DNADTC составляет 895 долларов США. В октябре 2013 года BGI объявила о рекламной акции для персонального полного секвенирования экзома с покрытием 50X за 499 долларов США. [31] В июне 2016 года Genos удалось добиться еще более низкой цены в 399 долларов США с сертифицированным CLIA потребительским экзомом 75X, секвенированным из слюны. [32] [33] [34]
Обзор 36 исследований, проведенный в 2018 году, показал, что стоимость экзомного секвенирования составляет от 555 до 5169 долларов США, а диагностическая ценность — от 3% до 79% в зависимости от группы пациентов. [16]