
Биоинформатика ( / ˌ b aɪ . oʊ ˌ ɪ n f ər ˈ m æ t ɪ k s / ⓘ ) —междисциплинарнаяобластьнауки, которая разрабатывает методы ипрограммные средствадля пониманиябиологическихданных, особенно когда наборы данных большие и сложные. Биоинформатика используетбиологию,химию,физику,информатику,компьютерное программирование,информационную инженерию,математикуистатистикудля анализа и интерпретациибиологических данных. Последующий процесс анализа и интерпретации данных называетсявычислительной биологией.
Вычислительные, статистические методы и методы компьютерного программирования использовались для компьютерного моделирования биологических запросов. Они включают в себя повторно используемые специфические «конвейеры» анализа, особенно в области геномики , например, путем идентификации генов и однонуклеотидных полиморфизмов ( SNP ). Эти конвейеры используются для лучшего понимания генетической основы болезней, уникальных адаптаций, желаемых свойств (особенно у сельскохозяйственных видов) или различий между популяциями. Биоинформатика также включает протеомику , которая пытается понять принципы организации последовательностей нуклеиновых кислот и белков . [1]
Обработка изображений и сигналов позволяет извлекать полезные результаты из больших объемов необработанных данных. В области генетики он помогает секвенировать и аннотировать геномы и их наблюдаемые мутации . Биоинформатика включает в себя анализ текста биологической литературы и разработку биологических и генных онтологий для организации и запроса биологических данных. Он также играет роль в анализе экспрессии и регуляции генов и белков. Инструменты биоинформатики помогают сравнивать, анализировать и интерпретировать генетические и геномные данные и, в более общем плане, понимать эволюционные аспекты молекулярной биологии. На более интегративном уровне это помогает анализировать и каталогизировать биологические пути и сети, которые являются важной частью системной биологии . В структурной биологии он помогает моделировать ДНК, [2] РНК, [2] [3] белки [4] , а также биомолекулярные взаимодействия. [5] [6] [7] [8]
Первое определение термина «биоинформатика» было дано Паулином Хогевегом и Беном Хеспером в 1970 году для обозначения изучения информационных процессов в биотических системах. [9] [10] [11] [12] [13] Это определение помещало биоинформатику как область, параллельную биохимии (изучение химических процессов в биологических системах). [10]
Биоинформатика и вычислительная биология включали анализ биологических данных, особенно последовательностей ДНК, РНК и белков. Область биоинформатики пережила взрывной рост, начиная с середины 1990-х годов, во многом благодаря проекту «Геном человека» и быстрому развитию технологии секвенирования ДНК.
Анализ биологических данных для получения значимой информации включает в себя написание и запуск программ, использующих алгоритмы теории графов , искусственного интеллекта , мягких вычислений , интеллектуального анализа данных , обработки изображений и компьютерного моделирования . Алгоритмы, в свою очередь, зависят от теоретических основ, таких как дискретная математика , теория управления , теория систем , теория информации и статистика .

С момента завершения проекта «Геном человека» произошел огромный прогресс в скорости и сокращении затрат: некоторые лаборатории могут секвенировать более 100 000 миллиардов оснований каждый год, а полный геном можно секвенировать за 1000 долларов или меньше. [14]
Компьютеры стали играть важную роль в молекулярной биологии, когда стали доступны последовательности белков после того, как Фредерик Сэнгер определил последовательность инсулина в начале 1950-х годов. Сравнение нескольких последовательностей вручную оказалось непрактичным. Маргарет Окли Дэйхофф , пионер в этой области, [15] составила одну из первых баз данных последовательностей белков, первоначально опубликованных в виде книг [16] , а также методы выравнивания последовательностей и молекулярной эволюции . [17] Еще одним ранним вкладчиком в биоинформатику был Элвин А. Кабат , который впервые ввел анализ биологических последовательностей в 1970 году, опубликовав обширные тома последовательностей антител вместе с Тай Те Ву в период с 1980 по 1991 год. [18]
В 1970-х годах к бактериофагам MS2 и øX174 были применены новые методы секвенирования ДНК, а расширенные нуклеотидные последовательности затем были проанализированы с помощью информационных и статистических алгоритмов. Эти исследования показали, что хорошо известные особенности, такие как сегменты кодирования и триплетный код, выявляются при простом статистическом анализе и стали доказательством концепции того, что биоинформатика будет проницательной. [19] [20]

Чтобы изучить, как нормальная клеточная активность изменяется при различных болезненных состояниях, необходимо объединить необработанные биологические данные, чтобы сформировать полную картину этой активности. Поэтому [ когда? ] , область биоинформатики развилась таким образом, что наиболее насущная задача теперь связана с анализом и интерпретацией различных типов данных. Сюда также входят нуклеотидные и аминокислотные последовательности , белковые домены и белковые структуры . [21]
Важные субдисциплины биоинформатики и вычислительной биологии включают:
Основная цель биоинформатики — улучшить понимание биологических процессов. Что отличает его от других подходов, так это его ориентация на разработку и применение вычислительно-интенсивных методов для достижения этой цели. Примеры включают: распознавание образов , интеллектуальный анализ данных , алгоритмы машинного обучения и визуализацию . Основные исследовательские усилия в этой области включают выравнивание последовательностей , поиск генов , сборку генома , разработку лекарств , открытие лекарств , выравнивание структуры белков , предсказание структуры белков , предсказание экспрессии генов и белок-белковых взаимодействий , полногеномные исследования ассоциаций , моделирование эволюции . и деление клеток/митоз.
Биоинформатика влечет за собой создание и развитие баз данных, алгоритмов, вычислительных и статистических методов, а также теории для решения формальных и практических проблем, возникающих в результате управления и анализа биологических данных.
За последние несколько десятилетий быстрое развитие геномных и других технологий молекулярных исследований, а также развитие информационных технологий в совокупности привело к появлению огромного количества информации, связанной с молекулярной биологией. Биоинформатика — это название, данное математическим и вычислительным подходам, используемым для понимания биологических процессов.
Обычные действия в биоинформатике включают картирование и анализ последовательностей ДНК и белков, выравнивание последовательностей ДНК и белков для их сравнения, а также создание и просмотр трехмерных моделей белковых структур.
С момента секвенирования бактериофага Phage Φ-X174 в 1977 году [22] последовательности ДНК тысяч организмов были расшифрованы и сохранены в базах данных. Эта информация о последовательностях анализируется для определения генов, которые кодируют белки , гены РНК, регуляторные последовательности, структурные мотивы и повторяющиеся последовательности. Сравнение генов внутри одного вида или между разными видами может показать сходство функций белков или отношения между видами (использование молекулярной систематики для построения филогенетических деревьев ). С ростом объема данных анализ последовательностей ДНК вручную уже давно стал непрактичным. Компьютерные программы, такие как BLAST , регулярно используются для поиска последовательностей — по состоянию на 2008 год из более чем 260 000 организмов, содержащих более 190 миллиардов нуклеотидов . [23]
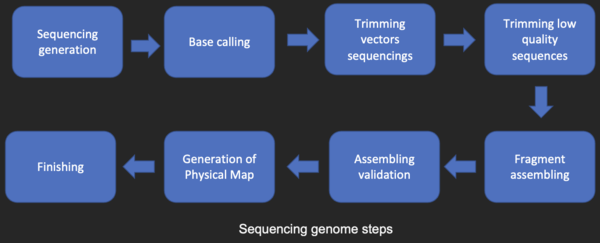
Прежде чем последовательности можно будет проанализировать, их получают из банка хранения данных, такого как GenBank. Секвенирование ДНК по-прежнему остается нетривиальной проблемой, поскольку необработанные данные могут быть зашумлены или на них могут влиять слабые сигналы. Были разработаны алгоритмы определения оснований для различных экспериментальных подходов к секвенированию ДНК.
Большинство методов секвенирования ДНК позволяют получить короткие фрагменты последовательности, которые необходимо собрать для получения полных последовательностей гена или генома. Метод секвенирования дробовика (используемый Институтом геномных исследований (TIGR) для секвенирования первого бактериального генома Haemophilus influenzae ) [24] генерирует последовательности многих тысяч небольших фрагментов ДНК (длиной от 35 до 900 нуклеотидов, в зависимости от технология секвенирования). Концы этих фрагментов перекрываются, и при правильном выравнивании с помощью программы сборки генома их можно использовать для реконструкции полного генома. Секвенирование методом дробовика позволяет быстро получить данные о последовательностях, но задача сборки фрагментов может быть довольно сложной для более крупных геномов. Для генома такого размера, как геном человека , на многопроцессорных компьютерах с большой памятью может потребоваться много дней процессорного времени, чтобы собрать фрагменты, и полученная сборка обычно содержит множество пробелов, которые необходимо заполнить позже. Секвенирование дробовика — метод выбора практически для всех секвенированных геномов (а не методы обрыва цепи или химической деградации), а алгоритмы сборки генома — важнейшая область исследований в области биоинформатики.
В геномике аннотация относится к процессу маркировки стоп- и стартовых областей генов и других биологических особенностей в секвенированной последовательности ДНК . Многие геномы слишком велики, чтобы их можно было аннотировать вручную. Поскольку скорость секвенирования превышает скорость аннотации генома, аннотация генома стала новым узким местом в биоинформатике [ когда? ] .
Аннотацию генома можно разделить на три уровня: уровень нуклеотидов , белков и уровень процессов.
Обнаружение генов является главным аспектом аннотации на уровне нуклеотидов. Для сложных геномов может оказаться успешным сочетание прогнозирования генов ab initio и сравнения последовательностей с базами данных экспрессируемых последовательностей и другими организмами. Аннотация на уровне нуклеотидов также позволяет интегрировать последовательность генома с другими генетическими и физическими картами генома.
Основная цель аннотации на уровне белка — присвоить функции белковым продуктам генома. Для этого типа аннотации используются базы данных белковых последовательностей, функциональных доменов и мотивов. Около половины предсказанных белков в новой последовательности генома, как правило, не имеют очевидной функции.
Понимание функции генов и их продуктов в контексте клеточной и организменной физиологии является целью аннотации на уровне процесса. Препятствием для аннотаций на уровне процесса было несоответствие терминов, используемых разными модельными системами. Консорциум Gene Ontology помогает решить эту проблему. [25]
Первое описание комплексной системы аннотаций было опубликовано в 1995 году [24] Институтом геномных исследований , который выполнил первое полное секвенирование и анализ генома свободноживущего (несимбиотического ) организма — бактерии Haemophilus influenzae . [24] Система идентифицирует гены, кодирующие все белки, транспортные РНК, рибосомальные РНК, чтобы выполнить первоначальные функциональные назначения. Программа GeneMark , предназначенная для поиска генов, кодирующих белок у Haemophilus influenzae , постоянно меняется и совершенствуется.
Следуя целям, которые проект «Геном человека» оставил после своего закрытия в 2003 году, Национальный исследовательский институт генома человека разработал проект ENCODE . Этот проект представляет собой совместный сбор данных о функциональных элементах человеческого генома, в котором используются технологии секвенирования ДНК нового поколения и массивы геномных плиток, технологии, способные автоматически генерировать большие объемы данных при значительно сниженных затратах на каждую базу, но с теми же точность (ошибка вызова основания) и верность (ошибка сборки).
Хотя аннотация генома в первую очередь основана на сходстве последовательностей (и, следовательно, гомологии ), другие свойства последовательностей могут использоваться для прогнозирования функции генов. Фактически, большинство методов прогнозирования функций генов сосредоточены на белковых последовательностях, поскольку они более информативны и содержат больше возможностей. Например, распределение гидрофобных аминокислот предсказывает наличие трансмембранных сегментов в белках. Однако для прогнозирования функции белка можно также использовать внешнюю информацию, такую как данные об экспрессии генов (или белков), структура белка или белок-белковые взаимодействия . [26]
Эволюционная биология — это изучение происхождения и происхождения видов , а также их изменений с течением времени. Информатика помогла биологам-эволюционистам, позволив исследователям:
Будущие работы направлены на реконструкцию ныне более сложного древа жизни . [ по мнению кого? ]
Основой сравнительного анализа генома является установление соответствия между генами ( ортологический анализ) или другими признаками генома у разных организмов. Межгеномные карты создаются для отслеживания эволюционных процессов, ответственных за расхождение двух геномов. Множество эволюционных событий, действующих на различных организационных уровнях, формируют эволюцию генома. На самом низком уровне точковые мутации затрагивают отдельные нуклеотиды. На более высоком уровне крупные хромосомные сегменты подвергаются дупликации, латеральному переносу, инверсии, транспозиции, делеции и вставке. [28] Целые геномы участвуют в процессах гибридизации, полиплоидизации и эндосимбиоза , которые приводят к быстрому видообразованию. Сложность эволюции генома ставит множество интересных задач перед разработчиками математических моделей и алгоритмов, которые прибегают к широкому спектру алгоритмических, статистических и математических методов, начиная от точных, эвристических алгоритмов с фиксированным параметром и алгоритмов аппроксимации для задач, основанных на экономных моделях, до марковских алгоритмов. цепные алгоритмы Монте-Карло для байесовского анализа задач на основе вероятностных моделей.
Многие из этих исследований основаны на обнаружении гомологии последовательностей для отнесения последовательностей к семействам белков . [29]
Пангеномика — это концепция, предложенная в 2005 году Теттелином и Медини. Пангеном — это полный репертуар генов определенной монофилетической таксономической группы. Хотя первоначально оно применялось к близкородственным штаммам вида, его можно применять и к более широкому контексту, например, к роду, типу и т. д. Он разделен на две части: основной геном, набор генов, общий для всех изучаемых геномов (часто гены «домашнего хозяйства», жизненно важные для выживания), а также необязательный/гибкий геном: набор генов, присутствующих не во всех изучаемых геномах, кроме одного или некоторых. Инструмент биоинформатики BPGA можно использовать для характеристики пангенома видов бактерий. [30]
По состоянию на 2013 год существование эффективной высокопроизводительной технологии секвенирования нового поколения позволяет выявлять причины множества различных заболеваний человека. Простое менделевское наследование наблюдалось для более чем 3000 расстройств, которые были идентифицированы в онлайн-базе данных «Менделевское наследование у человека» , но сложные заболевания представляют собой более сложную задачу. Исследования ассоциаций обнаружили множество отдельных генетических областей, которые по отдельности слабо связаны со сложными заболеваниями (такими как бесплодие , [31] рак молочной железы [32] и болезнь Альцгеймера [33] ), а не с одной причиной. [34] [35] В настоящее время существует множество проблем с использованием генов для диагностики и лечения, например, то, что мы не знаем, какие гены важны, или насколько стабильный выбор обеспечивает алгоритм. [36]
Полногеномные исследования ассоциаций успешно выявили тысячи общих генетических вариантов сложных заболеваний и признаков; однако эти общие варианты объясняют лишь небольшую часть наследственности. [37] Редкие варианты могут частично объяснять недостающую наследственность . [38] Крупномасштабные исследования секвенирования всего генома быстро секвенировали миллионы целых геномов, и такие исследования выявили сотни миллионов редких вариантов . [39] Функциональные аннотации предсказывают эффект или функцию генетического варианта и помогают определить приоритет редких функциональных вариантов, а включение этих аннотаций может эффективно повысить эффективность генетической ассоциации анализа редких вариантов в исследованиях полногеномного секвенирования. [40] Были разработаны некоторые инструменты для комплексного анализа ассоциаций редких вариантов для данных полногеномного секвенирования, включая интеграцию данных генотипа и их функциональных аннотаций, анализ ассоциаций, сводку результатов и визуализацию. [41] [42] Мета-анализ исследований полногеномного секвенирования обеспечивает привлекательное решение проблемы сбора больших размеров выборки для обнаружения редких вариантов, связанных со сложными фенотипами. [43]
При раке геномы пораженных клеток перестраиваются сложным или непредсказуемым образом. В дополнение к массивам однонуклеотидного полиморфизма , выявляющим точечные мутации , вызывающие рак, олигонуклеотидные микрочипы могут использоваться для выявления хромосомных приобретений и потерь (так называемая сравнительная геномная гибридизация ). Эти методы обнаружения генерируют терабайты данных за эксперимент. Часто обнаруживается, что данные содержат значительную изменчивость или шум , и поэтому разрабатываются скрытая модель Маркова и методы анализа точек изменений, чтобы сделать вывод о реальных изменениях количества копий . [ нужна цитата ]
Для идентификации рака по мутациям в экзоме можно использовать два важных принципа . Во-первых, рак — это заболевание, вызванное накопленными соматическими мутациями в генах. Во-вторых, рак содержит мутации водителя, которые необходимо отличать от мутаций пассажиров. [44]
Дальнейшие улучшения в биоинформатике могут позволить классифицировать типы рака путем анализа вызванных раком мутаций в геноме. Более того, отслеживание пациентов по мере прогрессирования заболевания может стать возможным в будущем с помощью последовательности образцов рака. Другой тип данных, требующий развития новой информатики, — это анализ поражений , которые оказались рецидивирующими среди многих опухолей. [45]
Экспрессию многих генов можно определить путем измерения уровней мРНК с помощью нескольких методов, включая микрочипы , секвенирование меток экспрессированной последовательности кДНК (EST), секвенирование меток последовательного анализа экспрессии генов (SAGE), массово - параллельное сигнатурное секвенирование (MPSS), RNA-Seq , также известный как «целое транскриптомное секвенирование» (WTSS), или различные применения мультиплексной гибридизации in-situ. Все эти методы чрезвычайно подвержены шуму и/или подвержены систематической ошибке при биологических измерениях, а основная область исследований в вычислительной биологии включает разработку статистических инструментов для отделения сигнала от шума в высокопроизводительных исследованиях экспрессии генов. [46] Такие исследования часто используются для определения генов, участвующих в заболевании: можно сравнить данные микрочипов раковых эпителиальных клеток с данными нераковых клеток, чтобы определить транскрипты, активация которых повышена или понижена в конкретной популяции. раковых клеток.

Белковые микроматрицы и масс-спектрометрия (МС) с высокой пропускной способностью (HT) могут предоставить моментальный снимок белков, присутствующих в биологическом образце. Первый подход сталкивается с теми же проблемами, что и микрочипы, нацеленные на мРНК, второй включает в себя проблему сопоставления больших объемов данных о массах с предсказанными массами из баз данных последовательностей белков, а также сложный статистический анализ образцов при обнаружении нескольких неполных пептидов из каждого белка. Локализация клеточного белка в тканевом контексте может быть достигнута с помощью аффинной протеомики , отображаемой в виде пространственных данных, основанных на иммуногистохимии и тканевых микрочипах . [47]
Регуляция генов — это сложный процесс, при котором сигнал, например внеклеточный сигнал, такой как гормон , в конечном итоге приводит к увеличению или снижению активности одного или нескольких белков . Методы биоинформатики применялись для изучения различных этапов этого процесса.
Например, экспрессия генов может регулироваться близлежащими элементами генома. Анализ промотора включает идентификацию и изучение мотивов последовательности в ДНК, окружающей белок-кодирующую область гена. Эти мотивы влияют на степень, в которой эта область транскрибируется в мРНК. Элементы -энхансеры, расположенные далеко от промотора, также могут регулировать экспрессию генов посредством трехмерных петлевых взаимодействий. Эти взаимодействия могут быть определены с помощью биоинформатического анализа экспериментов по захвату конформации хромосом .
Данные об экспрессии можно использовать для вывода о регуляции генов: можно сравнить данные микрочипов из самых разных состояний организма, чтобы сформировать гипотезы о генах, участвующих в каждом состоянии. В одноклеточном организме можно сравнить стадии клеточного цикла , а также различные стрессовые состояния (тепловой шок, голодание и т. д.). Затем к данным экспрессии можно применить алгоритмы кластеризации , чтобы определить, какие гены экспрессируются совместно. Например, в вышележащих регионах (промоторах) совместно экспрессируемых генов можно искать чрезмерно представленные регуляторные элементы . Примерами алгоритмов кластеризации, применяемых при кластеризации генов, являются кластеризация k-средних , самоорганизующиеся карты (SOM), иерархическая кластеризация и методы консенсусной кластеризации .
Было разработано несколько подходов для анализа расположения органелл, генов, белков и других компонентов внутри клеток. Категория онтологии генов , клеточный компонент , была разработана для определения субклеточной локализации во многих биологических базах данных .
Микроскопические изображения позволяют определить местонахождение органелл , а также молекул, которые могут быть источником аномалий при заболеваниях.
Обнаружение местоположения белков позволяет нам предсказать, что они делают. Это называется предсказанием функции белка . Например, если белок обнаружен в ядре , он может участвовать в регуляции генов или сплайсинге . Напротив, если белок обнаружен в митохондриях , он может участвовать в дыхании или других метаболических процессах . Существуют хорошо развитые ресурсы для прогнозирования субклеточной локализации белков , включая базы данных субклеточной локализации белков и инструменты прогнозирования. [48] [49]
Данные высокопроизводительных экспериментов по захвату конформации хромосом , таких как Hi-C (эксперимент) и ChIA-PET , могут предоставить информацию о трехмерной структуре и ядерной организации хроматина . Биоинформационные задачи в этой области включают разделение генома на домены, такие как топологически ассоциированные домены (TAD), которые организованы вместе в трехмерном пространстве. [50]

Поиск структуры белков является важным применением биоинформатики. Критическая оценка прогнозирования структуры белка (CASP) — это открытый конкурс, в ходе которого исследовательские группы со всего мира представляют модели белков для оценки неизвестных моделей белков. [51] [52]
Линейная аминокислотная последовательность белка называется первичной структурой . Первичную структуру можно легко определить по последовательности кодонов гена ДНК, который ее кодирует. В большинстве белков первичная структура однозначно определяет трехмерную структуру белка в его нативной среде. Исключением является неправильно свернутый белок , участвующий в губчатой энцефалопатии крупного рогатого скота . Эта структура связана с функцией белка. Дополнительная структурная информация включает вторичную , третичную и четвертичную структуру. Жизнеспособное общее решение для предсказания функции белка остается открытой проблемой. До сих пор большинство усилий было направлено на эвристику, которая работает большую часть времени. [ нужна цитата ]
В геномной отрасли биоинформатики гомология используется для предсказания функции гена: если последовательность гена A , функция которого известна, гомологична последовательности гена B, функция которого неизвестна, можно сделать вывод, что B может разделить функцию А. В структурной биоинформатике гомология используется для определения того, какие части белка важны для формирования структуры и взаимодействия с другими белками. Моделирование гомологии используется для прогнозирования структуры неизвестного белка на основе существующих гомологичных белков.
Одним из примеров этого является гемоглобин человека и гемоглобин бобовых ( леггемоглобин ), которые являются дальними родственниками одного и того же суперсемейства белков . Оба служат одной и той же цели – транспортировке кислорода в организме. Хотя оба этих белка имеют совершенно разные аминокислотные последовательности, их белковые структуры практически идентичны, что отражает их почти идентичные цели и общего предка. [53]
Другие методы прогнозирования структуры белка включают в себя создание нитей белка и моделирование на основе физики de novo (с нуля).
Другой аспект структурной биоинформатики включает использование белковых структур для моделей виртуального скрининга , таких как модели количественной взаимосвязи структура-активность и протеохемометрические модели (PCM). Кроме того, кристаллическую структуру белка можно использовать при моделировании, например, исследований связывания лигандов и в исследованиях кремнеземного мутагенеза.
Программное обеспечение 2021 года на основе алгоритмов глубокого обучения под названием AlphaFold , разработанное компанией Google DeepMind , значительно превосходит все другие методы программного обеспечения для прогнозирования [54] [ как? ] и опубликовал предсказанные структуры для сотен миллионов белков в базе данных структур белков AlphaFold. [55]
Сетевой анализ направлен на понимание взаимоотношений внутри биологических сетей, таких как метаболические сети или сети межбелкового взаимодействия . Хотя биологические сети могут быть построены из одного типа молекул или объектов (например, генов), сетевая биология часто пытается интегрировать множество различных типов данных, таких как белки, небольшие молекулы, данные об экспрессии генов и другие, которые все связаны физически. , функционально или и то, и другое.
Системная биология предполагает использование компьютерного моделирования клеточных подсистем (таких как сети метаболитов и ферментов , которые включают метаболизм , пути передачи сигналов и сети регуляции генов ) для анализа и визуализации сложных связей этих клеточных процессов. Искусственная жизнь или виртуальная эволюция пытается понять эволюционные процессы посредством компьютерного моделирования простых (искусственных) форм жизни.

Десятки тысяч трехмерных белковых структур были определены с помощью рентгеновской кристаллографии и спектроскопии ядерного магнитного резонанса белков (ЯМР белков), и центральный вопрос структурной биоинформатики заключается в том, практично ли прогнозировать возможные белок-белковые взаимодействия только на основе этих структур. 3D-формы без проведения экспериментов по межбелковому взаимодействию . Для решения проблемы докинга белков было разработано множество методов , хотя кажется, что в этой области еще предстоит проделать большую работу.
Другие взаимодействия, встречающиеся в этой области, включают белок-лиганд (включая лекарственное средство) и белок-пептид. Молекулярно-динамическое моделирование движения атомов вокруг вращающихся связей является фундаментальным принципом вычислительных алгоритмов , называемых алгоритмами стыковки, для изучения молекулярных взаимодействий .
Информатика биоразнообразия занимается сбором и анализом данных о биоразнообразии , таких как таксономические базы данных или данные микробиома . Примеры такого анализа включают филогенетику , моделирование ниш , картирование видового богатства , штрих-кодирование ДНК или инструменты идентификации видов . Растущей областью также является макроэкология , т.е. изучение того, как биоразнообразие связано с экологией и воздействием человека, например, изменением климата .
Огромное количество опубликованной литературы делает практически невозможным для отдельных лиц читать каждую статью, что приводит к разрознению подобластей исследований. Целью анализа литературы является использование компьютерной и статистической лингвистики для изучения этой растущей библиотеки текстовых ресурсов. Например:
Область исследований опирается на статистику и компьютерную лингвистику .
Вычислительные технологии используются для автоматизации обработки, количественной оценки и анализа больших объемов биомедицинских изображений с высоким содержанием информации . Современные системы анализа изображений могут повысить точность , объективность и скорость наблюдения наблюдателя . Анализ изображений важен как для диагностики, так и для исследований. Некоторые примеры:
Вычислительные методы используются для анализа высокопроизводительных и малообъемных данных об отдельных клетках, например, полученных с помощью проточной цитометрии . Эти методы обычно включают поиск популяций клеток, соответствующих конкретному болезненному состоянию или экспериментальному состоянию.
Биологические онтологии представляют собой ориентированные ациклические графы управляемых словарей . Они создают категории для биологических концепций и описаний, чтобы их можно было легко анализировать с помощью компьютеров. При такой классификации можно получить дополнительную выгоду от целостного и интегрированного анализа. [ нужна цитата ]
OBO Foundry была попыткой стандартизировать определенные онтологии. Одной из наиболее распространенных является онтология генов , описывающая функции генов. Существуют также онтологии, описывающие фенотипы.
Базы данных необходимы для исследований и приложений в области биоинформатики. Базы данных существуют для множества различных типов информации, включая последовательности ДНК и белков, молекулярные структуры, фенотипы и биоразнообразие. Базы данных могут содержать как эмпирические данные (полученные непосредственно в результате экспериментов), так и прогнозируемые данные (полученные в результате анализа существующих данных). Они могут быть специфичными для конкретного организма, пути или молекулы, представляющей интерес. Альтернативно они могут включать данные, собранные из нескольких других баз данных. Базы данных могут иметь разные форматы, механизмы доступа и быть общедоступными или частными.
Ниже перечислены некоторые из наиболее часто используемых баз данных:
Программные инструменты для биоинформатики включают простые инструменты командной строки, более сложные графические программы и автономные веб-сервисы. Их производят биоинформатические компании или государственные учреждения.
Многие бесплатные программные инструменты с открытым исходным кодом существовали и продолжали расти с 1980-х годов. [57] Сочетание постоянной потребности в новых алгоритмах для анализа новых типов биологических данных, потенциала инновационных экспериментов in silico и свободно доступных баз открытого кода создало для исследовательских групп возможности внести свой вклад в биоинформатику независимо от финансирования. . Инструменты с открытым исходным кодом часто выступают в качестве инкубаторов идей или поддерживаемых сообществом плагинов для коммерческих приложений. Они также могут предоставлять стандарты де-факто и общие объектные модели для решения проблемы интеграции биоинформации.
Программное обеспечение для биоинформатики с открытым исходным кодом включает Bioconductor , BioPerl , Biopython , BioJava , BioJS , BioRuby , Bioclipse , EMBOSS , .NET Bio , Orange с надстройкой для биоинформатики, Apache Taverna , UGENE и GenoCAD .
Некоммерческий Фонд открытой биоинформатики [57] и ежегодная конференция по биоинформатике с открытым исходным кодом продвигают программное обеспечение для биоинформатики с открытым исходным кодом. [58]
Интерфейсы на основе SOAP и REST были разработаны, чтобы позволить клиентским компьютерам использовать алгоритмы, данные и вычислительные ресурсы с серверов в других частях мира. Основное преимущество заключается в том, что конечным пользователям не придется иметь дело с накладными расходами на обслуживание программного обеспечения и баз данных.
Основные биоинформатические услуги классифицируются EBI на три категории: SSS (службы поиска последовательностей), MSA (множественное выравнивание последовательностей) и BSA (анализ биологических последовательностей). [59] Доступность этих сервисно-ориентированных биоинформатических ресурсов демонстрирует применимость сетевых биоинформатических решений и варьируется от набора автономных инструментов с общим форматом данных под единым веб-интерфейсом до интегративной, распределенной и расширяемой биоинформатики . системы управления рабочим процессом .
Система управления рабочими процессами биоинформатики — это специализированная форма системы управления рабочими процессами , разработанная специально для составления и выполнения ряда шагов вычислений или обработки данных или рабочего процесса в приложении биоинформатики. Такие системы предназначены для
Некоторые платформы, предоставляющие эту услугу: Galaxy , Kepler , Taverna , UGENE , Anduril , HIVE .
В 2014 году Управление по санитарному надзору за качеством пищевых продуктов и медикаментов США спонсировало конференцию, проведенную в кампусе Национального института здравоохранения в Бетесде, чтобы обсудить воспроизводимость в биоинформатике. [60] В течение следующих трех лет консорциум заинтересованных сторон регулярно встречался, чтобы обсудить то, что впоследствии станет парадигмой биокомпьютеров. [61] В число заинтересованных сторон входили представители правительства, промышленности и научных учреждений. Лидеры сессии представляли многочисленные филиалы институтов и центров FDA и NIH, некоммерческие организации, включая проект Human Variome и Европейскую федерацию медицинской информатики , а также исследовательские институты, включая Стэнфорд , Нью-Йоркский центр генома и Университет Джорджа Вашингтона .
Было решено, что парадигма BioCompute будет иметь форму цифровых «лабораторных ноутбуков», которые позволят обеспечить воспроизводимость, репликацию, анализ и повторное использование протоколов биоинформатики. Это было предложено для обеспечения большей преемственности внутри исследовательской группы в ходе обычного потока кадров, одновременно способствуя обмену идеями между группами. FDA США профинансировало эту работу, чтобы информация о трубопроводах была более прозрачной и доступной для сотрудников регулирующих органов. [62]
В 2016 году группа вновь собралась в НИЗ в Бетесде и обсудила потенциал объекта BioCompute — примера парадигмы BioCompute. Эта работа была скопирована как документ «стандартного пробного использования», так и как препринт, загруженный в bioRxiv. Объект BioCompute позволяет делиться записью в формате JSON между сотрудниками, сотрудниками и регулирующими органами. [63] [64]
Биоинформатика во многих университетах преподается не только в магистратуре . Вычислительная природа биоинформатики позволяет использовать ее для компьютерного и онлайн-обучения . [65] [66] Программные платформы, предназначенные для обучения концепциям и методам биоинформатики, включают Розалинду и онлайн-курсы, предлагаемые через учебный портал Швейцарского института биоинформатики . Канадские семинары по биоинформатике размещают на своем веб-сайте видео и слайды с учебных семинаров по лицензии Creative Commons . Проект 4273π или проект 4273pi [67] также предлагает бесплатные образовательные материалы с открытым исходным кодом. Курс проводится на недорогих компьютерах Raspberry Pi и используется для обучения взрослых и школьников. [68] [69] 4283 активно разрабатывается консорциумом ученых и исследователей, которые проводили исследования в области биоинформатики с использованием компьютеров Raspberry Pi и операционной системы 4283π. [70] [71]
Платформы MOOC также предоставляют онлайн-сертификацию по биоинформатике и смежным дисциплинам, включая специализацию Coursera по биоинформатике ( Калифорнийский университет в Сан-Диего ) и специализацию по геномным данным ( Джонс Хопкинс ), а также по анализу данных EdX для наук о жизни XSeries ( Гарвард ).
Есть несколько крупных конференций, посвященных биоинформатике. Некоторые из наиболее ярких примеров — «Интеллектуальные системы для молекулярной биологии» (ISMB), Европейская конференция по вычислительной биологии (ECCB) и «Исследования в области вычислительной молекулярной биологии» (RECOMB).