
Протеомика — это крупномасштабное исследование белков . [1] [2] Белки — это жизненно важные макромолекулы всех живых организмов, обладающие множеством функций, таких как формирование структурных волокон мышечной ткани , ферментативное переваривание пищи или синтез и репликация ДНК . Кроме того, другие виды белков включают антитела , которые защищают организм от инфекции, и гормоны , которые посылают важные сигналы по всему телу.
Протеом — это весь набор белков, производимых или модифицируемых организмом или системой. Протеомика позволяет идентифицировать все большее количество белков. Это меняется со временем и различными требованиями или стрессами, которым подвергается клетка или организм. [3]
Протеомика — это междисциплинарная область, которая извлекла большую пользу из генетической информации из различных геномных проектов, включая проект «Геном человека» . [4] Она охватывает исследование протеомов на общем уровне состава, структуры и активности белков и является важным компонентом функциональной геномики .
Протеомика обычно обозначает крупномасштабный экспериментальный анализ белков и протеомов, но часто относится конкретно к очистке белков и масс-спектрометрии . Действительно, масс-спектрометрия является наиболее мощным методом анализа протеомов, как в больших образцах, состоящих из миллионов клеток [5], так и в отдельных клетках. [6] [7]
Первые исследования белков, которые можно было бы отнести к протеомике, начались в 1974 году после введения двумерного геля и картирования белков из бактерии Escherichia coli . [8]
Протеом — это смесь слов «белок» и «геном». Он был придуман в 1994 году тогдашним аспирантом Марком Уилкинсом в Университете Маккуори , [9] который основал первую специализированную лабораторию протеомики в 1995 году. [10] [11]
После геномики и транскриптомики протеомика является следующим шагом в изучении биологических систем. Она сложнее геномики, поскольку геном организма более или менее постоянен, тогда как протеомы различаются от клетки к клетке и время от времени. Отдельные гены экспрессируются в разных типах клеток, что означает, что даже базовый набор белков, производимых в клетке, должен быть идентифицирован. [12]
В прошлом это явление оценивалось с помощью анализа РНК, который, как было обнаружено, не коррелировал с содержанием белка. [13] [14] Теперь известно, что мРНК не всегда транслируется в белок, [15] и количество белка, произведенного для данного количества мРНК, зависит от гена, с которого он транскрибируется, и от физиологического состояния клетки. Протеомика подтверждает наличие белка и обеспечивает прямое измерение его количества. [ необходима цитата ]
Не только перевод с мРНК вызывает различия, но и многие белки также подвергаются широкому спектру химических модификаций после трансляции. Наиболее распространенные и широко изученные посттрансляционные модификации включают фосфорилирование и гликозилирование. Многие из этих посттрансляционных модификаций имеют решающее значение для функции белка. [ необходима цитата ]
Одной из таких модификаций является фосфорилирование , которое происходит со многими ферментами и структурными белками в процессе передачи сигналов клетками . Добавление фосфата к определенным аминокислотам — чаще всего серину и треонину [16], опосредованное серин-треониновыми киназами , или, реже, тирозину, опосредованное тирозинкиназами, — приводит к тому, что белок становится мишенью для связывания или взаимодействия с определенным набором других белков, которые распознают фосфорилированный домен. [ необходима цитата ]
Поскольку фосфорилирование белков является одной из наиболее изученных модификаций белков, многие «протеомные» усилия направлены на определение набора фосфорилированных белков в конкретной клетке или типе ткани при определенных обстоятельствах. Это предупреждает ученого о сигнальных путях, которые могут быть активны в этом случае.
Убиквитин — это небольшой белок, который может быть прикреплен к определенным белковым субстратам ферментами, называемыми E3 убиквитинлигазами . Определение того, какие белки полиубиквитинированы, помогает понять, как регулируются белковые пути. Таким образом, это дополнительное законное «протеомное» исследование. Аналогично, как только исследователь определяет, какие субстраты убиквитинируются каждой лигазой, определение набора лигаз, экспрессируемых в определенном типе клеток, оказывается полезным. [ необходима цитата ]
В дополнение к фосфорилированию и убиквитинированию , белки могут подвергаться (среди прочего) метилированию , ацетилированию , гликозилированию , окислению и нитрозилированию . Некоторые белки подвергаются всем этим модификациям, часто в зависящих от времени комбинациях. Это иллюстрирует потенциальную сложность изучения структуры и функции белков.
Клетка может производить различные наборы белков в разное время или при разных условиях, например, во время развития , клеточной дифференциации , клеточного цикла или канцерогенеза . Дальнейшее увеличение сложности протеома, как уже упоминалось, заключается в том, что большинство белков способны подвергаться широкому спектру посттрансляционных модификаций.
Таким образом, исследование «протеомики» может очень быстро стать сложным, даже если тема исследования ограничена. В более амбициозных условиях, например, когда ищут биомаркер для определенного подтипа рака, ученый-протеомик может выбрать изучение нескольких образцов сыворотки крови от нескольких пациентов с раком, чтобы минимизировать мешающие факторы и учесть экспериментальный шум. [17] Таким образом, сложные экспериментальные конструкции иногда необходимы для учета динамической сложности протеома.
Протеомика дает иной уровень понимания, чем геномика, по многим причинам:
Воспроизводимость . Одним из основных факторов, влияющих на воспроизводимость в экспериментах по протеомике, является одновременное элюирование гораздо большего количества пептидов, чем могут измерить масс-спектрометры. Это приводит к стохастическим различиям между экспериментами из-за зависящего от данных получения триптических пептидов. Хотя ранние крупномасштабные протеомные анализы методом дробовика показали значительную вариабельность между лабораториями, [20] [21] предположительно из-за технических и экспериментальных различий между лабораториями, воспроизводимость была улучшена в более поздних масс-спектрометрических анализах, особенно на уровне белков. [22] Примечательно, что целевая протеомика показывает повышенную воспроизводимость и повторяемость по сравнению с методами дробовика, хотя и за счет плотности данных и эффективности. [23]
Качество данных . Протеомный анализ легко поддается автоматизации, и создаются большие наборы данных, которые обрабатываются программными алгоритмами. Параметры фильтров используются для уменьшения количества ложных срабатываний, но их невозможно полностью исключить. Ученые выразили необходимость осознания того, что эксперименты по протеомике должны соответствовать критериям аналитической химии (достаточное качество данных, проверка работоспособности, валидация). [24] [25] [26] [27]
В протеомике существует множество методов изучения белков. Обычно белки можно обнаружить с помощью антител (иммуноанализ), электрофоретического разделения или масс-спектрометрии . Если анализируется сложный биологический образец, необходимо либо использовать очень специфичное антитело в количественном дот-блот-анализе (QDB), либо использовать биохимическое разделение перед этапом обнаружения, поскольку в образце слишком много аналитов для проведения точного обнаружения и количественной оценки.
Антитела к определенным белкам или их модифицированные формы использовались в исследованиях биохимии и клеточной биологии . Это одни из самых распространенных инструментов, используемых молекулярными биологами сегодня. Существует несколько специальных методов и протоколов, которые используют антитела для обнаружения белков. Иммуноферментный анализ (ИФА) использовался в течение десятилетий для обнаружения и количественного измерения белков в образцах. Вестерн-блот может использоваться для обнаружения и количественного определения отдельных белков, где на начальном этапе сложная белковая смесь разделяется с помощью SDS-PAGE , а затем интересующий белок идентифицируется с помощью антитела. [ необходима цитата ]
Модифицированные белки могут быть изучены путем разработки антитела, специфичного для этой модификации. Например, некоторые антитела распознают только определенные белки, когда они фосфорилированы по тирозину , они известны как фосфоспецифичные антитела. Также существуют антитела, специфичные для других модификаций. Их можно использовать для определения набора белков, которые подверглись интересующей модификации. [ необходима цитата ]
Иммуноанализы также могут проводиться с использованием рекомбинантно созданных производных иммуноглобулина или синтетически разработанных белковых каркасов, которые выбираются для высокой антигенной специфичности. Такие связующие включают фрагменты антител с одним доменом (нанотела), [28] разработанные анкириновые повторные белки (DARPins) [29] и аптамеры. [30]
Выявление заболеваний на молекулярном уровне является движущей силой новой революции ранней диагностики и лечения. Проблема, с которой сталкивается эта область, заключается в том, что белковые биомаркеры для ранней диагностики могут присутствовать в очень малом количестве. Нижний предел обнаружения с помощью традиционной технологии иммуноанализа составляет верхний фемтомолярный диапазон (10−13 M ). Технология цифрового иммуноанализа повысила чувствительность обнаружения на три логарифма, до аттомолярного диапазона (10−16 M ). Эта возможность может открыть новые возможности в диагностике и терапии, но такие технологии были отнесены к ручным процедурам, которые не очень подходят для эффективного рутинного использования. [31]
Хотя обнаружение белков с помощью антител все еще очень распространено в молекулярной биологии, были разработаны и другие методы, которые не полагаются на антитела. Эти методы предлагают различные преимущества, например, они часто способны определять последовательность белка или пептида, они могут иметь более высокую пропускную способность, чем основанные на антителах, и иногда они могут идентифицировать и количественно определять белки, для которых не существует антител.
Одним из самых ранних методов анализа белков был метод деградации Эдмана (введенный в 1967 году), при котором один пептид подвергается нескольким этапам химической деградации для определения его последовательности. Эти ранние методы в основном были вытеснены технологиями, которые предлагают более высокую производительность. [ необходима цитата ]
В более поздних методах используются методы, основанные на масс-спектрометрии , разработка которых стала возможной благодаря открытию методов «мягкой ионизации», разработанных в 1980-х годах, таких как матрично-ассистированная лазерная десорбция/ионизация (MALDI) и электрораспылительная ионизация (ESI) . Эти методы дали начало рабочим процессам протеомики «сверху вниз» и « снизу вверх» , где часто перед анализом выполняется дополнительное разделение (см. ниже).
Для анализа сложных биологических образцов требуется снижение сложности образца. Это может быть выполнено в автономном режиме путем одномерного или двумерного разделения. Совсем недавно были разработаны методы в режиме онлайн, в которых отдельные пептиды (в подходах протеомики снизу вверх) разделяются с помощью обращенно-фазовой хроматографии , а затем напрямую ионизируются с помощью ESI ; прямое сопряжение разделения и анализа объясняет термин «онлайн» анализ.
Несколько гибридных технологий используют очистку отдельных аналитов на основе антител, а затем выполняют масс-спектрометрический анализ для идентификации и количественной оценки. Примерами этих методов являются MSIA (масс-спектрометрический иммуноанализ) , разработанный Рэндаллом Нельсоном в 1995 году [32] и метод SISCAPA (захват стандартного стабильного изотопа с помощью антипептидных антител), представленный Ли Андерсоном в 2004 году [33].
Флуоресцентный двумерный дифференциальный гель-электрофорез (2-D DIGE) [34] может быть использован для количественной оценки вариаций в процессе 2-D DIGE и установления статистически обоснованных пороговых значений для назначения количественных изменений между образцами. [34]
Сравнительный протеомный анализ может выявить роль белков в сложных биологических системах, включая воспроизводство. Например, обработка инсектицидом триазофосом вызывает увеличение содержания белков вспомогательных желез самцов цикадки бурой ( Nilaparvata lugens (Stål)) (Acps), которые могут передаваться самкам при спаривании, вызывая увеличение плодовитости (т. е. рождаемости) самок. [35] Чтобы выявить изменения в типах белков вспомогательных желез (Acps) и репродуктивных белков, которые спаривающиеся самки цикадки получали от самцов цикадки, исследователи провели сравнительный протеомный анализ спаривающихся самок N. lugens . [36] Результаты показали, что эти белки участвуют в репродуктивном процессе взрослых самок и самцов N. lugens . [36]
Протеомный анализ пероксисом Arabidopsis [37] был признан основным беспристрастным подходом к идентификации новых пероксисомальных белков в больших масштабах. [37]
Существует множество подходов к характеристике человеческого протеома, который, по оценкам, содержит от 20 000 до 25 000 не избыточных белков. Количество уникальных видов белков, вероятно, увеличится на 50 000–500 000 из-за сплайсинга РНК и протеолиза, а если также учесть посттрансляционную модификацию, общее количество уникальных человеческих белков оценивается в диапазоне нескольких миллионов. [38] [39]
Кроме того, недавно были опубликованы первые многообещающие попытки расшифровать протеом опухолей животных. [40] Этот метод использовался как функциональный метод при профилировании белков Macrobrachium rosenbergii . [41]
Протеомика неуклонно набирала обороты в течение последнего десятилетия с развитием нескольких подходов. Некоторые из них являются новыми, а другие основываются на традиционных методах. Методы, основанные на масс-спектрометрии, аффинная протеомика и микроматрицы являются наиболее распространенными технологиями для крупномасштабного изучения белков.
.jpg/440px-Thermo_-_Finnigan_LCQ_Mass_Spectrometer_(15797493459).jpg)
В настоящее время для профилирования белков используются два метода на основе масс-спектрометрии. Более устоявшийся и распространенный метод использует двумерный электрофорез высокого разрешения для параллельного разделения белков из разных образцов с последующим отбором и окрашиванием дифференциально экспрессируемых белков для идентификации с помощью масс-спектрометрии. Несмотря на достижения в области 2-DE и его зрелости, у него также есть свои ограничения. Главной проблемой является невозможность разрешить все белки в образце, учитывая их драматический диапазон в уровне экспрессии и различные свойства. Сочетание размера пор и заряда белка, размера и формы может в значительной степени определять скорость миграции, что приводит к другим осложнениям. [42]
Второй количественный подход использует стабильные изотопные метки для дифференциальной маркировки белков из двух различных сложных смесей. [43] [44] Здесь белки в сложной смеси сначала маркируются изотопно, а затем расщепляются для получения меченых пептидов. Затем меченые смеси объединяются, пептиды разделяются многомерной жидкостной хроматографией и анализируются тандемной масс-спектрометрией. Реагенты Isotope coded affinity tag (ICAT) являются широко используемыми изотопными метками. В этом методе остатки цистеина белков ковалентно прикрепляются к реагенту ICAT, тем самым уменьшая сложность смесей, исключая нецистеиновые остатки.
Количественная протеомика с использованием стабильной изотопной маркировки становится все более полезным инструментом в современных разработках. Во-первых, химические реакции использовались для введения меток в определенные сайты или белки с целью исследования определенных функциональных возможностей белка. Изоляция фосфорилированных пептидов была достигнута с использованием изотопной маркировки и селективной химии для захвата фракции белка среди сложной смеси. Во-вторых, технология ICAT использовалась для дифференциации частично очищенных или очищенных макромолекулярных комплексов, таких как большой комплекс преинициации РНК-полимеразы II и белков, комплексированных с фактором транскрипции дрожжей. В-третьих, маркировка ICAT недавно была объединена с изоляцией хроматина для идентификации и количественной оценки хроматин-ассоциированных белков. Наконец, реагенты ICAT полезны для протеомного профилирования клеточных органелл и определенных клеточных фракций. [42]
Другим количественным подходом является подход с точной массой и временем (AMT), разработанный Ричардом Д. Смитом и его коллегами в Pacific Northwest National Laboratory . В этом подходе повышенная пропускная способность и чувствительность достигаются за счет исключения необходимости в тандемной масс-спектрометрии и использования точно определенной информации о времени разделения и высокоточных определений массы для идентификации пептидов и белков.
Аффинная протеомика использует антитела или другие аффинные реагенты (например, аптамеры на основе олигонуклеотидов) в качестве зондов для специфического обнаружения белков. [45] В настоящее время этот метод может исследовать несколько тысяч белков, как правило, из биологических жидкостей, таких как плазма, сыворотка или спинномозговая жидкость (СМЖ). Ключевым отличием этой технологии является возможность анализировать сотни или тысячи образцов в разумные сроки (в течение нескольких дней или недель); методы, основанные на масс-спектрометрии, не масштабируются до такого уровня пропускной способности образцов для протеомных анализов.
Балансирование использования масс-спектрометров в протеомике и медицине заключается в использовании белковых микромассивов. Целью белковых микромассивов является печать тысяч признаков обнаружения белков для исследования биологических образцов. Массивы антител являются примером, в котором множество различных антител выстраиваются для обнаружения соответствующих антигенов из образца человеческой крови. Другой подход - это выстраивание нескольких типов белков для изучения таких свойств, как взаимодействия белок-ДНК, белок-белок и белок-лиганд. В идеале функциональные протеомные массивы должны содержать полный набор белков данного организма. Первая версия таких массивов состояла из 5000 очищенных белков из дрожжей, нанесенных на стеклянные предметные стекла микроскопа. Несмотря на успех первого чипа, реализация белковых массивов была более сложной задачей. С белками по своей природе гораздо сложнее работать, чем с ДНК. Они имеют широкий динамический диапазон, менее стабильны, чем ДНК, и их структуру трудно сохранить на стеклянных предметных стеклах, хотя они необходимы для большинства анализов. Глобальная технология ICAT имеет поразительные преимущества по сравнению с технологиями белковых чипов. [42]
Это многообещающее и новое применение микрочипов для диагностики, изучения и лечения сложных заболеваний, таких как рак. Технология объединяет лазерную микродиссекцию захвата (LCM) с технологией микрочипов для получения микрочипов белков с обратной фазой. В этом типе микрочипов весь набор белков иммобилизуется с целью захвата различных стадий заболевания у отдельного пациента. При использовании с LCM, массивы с обратной фазой могут контролировать флуктуирующее состояние протеома среди различных популяций клеток в пределах небольшой области человеческой ткани. Это полезно для профилирования статуса клеточных сигнальных молекул среди поперечного сечения ткани, которая включает как нормальные, так и раковые клетки. Этот подход полезен для мониторинга статуса ключевых факторов в нормальном эпителии простаты и тканях инвазивного рака простаты. Затем LCM рассекает эту ткань, и лизаты белков были выстроены на нитроцеллюлозных слайдах, которые были исследованы специфическими антителами. Этот метод может отслеживать все виды молекулярных событий и сравнивать больные и здоровые ткани у одного и того же пациента, что позволяет разрабатывать стратегии лечения и диагностику. Возможность получать протеомные снимки соседних популяций клеток с использованием микрочипов с обратной фазой в сочетании с LCM имеет ряд применений за пределами изучения опухолей. Этот подход может дать представление о нормальной физиологии и патологии всех тканей и бесценен для характеристики процессов развития и аномалий. [42]
Недавние достижения в области биоортогональной химии выявили применение в анализе белков. Расширение использования органических молекул для наблюдения за их реакцией с белками открывает обширные методы их маркировки. Неприродные аминокислоты и различные функциональные группы представляют собой новые растущие технологии в протеомике.
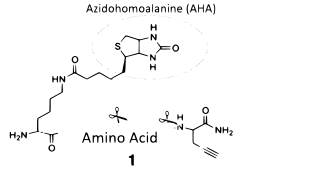
Определенные биомолекулы, которые способны метаболизироваться в клетках или тканях, встраиваются в белки или гликаны. Молекула будет иметь метку сродства, модифицирующую белок, что позволит ее обнаружить. Азидогомоаланин (AHA) использует эту метку сродства посредством включения с Met-t-RNA синтетазой для включения в белки. Это позволило AHA помочь в определении идентичности вновь синтезированных белков, созданных в ответ на возмущения , и идентифицировать белки, секретируемые клетками. [46]
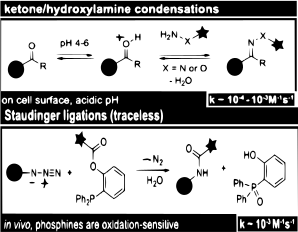
Недавние исследования [47] с использованием конденсаций кетонов и альдегидов показывают, что они лучше всего подходят для маркировки in vitro или клеточной поверхности . Однако использование кетонов и альдегидов в качестве биоортогональных репортеров выявило медленную кинетику, что указывает на то, что, хотя они эффективны для маркировки, концентрация должна быть высокой.
Некоторые белки могут быть обнаружены по их реактивности к азидным группам . Непротеиногенные аминокислоты могут иметь азидные группы, которые реагируют с фосфинами в лигированиях Штаудингера . Эта реакция уже использовалась для маркировки других биомолекул в живых клетках и животных. [48]
Биоортогональное поле расширяется и стимулирует дальнейшие приложения в протеомике. Стоит отметить ограничения и преимущества. Быстрые реакции могут создавать биоконъюнкции и создавать высокие концентрации с малым количеством реагентов. Напротив, медленные кинетические реакции, такие как конденсация альдегидов и кетонов, хотя и эффективны, требуют высокой концентрации, что делает их экономически неэффективными.
Одним из основных достижений в изучении человеческих генов и белков стало выявление потенциальных новых лекарств для лечения заболеваний. Это основано на информации о геноме и протеоме для определения белков, связанных с заболеванием, которые компьютерное программное обеспечение затем может использовать в качестве целей для новых лекарств. Например, если определенный белок участвует в заболевании, его трехмерная структура предоставляет информацию для разработки лекарств, которые будут мешать действию белка. Молекула, которая подходит к активному участку фермента, но не может быть высвобождена ферментом, инактивирует фермент. Это основа новых инструментов открытия лекарств, которые направлены на поиск новых лекарств для инактивации белков, вовлеченных в заболевание. По мере обнаружения генетических различий между людьми исследователи рассчитывают использовать эти методы для разработки персонализированных лекарств, которые будут более эффективны для человека. [49]
Протеомика также используется для выявления сложных взаимодействий растений и насекомых, которые помогают идентифицировать гены-кандидаты, участвующие в защитной реакции растений на травоядных. [50] [51] [52]
Раздел протеомики, называемый хемопротеомикой, предоставляет многочисленные инструменты и методы для обнаружения белковых мишеней лекарственных препаратов. [53]
Протеомика взаимодействия — это анализ взаимодействия белков от масштабов бинарных взаимодействий до протеомных или сетевых. Большинство белков функционируют посредством белок-белковых взаимодействий , и одной из целей протеомики взаимодействия является идентификация бинарных взаимодействий белков , белковых комплексов и интерактомов .
Существует несколько методов исследования белок-белковых взаимодействий . Хотя наиболее традиционным методом является двугибридный анализ дрожжей , мощным новым методом является аффинная очистка с последующей масс-спектрометрией белка с использованием меченых белковых приманок . Другие методы включают поверхностный плазмонный резонанс (SPR), [54] [55] белковые микроматрицы , двойную поляризационную интерферометрию , микромасштабный термофорез , кинетический эксклюзионный анализ и экспериментальные методы, такие как фаговый дисплей и вычислительные методы in silico .
Знание белок-белковых взаимодействий особенно полезно в отношении биологических сетей и системной биологии , например, в каскадах клеточных сигналов и сетях регуляции генов (GRN, где знание белок-ДНК взаимодействий также информативно). Анализ белковых взаимодействий на уровне протеома и интеграция этих моделей взаимодействия в более крупные биологические сети имеют решающее значение для понимания биологии системного уровня. [56] [57]
Протеомика экспрессии включает анализ экспрессии белка в более крупном масштабе. Она помогает идентифицировать основные белки в конкретном образце и те белки, которые дифференциально экспрессируются в родственных образцах, например, в больной и здоровой ткани. Если белок обнаружен только в больном образце, то он может быть полезной мишенью для препарата или диагностическим маркером. Белки с одинаковыми или похожими профилями экспрессии также могут быть функционально связаны. Существуют такие технологии, как 2D-PAGE и масс-спектрометрия , которые используются в протеомике экспрессии. [58]
Национальный институт здравоохранения определил биомаркер как «характеристику, которая объективно измеряется и оценивается как индикатор нормальных биологических процессов, патогенных процессов или фармакологических реакций на терапевтическое вмешательство». [59] [60]
Понимание протеома, структуры и функции каждого белка и сложности белок-белковых взаимодействий имеют решающее значение для разработки наиболее эффективных диагностических методов и методов лечения заболеваний в будущем. Например, протеомика очень полезна для идентификации кандидатов на биомаркеры (белки в жидкостях организма, которые имеют ценность для диагностики), идентификации бактериальных антигенов, на которые направлен иммунный ответ, и идентификации возможных иммуногистохимических маркеров инфекционных или неопластических заболеваний. [61]
Интересным применением протеомики является использование специфических белковых биомаркеров для диагностики заболеваний. Ряд методов позволяет тестировать белки, вырабатываемые во время определенного заболевания, что помогает быстро диагностировать заболевание. Методы включают вестерн-блот , иммуногистохимическое окрашивание , иммуноферментный анализ (ELISA) или масс-спектрометрию . [40] [62] Секретомика , подраздел протеомики, изучающий секретируемые белки и пути секреции с использованием протеомных подходов, недавно стала важным инструментом для обнаружения биомаркеров заболеваний. [63]
В протеогеномике протеомные технологии, такие как масс-спектрометрия, используются для улучшения аннотаций генов . Параллельный анализ генома и протеома облегчает обнаружение посттрансляционных модификаций и протеолитических событий, [64] особенно при сравнении нескольких видов (сравнительная протеогеномика). [65]
Структурная протеомика включает анализ белковых структур в больших масштабах. Она сравнивает белковые структуры и помогает идентифицировать функции недавно открытых генов. Структурный анализ также помогает понять, где лекарства связываются с белками, а также показывает, где белки взаимодействуют друг с другом. Это понимание достигается с помощью различных технологий, таких как рентгеновская кристаллография и ЯМР-спектроскопия. [58]
Многие данные протеомики собираются с помощью высокопроизводительных технологий, таких как масс-спектрометрия и микрочипы. Часто для анализа данных и выполнения сравнений вручную требуются недели или месяцы. По этой причине биологи и химики сотрудничают с компьютерщиками и математиками для создания программ и конвейеров для вычислительного анализа данных о белках. Используя методы биоинформатики , исследователи способны выполнять более быстрый анализ и хранение данных. Хорошее место для поиска списков текущих программ и баз данных — портал биоинформатических ресурсов ExPASy . Приложения протеомики на основе биоинформатики включают медицину, диагностику заболеваний, идентификацию биомаркеров и многое другое.
Масс-спектрометрия и микрочипы выдают информацию о фрагментации пептидов, но не дают идентификацию конкретных белков, присутствующих в исходном образце. Из-за отсутствия конкретной идентификации белков, прошлые исследователи были вынуждены расшифровывать сами фрагменты пептидов. Однако в настоящее время существуют программы для идентификации белков. Эти программы берут выходные данные пептидных последовательностей из масс-спектрометрии и микрочипа и возвращают информацию о соответствующих или похожих белках. Это делается с помощью алгоритмов, реализованных программой, которые выполняют выравнивание с белками из известных баз данных, таких как UniProt [66] и PROSITE [67], чтобы предсказать, какие белки находятся в образце с определенной степенью уверенности.
Биомолекулярная структура формирует трехмерную конфигурацию белка. Понимание структуры белка помогает в идентификации взаимодействий и функций белка. Раньше считалось, что трехмерную структуру белков можно было определить только с помощью рентгеновской кристаллографии и ЯМР-спектроскопии . По состоянию на 2017 год криоэлектронная микроскопия является ведущей техникой, решающей трудности с кристаллизацией (в рентгеновской кристаллографии) и конформационной неоднозначностью (в ЯМР); разрешение составляло 2,2Å по состоянию на 2015 год. Теперь, благодаря биоинформатике, существуют компьютерные программы, которые в некоторых случаях могут предсказывать и моделировать структуру белков. Эти программы используют химические свойства аминокислот и структурные свойства известных белков для прогнозирования трехмерной модели образцов белков. Это также позволяет ученым моделировать взаимодействия белков в большем масштабе. Кроме того, биомедицинские инженеры разрабатывают методы, учитывающие гибкость структур белков для проведения сравнений и прогнозов. [68]
Большинство программ, доступных для анализа белков, не написаны для белков, которые подверглись посттрансляционным модификациям . [69] Некоторые программы принимают посттрансляционные модификации для помощи в идентификации белков, но затем игнорируют модификации во время дальнейшего анализа белков. Важно учитывать эти модификации, поскольку они могут влиять на структуру белка. В свою очередь, вычислительный анализ посттрансляционных модификаций привлек внимание научного сообщества. Текущие программы посттрансляционной модификации являются только предиктивными. [70] Химики, биологи и компьютерные специалисты работают вместе, чтобы создать и внедрить новые конвейеры, которые позволяют анализировать посттрансляционные модификации, которые были экспериментально идентифицированы по их влиянию на структуру и функцию белка.
Одним из примеров использования биоинформатики и использования вычислительных методов является изучение белковых биомаркеров. Вычислительные прогностические модели [71] показали, что обширный и разнообразный фето-материнский трафик белков происходит во время беременности и может быть легко обнаружен неинвазивным способом в цельной материнской крови. Этот вычислительный подход обошел главное ограничение, обилие материнских белков, мешающих обнаружению фетальных белков , для фетального протеомного анализа материнской крови. Вычислительные модели могут использовать транскрипты фетальных генов, ранее идентифицированные в цельной материнской крови , для создания всеобъемлющей протеомной сети термина новорожденный . Такая работа показывает, что фетальные белки, обнаруженные в крови беременной женщины, происходят из разнообразной группы тканей и органов развивающегося плода. Протеомные сети содержат множество биомаркеров , которые являются прокси для развития и иллюстрируют потенциальное клиническое применение этой технологии как способа мониторинга нормального и аномального развития плода.
Также была введена информационно-теоретическая структура для обнаружения биомаркеров , интегрирующая информацию о биожидкостях и тканях. [72] Этот новый подход использует преимущества функциональной синергии между определенными биожидкостями и тканями с потенциалом для клинически значимых результатов, которые были бы невозможны, если бы ткани и биожидкости рассматривались по отдельности. Концептуализируя ткань-биожидкости как информационные каналы, можно идентифицировать значимые прокси биожидкостей и затем использовать их для направленной разработки клинической диагностики. Затем потенциальные биомаркеры прогнозируются на основе критериев передачи информации по каналам ткань-биожидкости. Значимые взаимосвязи биожидкости и ткани могут использоваться для приоритизации клинической валидации биомаркеров. [72]
Ряд новых концепций имеют потенциал для улучшения текущих возможностей протеомики. Получение абсолютной количественной оценки белков и мониторинг посттрансляционных модификаций являются двумя задачами, которые влияют на понимание функции белков в здоровых и больных клетках. Кроме того, пропускная способность и чувствительность протеомных анализов, часто измеряемые как образцы, анализируемые в день, и глубина покрытия протеома, соответственно, привели к разработке передовых инструментов и методологий. [73] Для многих клеточных событий концентрации белков не изменяются; скорее, их функция модулируется посттрансляционными модификациями (PTM). Методы мониторинга PTM являются недостаточно развитой областью в протеомике. Выбор определенного подмножества белков для анализа существенно снижает сложность белков, что делает его выгодным для диагностических целей, где кровь является исходным материалом. Другим важным аспектом протеомики, который пока не рассмотрен, является то, что методы протеомики должны быть сосредоточены на изучении белков в контексте окружающей среды. Растущее использование химических сшивающих агентов, вводимых в живые клетки для фиксации белок-белковых, белок-ДНК и других взаимодействий, может частично облегчить эту проблему. Задача состоит в том, чтобы определить подходящие методы сохранения соответствующих взаимодействий. Другая цель изучения белков — разработка более сложных методов для визуализации белков и других молекул в живых клетках и в реальном времени. [42]
Достижения в количественной протеомике, очевидно, позволят проводить более глубокий анализ клеточных систем. [56] [57] Другим направлением исследований является анализ отдельных клеток, [74] [75] и ковариации белков между отдельными клетками [76] , что отражает биологические процессы, такие как образование белковых комплексов, иммунные функции, [77] , а также клеточный цикл и подготовка раковых клеток к лекарственной устойчивости [78] Биологические системы подвержены различным возмущениям ( клеточный цикл , клеточная дифференциация , канцерогенез , окружающая среда (биофизическая) и т. д.). Транскрипционные и трансляционные ответы на эти возмущения приводят к функциональным изменениям в протеоме, вовлеченным в ответ на стимул. Поэтому описание и количественная оценка изменений в протеомном изобилии белков имеет решающее значение для более целостного понимания биологического явления на уровне всей системы. Таким образом, протеомику можно рассматривать как дополнение к геномике , транскриптомике , эпигеномике , метаболомике и другим подходам -omics в интегративном анализе, пытающемся более полно определить биологические фенотипы . Например, The Cancer Proteome Atlas предоставляет количественные данные об экспрессии белков для ~200 белков в более чем 4000 образцах опухолей с соответствующими транскриптомными и геномными данными из The Cancer Genome Atlas . [79] Аналогичные наборы данных в других типах клеток, типах тканей и видах, особенно с использованием глубокой дробовой масс-спектрометрии, будут чрезвычайно важным ресурсом для исследований в таких областях, как биология рака , биология развития и стволовых клеток , медицина и эволюционная биология .
Характеристика протеома плазмы человека стала главной целью в области протеомики, но это также самый сложный протеом из всех тканей человека. [80] Он содержит иммуноглобулин, цитокины, белковые гормоны и секретируемые белки, указывающие на инфекцию, помимо резидентных, гемостатических белков. Он также содержит белки утечки тканей из-за циркуляции крови через различные ткани в организме. Таким образом, кровь содержит информацию о физиологическом состоянии всех тканей и, в сочетании с ее доступностью, делает протеом крови бесценным для медицинских целей. Считается, что характеристика протеома плазмы крови является сложной задачей.
Глубина протеома плазмы охватывает динамический диапазон более 10 10 между самым распространенным белком (альбумин) и самым низким (некоторые цитокины) и считается одной из основных проблем для протеомики. [81] Временная и пространственная динамика еще больше усложняет изучение протеома плазмы человека. Оборот некоторых белков происходит гораздо быстрее, чем других, и содержание белка в артерии может существенно отличаться от содержания в вене. Все эти различия делают даже самую простую протеомную задачу каталогизации протеома недостижимой. Чтобы решить эту проблему, необходимо установить приоритеты. Одним из таких приоритетов является захват наиболее значимого подмножества белков среди всего протеома для создания диагностического инструмента. Во-вторых, поскольку рак связан с повышенным гликозилированием белков, методы, которые фокусируются на этой части белков, также будут полезны. Опять же: многопараметрический анализ лучше всего выявляет патологическое состояние. По мере совершенствования этих технологий профили заболеваний должны постоянно соотноситься с соответствующими изменениями экспрессии генов. [42] Из-за вышеупомянутых проблем плазменная протеомика оставалась сложной. Однако технологические достижения и постоянные разработки, похоже, приводят к возрождению плазменной протеомики, как это было недавно продемонстрировано технологией, называемой профилированием плазменного протеома. [82] Благодаря таким технологиям исследователи смогли изучить воспалительные процессы у мышей, наследуемость плазменных протеомов, а также показать влияние такого распространенного изменения образа жизни, как потеря веса, на плазменный протеом. [83] [84] [85]
Многочисленные журналы посвящены области протеомики и смежным областям. Обратите внимание, что журналы, посвященные белкам, обычно больше сосредоточены на структуре и функции, в то время как журналы по протеомике больше сосредоточены на крупномасштабном анализе целых протеомов или, по крайней мере, больших наборов белков. Некоторые соответствующие журналы по протеомике перечислены ниже (с их издателями).
{{cite book}}: |journal=проигнорировано ( помощь )