Секвенирование ДНК — это процесс определения последовательности нуклеиновой кислоты — порядка нуклеотидов в ДНК . Он включает в себя любой метод или технологию, которая используется для определения порядка четырех оснований: аденина , гуанина , цитозина и тимина . Появление методов быстрого секвенирования ДНК значительно ускорило биологические и медицинские исследования и открытия. [1] [2]
Знание последовательностей ДНК стало незаменимым для фундаментальных биологических исследований, ДНК-генографических проектов и в многочисленных прикладных областях, таких как медицинская диагностика , биотехнология , судебная биология , вирусология и биологическая систематика . Сравнение здоровых и мутировавших последовательностей ДНК может диагностировать различные заболевания, включая различные виды рака, [3] характеризовать репертуар антител, [4] и может использоваться для руководства лечением пациентов. [5] Наличие быстрого способа секвенирования ДНК позволяет быстрее и более индивидуально оказывать медицинскую помощь, а также идентифицировать и каталогизировать больше организмов. [4]
Высокая скорость секвенирования, достигнутая с помощью современной технологии секвенирования ДНК, сыграла важную роль в секвенировании полных последовательностей ДНК, или геномов , многочисленных типов и видов жизни, включая геном человека и другие полные последовательности ДНК многих видов животных, растений и микроорганизмов.

Первые последовательности ДНК были получены в начале 1970-х годов академическими исследователями с использованием трудоемких методов, основанных на двумерной хроматографии . После разработки методов секвенирования на основе флуоресценции с использованием секвенатора ДНК , [6] секвенирование ДНК стало проще и на порядки быстрее. [7] [8]
Секвенирование ДНК может использоваться для определения последовательности отдельных генов , более крупных генетических областей (т. е. кластеров генов или оперонов ), полных хромосом или целых геномов любого организма. Секвенирование ДНК также является наиболее эффективным способом косвенного секвенирования РНК или белков (через их открытые рамки считывания ). Фактически, секвенирование ДНК стало ключевой технологией во многих областях биологии и других наук, таких как медицина, судебная экспертиза и антропология .
Секвенирование используется в молекулярной биологии для изучения геномов и кодируемых ими белков. Информация, полученная с помощью секвенирования, позволяет исследователям выявлять изменения в генах и некодирующей ДНК (включая регуляторные последовательности), ассоциации с заболеваниями и фенотипами, а также определять потенциальные мишени для лекарственных препаратов.
Поскольку ДНК является информативной макромолекулой с точки зрения передачи от одного поколения к другому, секвенирование ДНК используется в эволюционной биологии для изучения того, как связаны между собой различные организмы и как они эволюционировали. В феврале 2021 года ученые впервые сообщили о секвенировании ДНК из останков животных , в данном случае мамонта , возрастом более миллиона лет, что является старейшей секвенированной ДНК на сегодняшний день. [9] [10]
Область метагеномики включает в себя идентификацию организмов, присутствующих в водоеме, сточных водах , грязи, отфильтрованном из воздуха мусоре или образцах мазков от организмов. Знание того, какие организмы присутствуют в определенной среде, имеет решающее значение для исследований в области экологии , эпидемиологии , микробиологии и других областях. Например, секвенирование позволяет исследователям определить, какие типы микробов могут присутствовать в микробиоме .
Поскольку большинство вирусов слишком малы, чтобы их можно было увидеть с помощью светового микроскопа, секвенирование является одним из основных инструментов в вирусологии для идентификации и изучения вируса. [11] Вирусные геномы могут быть основаны на ДНК или РНК. РНК-вирусы более чувствительны ко времени для секвенирования генома, поскольку они быстрее деградируют в клинических образцах. [12] Традиционное секвенирование по Сэнгеру и секвенирование следующего поколения используются для секвенирования вирусов в фундаментальных и клинических исследованиях, а также для диагностики возникающих вирусных инфекций, молекулярной эпидемиологии вирусных патогенов и тестирования устойчивости к лекарственным препаратам. В GenBank содержится более 2,3 миллиона уникальных вирусных последовательностей . [11] Недавно NGS превзошел традиционный метод Сэнгера как самый популярный подход к генерации вирусных геномов. [11]
Во время вспышки птичьего гриппа 1997 года вирусное секвенирование определило, что подтип гриппа возник в результате реассортации между перепелами и домашней птицей. Это привело к принятию в Гонконге законодательства , запрещающего продажу живых перепелов и домашней птицы вместе на рынке. Вирусное секвенирование также может быть использовано для оценки начала вирусной вспышки с помощью метода молекулярных часов . [12]
Медицинские техники могут секвенировать гены (или, теоретически, полные геномы) пациентов, чтобы определить, есть ли риск генетических заболеваний. Это форма генетического тестирования , хотя некоторые генетические тесты могут не включать секвенирование ДНК.
С 2013 года секвенирование ДНК все чаще используется для диагностики и лечения редких заболеваний. Поскольку все больше и больше генов идентифицируются, вызывающих редкие генетические заболевания, молекулярная диагностика для пациентов становится все более распространенной. Секвенирование ДНК позволяет врачам выявлять генетические заболевания, улучшать управление заболеваниями, предоставлять репродуктивное консультирование и более эффективную терапию. [13] Панели секвенирования генов используются для выявления множественных потенциальных генетических причин предполагаемого расстройства. [14]
Кроме того, секвенирование ДНК может быть полезным для определения конкретных бактерий, что позволит проводить более точное лечение антибиотиками , тем самым снижая риск возникновения устойчивости к противомикробным препаратам в популяциях бактерий. [15] [16] [17] [18] [19] [20]
Секвенирование ДНК может использоваться вместе с методами профилирования ДНК для судебной идентификации [21] и тестирования отцовства . Тестирование ДНК значительно развилось за последние несколько десятилетий, чтобы в конечном итоге связать отпечаток ДНК с тем, что находится под следствием. Модели ДНК в отпечатках пальцев, слюне, волосяных фолликулах и т. д. уникально отделяют каждый живой организм от другого. Тестирование ДНК — это метод, который может обнаружить определенные геномы в цепи ДНК для создания уникального и индивидуального рисунка.
Секвенирование ДНК может использоваться вместе с методами профилирования ДНК для судебной идентификации и тестирования отцовства , поскольку оно значительно развилось за последние несколько десятилетий, чтобы в конечном итоге связать отпечаток ДНК с тем, что находится под следствием. Образцы ДНК в отпечатках пальцев, слюне, волосяных фолликулах и других жидкостях организма уникально отделяют каждый живой организм от другого, что делает его бесценным инструментом в области судебной экспертизы . Процесс тестирования ДНК включает обнаружение определенных геномов в цепи ДНК для создания уникального и индивидуального образца, который может быть использован для идентификации людей или определения их отношений.
Достижения в области технологии секвенирования ДНК позволили быстро и точно анализировать и сравнивать большие объемы генетических данных , что позволяет следователям собирать доказательства и раскрывать преступления более эффективно. Эта технология использовалась в различных приложениях, включая судебную идентификацию, тестирование отцовства и идентификацию человека в случаях, когда традиционные методы идентификации недоступны или ненадежны. Использование секвенирования ДНК также привело к разработке новых методов судебной экспертизы, таких как фенотипирование ДНК , которое позволяет следователям предсказывать физические характеристики человека на основе его генетических данных.
Помимо применения в судебной науке, секвенирование ДНК также использовалось в медицинских исследованиях и диагностике. Оно позволило ученым идентифицировать генетические мутации и вариации, связанные с определенными заболеваниями и расстройствами, что позволило проводить более точную диагностику и целенаправленное лечение. Кроме того, секвенирование ДНК также использовалось в природоохранной биологии для изучения генетического разнообразия исчезающих видов и разработки стратегий их сохранения.
Кроме того, использование секвенирования ДНК также подняло важные этические и правовые вопросы. Например, существуют опасения по поводу конфиденциальности и безопасности генетических данных, а также возможности злоупотребления или дискриминации на основе генетической информации. В результате продолжаются дебаты о необходимости правил и руководств для обеспечения ответственного использования технологии секвенирования ДНК.
В целом, развитие технологии секвенирования ДНК произвело революцию в области судебной экспертизы и имеет далеко идущие последствия для нашего понимания генетики, медицины и биологии сохранения.
Каноническая структура ДНК имеет четыре основания: тимин (T), аденин (A), цитозин (C) и гуанин (G). Секвенирование ДНК — это определение физического порядка этих оснований в молекуле ДНК. Однако в молекуле может присутствовать множество других оснований. В некоторых вирусах (в частности, бактериофаге ) цитозин может быть заменен гидроксиметилом или гидроксиметилглюкозой цитозином. [22] В ДНК млекопитающих могут быть обнаружены вариантные основания с метильными группами или фосфосульфатом. [23] [24] В зависимости от метода секвенирования может быть обнаружена или не обнаружена конкретная модификация, например, 5mC ( 5-метилцитозин ), распространенная у людей. [25]
Почти во всех организмах ДНК синтезируется in vivo с использованием только 4 канонических оснований; модификация, которая происходит после репликации, создает другие основания, такие как 5 метил C. Однако некоторые бактериофаги могут напрямую включать нестандартное основание. [26]
В дополнение к модификациям, ДНК постоянно подвергается атакам со стороны агентов окружающей среды, таких как ультрафиолет и радикалы кислорода. В настоящее время наличие таких поврежденных оснований не обнаруживается большинством методов секвенирования ДНК, хотя PacBio опубликовала об этом. [27]
Дезоксирибонуклеиновая кислота ( ДНК ) была впервые обнаружена и выделена Фридрихом Мишером в 1869 году, но она оставалась недостаточно изученной в течение многих десятилетий, поскольку считалось, что белки, а не ДНК, хранят генетический план жизни. Эта ситуация изменилась после 1944 года в результате некоторых экспериментов Освальда Эвери , Колина Маклеода и Маклин Маккарти, продемонстрировавших, что очищенная ДНК может изменять один штамм бактерий в другой. Это был первый случай, когда было показано, что ДНК способна преобразовывать свойства клеток.
В 1953 году Джеймс Уотсон и Фрэнсис Крик выдвинули свою модель двойной спирали ДНК, основанную на кристаллизованных рентгеновских структурах, которые изучала Розалинд Франклин . Согласно модели, ДНК состоит из двух нитей нуклеотидов, скрученных вокруг друг друга, связанных вместе водородными связями и идущих в противоположных направлениях. Каждая нить состоит из четырех комплементарных нуклеотидов — аденина (A), цитозина (C), гуанина (G) и тимина (T) — с A на одной нити всегда в паре с T на другой, а C всегда в паре с G. Они предположили, что такая структура позволяет использовать каждую нить для реконструкции другой, идея, имеющая центральное значение для передачи наследственной информации между поколениями. [28]

Основа для секвенирования белков была впервые заложена работой Фредерика Сэнгера , который к 1955 году завершил последовательность всех аминокислот в инсулине , небольшом белке, секретируемом поджелудочной железой. Это дало первое убедительное доказательство того, что белки являются химическими образованиями с определенным молекулярным рисунком, а не случайной смесью материалов, взвешенных в жидкости. Успех Сэнгера в секвенировании инсулина подстегнул рентгеновских кристаллографов, включая Уотсона и Крика, которые к тому времени пытались понять, как ДНК направляет образование белков внутри клетки. Вскоре после посещения серии лекций, прочитанных Фредериком Сэнгером в октябре 1954 года, Крик начал разрабатывать теорию, которая утверждала, что расположение нуклеотидов в ДНК определяет последовательность аминокислот в белках, что, в свою очередь, помогает определить функцию белка. Он опубликовал эту теорию в 1958 году. [29]
Секвенирование РНК было одной из самых ранних форм секвенирования нуклеотидов. Главной вехой секвенирования РНК является последовательность первого полного гена и полного генома бактериофага MS2 , идентифицированного и опубликованного Уолтером Фирсом и его коллегами в Университете Гента ( Гент , Бельгия ) в 1972 [30] и 1976 годах . [31] Традиционные методы секвенирования РНК требуют создания молекулы кДНК , которая должна быть секвенирована. [32]
Традиционные методы секвенирования РНК
Традиционные методы секвенирования РНК включают несколько этапов:
1) Обратная транскрипция : первым шагом является преобразование молекулы РНК в комплементарную молекулу ДНК (кДНК) с помощью фермента, называемого обратной транскриптазой .
2) Синтез кДНК : молекула кДНК затем синтезируется с помощью процесса, называемого ПЦР ( полимеразная цепная реакция ), который амплифицирует кДНК для получения множественных копий.
3) Секвенирование : амплифицированная кДНК затем секвенируется с использованием таких методов, как секвенирование по Сэнгеру или секвенирование по Максаму-Гилберту .
Проблемы и ограничения
Традиционные методы секвенирования РНК имеют ряд ограничений. Например:
Они требуют создания молекулы кДНК, что может быть трудоемким и занимать много времени. Они подвержены ошибкам и предубеждениям, которые могут повлиять на точность результатов секвенирования. Они ограничены в своей способности обнаруживать редкие или малораспространенные транскрипты. Достижения в технологии секвенирования РНК
В последние годы достижения в технологии секвенирования РНК устранили некоторые из этих ограничений. Новые методы, такие как секвенирование следующего поколения (NGS) и секвенирование одиночной молекулы в реальном времени (SMRT), позволили проводить более быстрое, точное и экономически эффективное секвенирование молекул РНК. Эти достижения открыли новые возможности для изучения экспрессии генов, идентификации новых генов и понимания регуляции экспрессии генов.
Первый метод определения последовательностей ДНК включал стратегию удлинения праймера, специфичную для определенного местоположения, разработанную Рэем Ву в Корнелльском университете в 1970 году. [33] Катализ ДНК-полимеразы и специфическая маркировка нуклеотидов, оба из которых занимают видное место в современных схемах секвенирования, использовались для секвенирования липких концов ДНК фага лямбда. [34] [35] [36] В период с 1970 по 1973 год Ву, Р. Падманабхан и коллеги продемонстрировали, что этот метод можно использовать для определения любой последовательности ДНК с использованием синтетических праймеров, специфичных для определенного местоположения. [37] [38] [8] Затем Фредерик Сэнгер принял эту стратегию удлинения праймера для разработки более быстрых методов секвенирования ДНК в Центре MRC , Кембридж , Великобритания, и опубликовал метод «секвенирования ДНК с ингибиторами, обрывающими цепь» в 1977 году. [39] Уолтер Гилберт и Аллан Максам из Гарварда также разработали методы секвенирования, в том числе один для «секвенирования ДНК путем химической деградации». [40] [41] В 1973 году Гилберт и Максам сообщили о последовательности из 24 пар оснований с использованием метода, известного как анализ блуждающих пятен. [42] Достижения в области секвенирования были обусловлены параллельной разработкой технологии рекомбинантной ДНК , позволяющей изолировать образцы ДНК из источников, отличных от вирусов.

Первый полный ДНК-геном, который был секвенирован, был у бактериофага φX174 в 1977 году. [43] Ученые Медицинского исследовательского совета расшифровали полную последовательность ДНК вируса Эпштейна-Барр в 1984 году, обнаружив, что она содержит 172 282 нуклеотида. Завершение последовательности ознаменовало собой значительный поворотный момент в секвенировании ДНК, поскольку оно было достигнуто без предварительного знания генетического профиля вируса. [44] [8]
Нерадиоактивный метод переноса молекул ДНК из смесей для секвенирования реакции на иммобилизующую матрицу во время электрофореза был разработан Гербертом Полем и его коллегами в начале 1980-х годов. [45] [46] За этим последовало коммерциализированное производство ДНК-секвенатора «Direct-Blotting-Electrophoresis-System GATC 1500» компанией GATC Biotech , который интенсивно использовался в рамках программы ЕС по секвенированию генома, и была получена полная последовательность ДНК хромосомы II дрожжей Saccharomyces cerevisiae . [47] Лаборатория Лероя Э. Худа в Калифорнийском технологическом институте объявила о первой полуавтоматической машине для секвенирования ДНК в 1986 году. [48] За этим последовал маркетинг Applied Biosystems первой полностью автоматизированной машины для секвенирования, ABI 370, в 1987 году и Genesis 2000 от Dupont [49] , которая использовала новую технику флуоресцентной маркировки, позволяющую идентифицировать все четыре дидезоксинуклеотида на одной полосе. К 1990 году Национальные институты здравоохранения США (NIH) начали крупномасштабные испытания по секвенированию на Mycoplasma capricolum , Escherichia coli , Caenorhabditis elegans и Saccharomyces cerevisiae по цене 0,75 долл. США за основание. Тем временем в лаборатории Крейга Вентера началось секвенирование последовательностей человеческой ДНК , называемых экспрессируемыми метками последовательностей , в попытке захватить кодирующую фракцию человеческого генома . [50] В 1995 году Вентер, Гамильтон Смит и коллеги из Института геномных исследований (TIGR) опубликовали первый полный геном свободноживущего организма, бактерии Haemophilus influenzae . Кольцевая хромосома содержит 1 830 137 оснований, и ее публикация в журнале Science [51] ознаменовала первое опубликованное использование полногеномного дробового секвенирования, что исключило необходимость в первоначальных усилиях по картированию.
К 2001 году методы дробового секвенирования были использованы для получения черновой версии последовательности человеческого генома. [52] [53]

Несколько новых методов секвенирования ДНК были разработаны в середине-конце 1990-х годов и были реализованы в коммерческих секвенаторах ДНК к 2000 году. Вместе они были названы методами секвенирования «следующего поколения» или «второго поколения» (NGS), чтобы отличать их от более ранних методов, включая секвенирование по Сэнгеру . В отличие от первого поколения секвенирования, технология NGS обычно характеризуется высокой масштабируемостью, что позволяет секвенировать весь геном за один раз. Обычно это достигается путем фрагментации генома на небольшие части, случайного отбора фрагмента и его секвенирования с использованием одной из множества технологий, таких как описанные ниже. Весь геном возможен, поскольку несколько фрагментов секвенируются одновременно (что дает ему название «массово-параллельное» секвенирование) в автоматизированном процессе.
Технология NGS чрезвычайно расширила возможности исследователей в поиске информации о здоровье, антропологов в исследовании происхождения человека и является катализатором движения « Персонализированная медицина ». Однако она также открыла двери для большего количества возможностей для ошибок. Существует множество программных инструментов для проведения вычислительного анализа данных NGS, часто составленных на онлайн-платформах, таких как CSI NGS Portal, каждый из которых имеет свой собственный алгоритм. Даже параметры в пределах одного программного пакета могут изменить результат анализа. Кроме того, большие объемы данных, полученных путем секвенирования ДНК, также потребовали разработки новых методов и программ для анализа последовательностей. Было предпринято несколько попыток разработать стандарты в области NGS для решения этих проблем, большинство из которых были мелкомасштабными усилиями, возникшими в отдельных лабораториях. Совсем недавно крупные, организованные, финансируемые FDA усилия увенчались стандартом BioCompute .
26 октября 1990 года Роджер Циен , Пепи Росс, Маргарет Фанесток и Аллан Дж. Джонстон подали патент, описывающий пошаговое («основание за основанием») секвенирование с удаляемыми 3'-блокаторами на ДНК-матрицах (блоты и отдельные молекулы ДНК). [55] В 1996 году Пол Нюрен и его студент Мостафа Ронаги в Королевском технологическом институте в Стокгольме опубликовали свой метод пиросеквенирования . [56]
1 апреля 1997 года Паскаль Майер и Лоран Фаринелли подали патенты во Всемирную организацию интеллектуальной собственности, описывающие секвенирование колоний ДНК. [57] Методы подготовки образцов ДНК и случайной поверхностной полимеразной цепной реакции (ПЦР), описанные в этом патенте, в сочетании с методом секвенирования «основание за основанием» Роджера Циена и др., теперь реализованы в геномных секвенаторах Hi-Seq компании Illumina .
В 1998 году Фил Грин и Брент Юинг из Вашингтонского университета описали свою оценку качества phred для анализа данных секвенатора [58] , знаменательную методику анализа, которая получила широкое распространение и которая до сих пор является наиболее распространенной метрикой для оценки точности платформы секвенирования. [59]
Lynx Therapeutics опубликовала и вывела на рынок метод массового параллельного секвенирования сигнатур (MPSS) в 2000 году. Этот метод включал в себя параллельную технологию секвенирования на основе адаптера/лигирования с использованием гранул и стал первым коммерчески доступным методом секвенирования «следующего поколения», хотя ни один ДНК-секвенатор не был продан независимым лабораториям. [60]
Аллан Максам и Уолтер Гилберт опубликовали метод секвенирования ДНК в 1977 году, основанный на химической модификации ДНК и последующем расщеплении по определенным основаниям. [40] Этот метод, также известный как химическое секвенирование, позволял использовать очищенные образцы двухцепочечной ДНК без дальнейшего клонирования. Использование радиоактивной маркировки в этом методе и его техническая сложность препятствовали широкому использованию после усовершенствования методов Сэнгера.
Секвенирование Максама-Гилберта требует радиоактивной маркировки на одном 5'-конце ДНК и очистки фрагмента ДНК, который будет секвенирован. Затем химическая обработка создает разрывы в небольшой пропорции одного или двух из четырех нуклеотидных оснований в каждой из четырех реакций (G, A+G, C, C+T). Концентрация модифицирующих химикатов контролируется для введения в среднем одной модификации на молекулу ДНК. Таким образом, генерируется серия меченых фрагментов, от радиоактивно меченого конца до первого «разрезанного» участка в каждой молекуле. Фрагменты в четырех реакциях электрофорезируются бок о бок в денатурирующих акриламидных гелях для разделения по размеру. Для визуализации фрагментов гель экспонируется на рентгеновской пленке для авторадиографии, что дает серию темных полос, каждая из которых соответствует радиоактивно меченому фрагменту ДНК, из которого можно вывести последовательность. [40]
Этот метод в основном устарел по состоянию на 2023 год. [61]
Метод обрыва цепи, разработанный Фредериком Сэнгером и его коллегами в 1977 году, вскоре стал методом выбора из-за его относительной простоты и надежности. [39] [62] После изобретения метод обрыва цепи использовал меньше токсичных химикатов и меньшее количество радиоактивности, чем метод Максама и Гилберта. Благодаря своей сравнительной простоте метод Сэнгера вскоре был автоматизирован и использовался в первом поколении секвенаторов ДНК .
Секвенирование по Сэнгеру — метод, который преобладал с 1980-х до середины 2000-х годов. За этот период в технике были достигнуты большие успехи, такие как флуоресцентная маркировка, капиллярный электрофорез и общая автоматизация. Эти разработки позволили проводить гораздо более эффективное секвенирование, что привело к снижению затрат. Метод Сэнгера в форме массового производства — это технология, которая произвела первый геном человека в 2001 году, открыв эру геномики . Однако позднее в этом десятилетии на рынок вышли радикально иные подходы, в результате чего стоимость генома снизилась со 100 миллионов долларов в 2001 году до 10 000 долларов в 2011 году. [63]
Целью последовательного секвенирования синтезом (SBS) является определение последовательности образца ДНК путем обнаружения включения нуклеотида ДНК - полимеразой . Сконструированная полимераза используется для синтеза копии одной цепи ДНК, и включение каждого нуклеотида отслеживается. Принцип секвенирования в реальном времени синтезом был впервые описан в 1993 году [64] с улучшениями, опубликованными несколько лет спустя. [65] Ключевые части очень похожи для всех вариантов SBS и включают в себя (1) амплификацию ДНК (для усиления последующего сигнала) и прикрепление ДНК, подлежащей секвенированию, к твердой подложке, (2) генерацию одноцепочечной ДНК на твердой подложке, (3) включение нуклеотидов с использованием сконструированной полимеразы и (4) обнаружение включения нуклеотида в реальном времени. Шаги 3-4 повторяются, и последовательность собирается из сигналов, полученных на шаге 4. Этот принцип секвенирования в реальном времени путем синтеза использовался почти для всех инструментов массового параллельного секвенирования , включая 454 , PacBio , IonTorrent , Illumina и MGI .
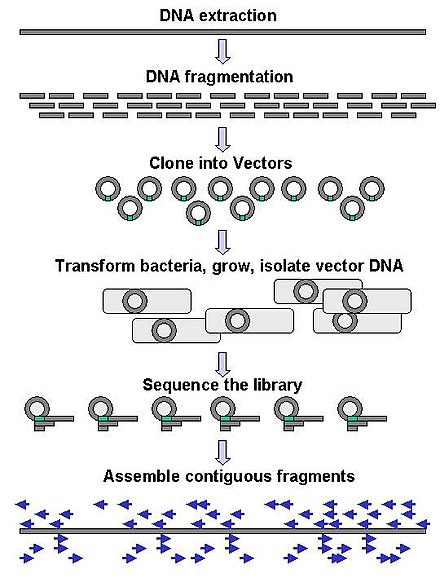
Крупномасштабное секвенирование часто направлено на секвенирование очень длинных фрагментов ДНК, таких как целые хромосомы , хотя крупномасштабное секвенирование может также использоваться для создания очень большого количества коротких последовательностей, таких как обнаруженные в фаговом дисплее . Для более длинных целей, таких как хромосомы, общие подходы состоят из разрезания (с помощью рестриктаз ) или сдвига (с помощью механических сил) больших фрагментов ДНК на более короткие фрагменты ДНК. Фрагментированная ДНК затем может быть клонирована в вектор ДНК и амплифицирована в бактериальном хозяине, таком как Escherichia coli . Короткие фрагменты ДНК, очищенные от отдельных бактериальных колоний, индивидуально секвенируются и собираются электронным способом в одну длинную непрерывную последовательность. Исследования показали, что добавление шага выбора размера для сбора фрагментов ДНК однородного размера может повысить эффективность секвенирования и точность сборки генома. В этих исследованиях автоматизированное определение размера оказалось более воспроизводимым и точным, чем ручное определение размера геля. [66] [67] [68]
Термин « секвенирование de novo » конкретно относится к методам, используемым для определения последовательности ДНК без ранее известной последовательности. De novo переводится с латыни как «с начала». Пробелы в собранной последовательности могут быть заполнены путем обхода праймера . Различные стратегии имеют разные компромиссы в скорости и точности; методы дробовика часто используются для секвенирования больших геномов, но его сборка сложна и трудна, особенно с повторами последовательностей , часто вызывающими пробелы в сборке генома.
Большинство подходов к секвенированию используют этап клонирования in vitro для амплификации отдельных молекул ДНК, поскольку их методы молекулярного обнаружения недостаточно чувствительны для секвенирования отдельных молекул. Эмульсионная ПЦР [69] изолирует отдельные молекулы ДНК вместе с покрытыми праймером гранулами в водных каплях в масляной фазе. Затем полимеразная цепная реакция (ПЦР) покрывает каждую гранулу клонированными копиями молекулы ДНК с последующей иммобилизацией для последующего секвенирования. Эмульсионная ПЦР используется в методах, разработанных Маргилисом и др. (коммерциализированных 454 Life Sciences ), Шендуре и Поррекой и др. (также известных как « секвенирование полонии ») и секвенирование SOLiD (разработанных Agencourt , позже Applied Biosystems , теперь Life Technologies ). [70] [71] [72] Эмульсионная ПЦР также используется в платформах GemCode и Chromium, разработанных 10x Genomics . [73]
Shotgun-секвенирование — это метод секвенирования, разработанный для анализа последовательностей ДНК длиной более 1000 пар оснований, вплоть до целых хромосом. Этот метод требует, чтобы целевая ДНК была разбита на случайные фрагменты. После секвенирования отдельных фрагментов с использованием метода обрыва цепи последовательности могут быть повторно собраны на основе их перекрывающихся областей. [74]

Высокопроизводительное секвенирование, которое включает методы секвенирования следующего поколения «короткого прочтения» и третьего поколения «длинного прочтения», [nt 1] применяется к секвенированию экзома , секвенированию генома, повторному секвенированию генома, профилированию транскриптома ( РНК-секвенирование ), взаимодействиям ДНК-белок ( ChIP-секвенирование ) и характеристике эпигенома . [75]
Высокий спрос на недорогое секвенирование привел к разработке технологий высокопроизводительного секвенирования, которые распараллеливают процесс секвенирования, производя тысячи или миллионы последовательностей одновременно. [76] [77] [78] Технологии высокопроизводительного секвенирования предназначены для снижения стоимости секвенирования ДНК по сравнению с тем, что возможно при использовании стандартных методов терминации красителем. [79] При сверхвысокопроизводительном секвенировании параллельно может выполняться до 500 000 операций секвенирования путем синтеза. [80] [81] [82] Такие технологии привели к возможности секвенировать весь геном человека всего за один день. [83] По состоянию на 2019 год [обновлять]лидерами корпораций в разработке продуктов для высокопроизводительного секвенирования были Illumina , Qiagen и ThermoFisher Scientific . [83]
Секвенирование SMRT основано на подходе секвенирования путем синтеза. ДНК синтезируется в волноводах нулевой моды (ZMW) — небольших контейнерах, похожих на лунки, с инструментами захвата, расположенными на дне лунки. Секвенирование выполняется с использованием немодифицированной полимеразы (прикрепленной к дну ZMW) и флуоресцентно меченых нуклеотидов, свободно текущих в растворе. Лунки сконструированы таким образом, что обнаруживается только флуоресценция, возникающая у дна лунки. Флуоресцентная метка отделяется от нуклеотида при его включении в цепь ДНК, оставляя немодифицированную цепь ДНК. По данным Pacific Biosciences (PacBio), разработчика технологии SMRT, эта методология позволяет обнаруживать модификации нуклеотидов (такие как метилирование цитозина). Это происходит посредством наблюдения за кинетикой полимеразы. Этот подход позволяет считывать 20 000 нуклеотидов и более со средней длиной считывания 5 килобаз. [90] [100] В 2015 году Pacific Biosciences объявила о запуске нового инструмента для секвенирования под названием Sequel System с 1 миллионом ZMW по сравнению со 150 000 ZMW в инструменте PacBio RS II. [101] [102] Секвенирование SMRT называют секвенированием « третьего поколения » или «длиннокодовым» секвенированием.
ДНК, проходящая через нанопору, изменяет свой ионный ток. Это изменение зависит от формы, размера и длины последовательности ДНК. Каждый тип нуклеотида блокирует поток ионов через пору на разный период времени. Метод не требует модифицированных нуклеотидов и выполняется в режиме реального времени. Секвенирование нанопор называют секвенированием « третьего поколения » или «длиннопрочитанным» секвенированием, наряду с секвенированием SMRT.
Ранние промышленные исследования этого метода основывались на технике, называемой «экзонуклеазное секвенирование», где считывание электрических сигналов происходило, когда нуклеотиды проходили через поры альфа(α)-гемолизина, ковалентно связанные с циклодекстрином . [103] Однако последующий коммерческий метод, «секвенирование нити», секвенировал основания ДНК в неповрежденной нити.
Две основные области нанопорового секвенирования в разработке — это твердотельное нанопоровое секвенирование и белковое нанопоровое секвенирование. Белковое нанопоровое секвенирование использует мембранные белковые комплексы, такие как α-гемолизин, MspA ( Mycobacterium smegmatis Porin A) или CssG, которые показывают большие перспективы, учитывая их способность различать отдельные нуклеотиды и группы. [104] Напротив, твердотельное нанопоровое секвенирование использует синтетические материалы, такие как нитрид кремния и оксид алюминия, и оно является предпочтительным из-за его превосходной механической способности и термической и химической стабильности. [105] Метод изготовления имеет важное значение для этого типа секвенирования, учитывая, что массив нанопор может содержать сотни пор с диаметром менее восьми нанометров. [104]
Концепция возникла из идеи, что одноцепочечные молекулы ДНК или РНК могут быть электрофоретически направлены в строгой линейной последовательности через биологическую пору, которая может быть меньше восьми нанометров, и могут быть обнаружены, учитывая, что молекулы высвобождают ионный ток при движении через пору. Пора содержит область обнаружения, способную распознавать различные основания, причем каждое основание генерирует различные сигналы, зависящие от времени, соответствующие последовательности оснований, когда они пересекают пору, которые затем оцениваются. [105] Точный контроль над транспортом ДНК через пору имеет решающее значение для успеха. Различные ферменты, такие как экзонуклеазы и полимеразы, использовались для смягчения этого процесса, размещая их вблизи входа в пору. [106]
Первая из высокопроизводительных технологий секвенирования, массовое параллельное сигнатурное секвенирование (или MPSS, также называемое секвенированием следующего поколения), была разработана в 1990-х годах в Lynx Therapeutics, компании, основанной в 1992 году Сидни Бреннером и Сэмом Элетром . MPSS был методом на основе бусин, который использовал сложный подход лигирования адаптеров с последующим декодированием адаптеров, считывая последовательность с шагом в четыре нуклеотида. Этот метод делал его восприимчивым к смещению, специфичному для последовательности, или потере определенных последовательностей. Поскольку технология была настолько сложной, MPSS выполнялась только «внутри» Lynx Therapeutics, и ни одно оборудование для секвенирования ДНК не было продано независимым лабораториям. Lynx Therapeutics объединилась с Solexa (позже приобретенной Illumina ) в 2004 году, что привело к разработке секвенирования путем синтеза, более простого подхода, приобретенного у Manteia Predictive Medicine , что сделало MPSS устаревшим. Однако основные свойства выходных данных MPSS были типичны для более поздних типов данных с высокой пропускной способностью, включая сотни тысяч коротких последовательностей ДНК. В случае MPSS они обычно использовались для секвенирования кДНК для измерения уровней экспрессии генов . [60]
Метод секвенирования полоний , разработанный в лаборатории Джорджа М. Чёрча в Гарварде, был одной из первых высокопроизводительных систем секвенирования и использовался для секвенирования полного генома E. coli в 2005 году. [107] Он объединил in vitro библиотеку парных меток с эмульсионной ПЦР, автоматизированным микроскопом и химией секвенирования на основе лигирования для секвенирования генома E. coli с точностью >99,9999% и стоимостью примерно 1/9 от стоимости секвенирования по Сэнгеру. [107] Технология была лицензирована Agencourt Biosciences, впоследствии выделена в Agencourt Personal Genomics и в конечном итоге включена в платформу SOLiD компании Applied Biosystems . Позднее компания Applied Biosystems была приобретена компанией Life Technologies , которая теперь является частью Thermo Fisher Scientific .
Параллельная версия пиросеквенирования была разработана 454 Life Sciences , которая с тех пор была приобретена Roche Diagnostics . Метод амплифицирует ДНК внутри капель воды в масляном растворе (эмульсионная ПЦР), причем каждая капля содержит один шаблон ДНК, прикрепленный к одному покрытому праймером шарику, который затем образует клональную колонию. Секвенирующая машина содержит много лунок объемом пиколитр, каждая из которых содержит один шарик и секвенирующие ферменты. Пиросеквенирование использует люциферазу для генерации света для обнаружения отдельных нуклеотидов, добавленных к зарождающейся ДНК, а объединенные данные используются для генерации считываний последовательности . [70] Эта технология обеспечивает промежуточную длину считывания и цену за основание по сравнению с секвенированием по Сэнгеру с одной стороны и Solexa и SOLiD с другой. [79]
Solexa , теперь часть Illumina , была основана Шанкаром Баласубраманьяном и Дэвидом Кленерманом в 1998 году и разработала метод секвенирования, основанный на технологии обратимых терминаторов красителей и сконструированных полимеразах. [108] Концепция обратимой терминированной химии была изобретена Бруно Канардом и Саймоном Сарфати в Институте Пастера в Париже. [109] [110] Она была разработана внутри Solexa теми, кто указан в соответствующих патентах. В 2004 году Solexa приобрела компанию Manteia Predictive Medicine , чтобы получить технологию массового параллельного секвенирования, изобретенную в 1997 году Паскалем Майером и Лораном Фаринелли. [57] Она основана на «кластерах ДНК» или «колониях ДНК», которая включает клональную амплификацию ДНК на поверхности. Кластерная технология была приобретена совместно с Lynx Therapeutics из Калифорнии. Позднее Solexa Ltd. объединилась с Lynx, образовав Solexa Inc.


В этом методе молекулы ДНК и праймеры сначала прикрепляются к слайду или проточной ячейке и амплифицируются полимеразой, так что образуются локальные клональные колонии ДНК, позже названные «кластерами ДНК». Для определения последовательности добавляются четыре типа обратимых терминаторных оснований (RT-оснований), а невключенные нуклеотиды смываются. Камера делает снимки флуоресцентно меченых нуклеотидов. Затем краситель вместе с терминальным 3'-блокатором химически удаляется из ДНК, что позволяет начать следующий цикл. В отличие от пиросеквенирования, цепи ДНК удлиняются по одному нуклеотиду за раз, и получение изображения может быть выполнено в отложенный момент, что позволяет захватывать очень большие массивы колоний ДНК с помощью последовательных изображений, полученных с одной камеры.

Разделение ферментативной реакции и захвата изображения обеспечивает оптимальную пропускную способность и теоретически неограниченную емкость секвенирования. При оптимальной конфигурации в конечном итоге достижимая пропускная способность инструмента, таким образом, диктуется исключительно скоростью аналого-цифрового преобразования камеры, умноженной на количество камер и деленной на количество пикселей на колонию ДНК, необходимых для их оптимальной визуализации (приблизительно 10 пикселей/колония). В 2012 году с камерами, работающими со скоростью АЦП более 10 МГц и доступной оптикой, струйной техникой и ферментативной техникой, пропускная способность может быть кратна 1 миллиону нуклеотидов/секунду, что примерно соответствует 1 эквиваленту человеческого генома при 1-кратном покрытии в час на инструмент и 1 повторно секвенированному (приблизительно 30-кратному) человеческому геному в день на инструмент (оснащенный одной камерой). [111]
Этот метод является усовершенствованной модификацией комбинаторной технологии лигирования якорных зондов (cPAL), описанной Complete Genomics [112], которая с тех пор стала частью китайской геномной компании BGI в 2013 году. [113] Обе компании усовершенствовали технологию, чтобы обеспечить более длинные считывания, сокращение времени реакции и более быстрое время получения результатов. Кроме того, данные теперь генерируются как смежные полноразмерные считывания в стандартном формате файла FASTQ и могут использоваться как есть в большинстве биоинформатических аналитических конвейеров на основе коротких считываний. [114] [ необходима цитата ]
Две технологии, которые формируют основу для этой высокопроизводительной технологии секвенирования, — это ДНК-наношарики (DNB) и шаблонные массивы для прикрепления наношариков к твердой поверхности. [112] ДНК-наношарики просто формируются путем денатурации двухцепочечных, лигированных адаптером библиотек и лигирования только прямой цепи с олигонуклеотидом-шплинтом для формирования круга одноцепочечной ДНК. Точные копии кругов, содержащих вставку ДНК, производятся с использованием амплификации по вращающемуся кольцу, которая генерирует приблизительно 300–500 копий. Длинная цепь одноцепочечной ДНК складывается сама с собой, образуя трехмерную структуру наношарика диаметром приблизительно 220 нм. Создание DNB заменяет необходимость создания ПЦР-копий библиотеки в проточной ячейке и, таким образом, может удалить большую часть дублирующих прочтений, лигирований адаптер-адаптер и ошибок, вызванных ПЦР. [114] [ необходима цитата ]

Узорчатый массив положительно заряженных пятен изготавливается с помощью фотолитографии и травления, за которыми следует химическая модификация для создания ячейки потока секвенирования. Каждое пятно на ячейке потока имеет диаметр приблизительно 250 нм, отделено друг от друга на 700 нм (от центра до центра) и позволяет легко прикрепить один отрицательно заряженный DNB к ячейке потока и, таким образом, уменьшить недо- или перегруппировку на ячейке потока. [112] [ необходима цитата ]
Затем секвенирование выполняется путем добавления олигонуклеотидного зонда, который прикрепляется в комбинации к определенным участкам в пределах DNB. Зонд действует как якорь, который затем позволяет одному из четырех отдельных обратимо инактивированных, меченых нуклеотидов связываться после протекания через проточную ячейку. Несвязанные нуклеотиды смываются перед лазерным возбуждением прикрепленных меток, затем испускают флуоресценцию, и сигнал улавливается камерами, которые преобразуются в цифровой выход для распознавания оснований. Прикрепленное основание имеет свой терминатор и метку, химически отщепленные по завершении цикла. Цикл повторяется с другим потоком свободных, меченых нуклеотидов через проточную ячейку, чтобы позволить следующему нуклеотиду связаться и захватить свой сигнал. Этот процесс завершается несколько раз (обычно от 50 до 300 раз), чтобы определить последовательность вставленного фрагмента ДНК со скоростью приблизительно 40 миллионов нуклеотидов в секунду по состоянию на 2018 год. [ необходима цитата ]


Технология SOLiD компании Applied Biosystems (теперь бренд Life Technologies ) использует секвенирование путем лигирования . Здесь пул всех возможных олигонуклеотидов фиксированной длины маркируется в соответствии с секвенированным положением. Олигонуклеотиды отжигаются и лигируются; предпочтительное лигирование ДНК-лигазой для сопоставления последовательностей приводит к сигналу, содержащему информацию о нуклеотиде в этом положении. Каждое основание в шаблоне секвенируется дважды, и полученные данные декодируются в соответствии со схемой кодирования двух оснований, используемой в этом методе. Перед секвенированием ДНК амплифицируется с помощью эмульсионной ПЦР. Полученные гранулы, каждая из которых содержит одну копию одной и той же молекулы ДНК, помещаются на предметное стекло. [115] Результатом являются последовательности, величины и длины которых сопоставимы с секвенированием Illumina. [79] Сообщалось, что этот метод секвенирования путем лигирования имеет некоторые проблемы с секвенированием палиндромных последовательностей. [98]
Ion Torrent Systems Inc. (теперь принадлежит Life Technologies ) разработала систему, основанную на использовании стандартной химии секвенирования, но с новой системой обнаружения на основе полупроводников. Этот метод секвенирования основан на обнаружении ионов водорода , которые высвобождаются во время полимеризации ДНК , в отличие от оптических методов, используемых в других системах секвенирования. Микролунку , содержащую шаблонную цепь ДНК, которую нужно секвенировать, заполняют одним типом нуклеотида . Если введенный нуклеотид комплементарен ведущему шаблонному нуклеотиду, он включается в растущую комплементарную цепь. Это вызывает высвобождение иона водорода, который запускает сверхчувствительный ионный датчик, что указывает на то, что произошла реакция. Если в шаблонной последовательности присутствуют гомополимерные повторы, несколько нуклеотидов будут включены в один цикл. Это приводит к соответствующему количеству высвобождаемых водородов и пропорционально более высокому электронному сигналу. [116]

Секвенирование ДНК с помощью наношариков — это тип высокопроизводительной технологии секвенирования, используемой для определения всей геномной последовательности организма. Компания Complete Genomics использует эту технологию для секвенирования образцов, предоставленных независимыми исследователями. Метод использует репликацию по принципу катящегося кольца для амплификации небольших фрагментов геномной ДНК в наношарики ДНК. Затем для определения нуклеотидной последовательности используется нецепочечное секвенирование путем лигирования. [117] Этот метод секвенирования ДНК позволяет секвенировать большое количество наношариков ДНК за один запуск и при низких затратах на реагенты по сравнению с другими высокопроизводительными платформами секвенирования. [118] Однако из каждого наношарика ДНК определяются только короткие последовательности ДНК, что затрудняет сопоставление коротких прочтений с эталонным геномом . [117]
Секвенирование Heliscope — это метод секвенирования отдельных молекул , разработанный Helicos Biosciences . Он использует фрагменты ДНК с добавленными адаптерами поли-А-хвоста, которые прикреплены к поверхности проточной ячейки. Следующие шаги включают секвенирование на основе расширения с циклическими промывками проточной ячейки флуоресцентно мечеными нуклеотидами (один тип нуклеотида за раз, как в методе Сэнгера). Считывания выполняются секвенатором Heliscope. [119] [120] Считывания короткие, в среднем 35 п.н. [121] Что делало эту технологию особенно новой, так это то, что она была первой в своем классе для секвенирования неамплифицированной ДНК, тем самым предотвращая любые ошибки считывания, связанные с этапами амплификации. [122] В 2009 году с помощью Heliscope был секвенирован геном человека, однако в 2012 году компания обанкротилась. [123]
Существуют две основные микрофлюидные системы, которые используются для секвенирования ДНК: микрофлюидика на основе капель и цифровая микрофлюидика . Микрофлюидные устройства решают многие из текущих ограничений современных массивов секвенирования.
Abate et al. изучали использование микрофлюидных устройств на основе капель для секвенирования ДНК. [4] Эти устройства способны формировать и обрабатывать капли размером с пиколитр со скоростью тысячи в секунду. Устройства были созданы из полидиметилсилоксана (PDMS) и использовали перенос энергии резонанса Форстера, анализы FRET для считывания последовательностей ДНК, заключенных в каплях. Каждая позиция на массиве тестировалась на определенную последовательность из 15 оснований. [4]
Фэйр и др. использовали цифровые микрофлюидные устройства для изучения пиросеквенирования ДНК . [124] Значительные преимущества включают портативность устройства, объем реагента, скорость анализа, возможности массового производства и высокую пропускную способность. Это исследование предоставило доказательство концепции, показывающее, что цифровые устройства могут быть использованы для пиросеквенирования; исследование включало использование синтеза, который включает расширение ферментов и добавление меченых нуклеотидов. [124]
Болес и др. также изучали пиросеквенирование на цифровых микрофлюидных устройствах. [125] Они использовали электросмачивающее устройство для создания, смешивания и разделения капель. Секвенирование использует трехферментный протокол и шаблоны ДНК, закрепленные магнитными шариками. Устройство было протестировано с использованием двух протоколов и показало 100% точность на основе уровней сырой пирограммы. Преимущества этих цифровых микрофлюидных устройств включают размер, стоимость и достижимые уровни функциональной интеграции. [125]
Исследования по секвенированию ДНК с использованием микрофлюидики также могут быть применены к секвенированию РНК с использованием аналогичных капельных микрофлюидных методов, таких как метод inDrops. [126] Это показывает, что многие из этих методов секвенирования ДНК могут применяться и в дальнейшем и использоваться для более глубокого понимания геномов и транскриптомов.
Методы секвенирования ДНК, которые в настоящее время находятся в стадии разработки, включают считывание последовательности, когда цепь ДНК проходит через нанопоры (метод, который сейчас является коммерческим, но последующие поколения, такие как твердотельные нанопоры, все еще находятся в стадии разработки), [127] [128] и основанные на микроскопии методы, такие как атомно-силовая микроскопия или просвечивающая электронная микроскопия , которые используются для определения положения отдельных нуклеотидов в длинных фрагментах ДНК (>5000 п.н.) путем маркировки нуклеотидов более тяжелыми элементами (например, галогенами) для визуального обнаружения и регистрации. [129] [130] Технологии третьего поколения направлены на увеличение производительности и сокращение времени получения результата и стоимости за счет устранения необходимости в избыточных реагентах и использования процессивности ДНК-полимеразы. [131]
Другой подход использует измерения электрических туннельных токов через одноцепочечную ДНК, когда она движется через канал. В зависимости от своей электронной структуры каждое основание влияет на туннельный ток по-разному, [132] позволяя дифференцировать различные основания. [133]
Использование туннельных токов имеет потенциал для секвенирования на порядки быстрее, чем методы ионных токов, и уже достигнуто секвенирование нескольких олигомеров ДНК и микроРНК. [134]
Секвенирование гибридизацией — это неферментативный метод, использующий ДНК-микрочип . Отдельный пул ДНК, последовательность которого необходимо определить, помечается флуоресцентно и гибридизуется с массивом, содержащим известные последовательности. Сильные сигналы гибридизации от заданного места на массиве идентифицируют его последовательность в ДНК, которая подвергается секвенированию. [135]
Этот метод секвенирования использует характеристики связывания библиотеки коротких одноцепочечных молекул ДНК (олигонуклеотидов), также называемых ДНК-зондами, для реконструкции целевой последовательности ДНК. Неспецифические гибриды удаляются промывкой, а целевая ДНК элюируется. [136] Гибриды перестраиваются таким образом, что последовательность ДНК может быть реконструирована. Преимуществом этого типа секвенирования является его способность захватывать большое количество целей с однородным покрытием. [137] Обычно требуется большое количество химикатов и исходной ДНК. Однако с появлением гибридизации на основе раствора требуется гораздо меньше оборудования и химикатов. [136]
Масс-спектрометрия может использоваться для определения последовательностей ДНК. Времяпролетная масс-спектрометрия с лазерной десорбцией и ионизацией с матрицей, или MALDI-TOF MS , специально исследовалась как альтернативный метод гель-электрофореза для визуализации фрагментов ДНК. С помощью этого метода фрагменты ДНК, полученные в результате реакций секвенирования с обрывом цепи, сравниваются по массе, а не по размеру. Масса каждого нуклеотида отличается от других, и это различие обнаруживается с помощью масс-спектрометрии. Однонуклеотидные мутации во фрагменте легче обнаружить с помощью MS, чем с помощью одного только гель-электрофореза. MALDI-TOF MS может легче обнаружить различия между фрагментами РНК, поэтому исследователи могут косвенно секвенировать ДНК с помощью методов, основанных на MS, сначала преобразуя ее в РНК. [138]
Более высокое разрешение фрагментов ДНК, допускаемое методами на основе MS, представляет особый интерес для исследователей в области судебной экспертизы, поскольку они могут захотеть найти однонуклеотидные полиморфизмы в образцах человеческой ДНК для идентификации людей. Эти образцы могут быть сильно деградированы, поэтому исследователи-криминалисты часто предпочитают митохондриальную ДНК из -за ее более высокой стабильности и применения для исследований родословной. Методы секвенирования на основе MS использовались для сравнения последовательностей человеческой митохондриальной ДНК из образцов в базе данных Федерального бюро расследований [139] и из костей, найденных в массовых захоронениях солдат Первой мировой войны. [140]
Ранние методы терминации цепи и масс-спектрометрии TOF продемонстрировали длину прочтения до 100 пар оснований. [141] Исследователи не смогли превзойти этот средний размер прочтения; как и только секвенирование терминации цепи, секвенирование ДНК на основе масс-спектрометрии может не подходить для крупных проектов по секвенированию de novo . Тем не менее, недавнее исследование использовало короткие прочтения последовательностей и масс-спектроскопию для сравнения однонуклеотидных полиморфизмов в патогенных штаммах стрептококков . [142]
При микрофлюидном секвенировании по Сэнгеру вся термоциклическая амплификация фрагментов ДНК, а также их разделение электрофорезом выполняется на одной стеклянной пластине (приблизительно 10 см в диаметре), что позволяет сократить использование реагентов и затраты. [143] В некоторых случаях исследователи показали, что они могут увеличить производительность обычного секвенирования за счет использования микрочипов. [144] Для того чтобы сделать такое использование технологии эффективным, еще предстоит провести исследования.
Этот подход напрямую визуализирует последовательность молекул ДНК с помощью электронной микроскопии. Была продемонстрирована первая идентификация пар оснований ДНК в неповрежденных молекулах ДНК путем ферментативного включения модифицированных оснований, содержащих атомы с увеличенным атомным номером, прямая визуализация и идентификация индивидуально маркированных оснований в синтетической молекуле ДНК из 3272 пар оснований и вирусном геноме из 7249 пар оснований. [145]
Этот метод основан на использовании РНК-полимеразы (РНКП), которая прикреплена к полистироловой бусине. Один конец ДНК, которую нужно секвенировать, прикреплен к другой бусине, при этом обе бусины помещаются в оптические ловушки. Движение РНКП во время транскрипции сближает бусины, и их относительное расстояние изменяется, что затем может быть записано с разрешением в один нуклеотид. Последовательность выводится на основе четырех показаний с пониженными концентрациями каждого из четырех типов нуклеотидов, аналогично методу Сэнгера. [146] Проводится сравнение между областями, и информация о последовательности выводится путем сравнения известных областей последовательности с неизвестными областями последовательности. [147]
Разработан метод анализа полных наборов белковых взаимодействий с использованием комбинации 454 пиросеквенирования и метода отображения мРНК вируса in vitro . В частности, этот метод ковалентно связывает интересующие белки с кодирующими их мРНК, а затем обнаруживает части мРНК с помощью обратной транскрипции ПЦР . Затем мРНК может быть амплифицирована и секвенирована. Комбинированный метод был назван IVV-HiTSeq и может быть выполнен в бесклеточных условиях, хотя его результаты могут не быть репрезентативными для условий in vivo . [148]
Хотя существует множество различных способов секвенирования ДНК, на рынке доминируют лишь немногие. В 2022 году Illumina занимала около 80% рынка; остальная часть рынка занята всего несколькими игроками (PacBio, Oxford, 454, MGI) [149]
Успех любого протокола секвенирования ДНК зависит от извлечения и подготовки образца ДНК или РНК из интересующего биологического материала.
После извлечения ДНК или РНК образцы могут потребовать дальнейшей подготовки в зависимости от метода секвенирования. Для секвенирования по Сэнгеру перед секвенированием требуются либо процедуры клонирования, либо ПЦР. В случае методов секвенирования следующего поколения перед обработкой требуется подготовка библиотеки. [151] Оценка качества и количества нуклеиновых кислот как после извлечения, так и после подготовки библиотеки позволяет выявить деградированные, фрагментированные и низкочистые образцы и получить высококачественные данные секвенирования. [152]

В октябре 2006 года фонд X Prize Foundation учредил инициативу по содействию развитию технологий полного секвенирования генома , названную Archon X Prize , намереваясь присудить 10 миллионов долларов «первой команде, которая сможет создать устройство и использовать его для секвенирования 100 человеческих геномов в течение 10 дней или менее, с точностью не более одной ошибки на каждые 100 000 секвенированных оснований, с последовательностями, точно покрывающими не менее 98% генома, и с текущими расходами не более 10 000 долларов США за геном». [153]
Каждый год Национальный институт исследований генома человека (NHGRI) выделяет гранты на новые исследования и разработки в области геномики . Гранты 2010 года и кандидаты 2011 года включают продолжение работы в области микрофлюидных, полонических и основанийно-тяжелых методологий секвенирования. [154]
Технологии секвенирования, описанные здесь, производят необработанные данные, которые необходимо собрать в более длинные последовательности, такие как полные геномы ( сборка последовательностей ). Для достижения этого существует множество вычислительных задач, таких как оценка необработанных данных последовательностей, которая выполняется такими программами и алгоритмами, как Phred и Phrap . Другие задачи связаны с повторяющимися последовательностями, которые часто мешают полной сборке генома, поскольку они встречаются во многих местах генома. Как следствие, многие последовательности не могут быть отнесены к определенным хромосомам . Производство необработанных данных последовательностей — это только начало их детального биоинформатического анализа. [155] Тем не менее, были разработаны новые методы секвенирования и исправления ошибок секвенирования. [156]
Иногда необработанные считывания, произведенные секвенатором, являются правильными и точными только в части своей длины. Использование всего считывания может привести к появлению артефактов в последующих анализах, таких как сборка генома, вызов SNP или оценка экспрессии генов. Было введено два класса программ обрезки, основанных на классах алгоритмов на основе окна или скользящей суммы. [157] Это частичный список алгоритмов обрезки, доступных в настоящее время, с указанием класса алгоритмов, к которым они принадлежат:
Генетика человека была включена в область биоэтики с начала 1970-х годов [164] , и рост использования секвенирования ДНК (особенно высокопроизводительного секвенирования) привел к появлению ряда этических проблем. Одним из ключевых вопросов является право собственности на ДНК человека и данные, полученные при секвенировании этой ДНК. [165] Что касается самой молекулы ДНК, то в главном судебном деле по этой теме Мур против Регентов Калифорнийского университета (1990) постановил, что у людей нет прав собственности на выброшенные клетки или на какую-либо прибыль, полученную с использованием этих клеток (например, в качестве запатентованной клеточной линии ). Однако у людей есть право на осознанное согласие относительно удаления и использования клеток. Что касается данных, полученных с помощью секвенирования ДНК, Мур не дает человеку никаких прав на информацию, полученную из его ДНК. [165]
По мере того, как секвенирование ДНК становится все более распространенным, хранение, безопасность и обмен геномными данными также становятся более важными. [165] [166] Например, одной из проблем является то, что страховщики могут использовать геномные данные человека для изменения своей расценки в зависимости от предполагаемого будущего здоровья человека на основе его ДНК. [166] [167] В мае 2008 года в Соединенных Штатах был подписан Закон о недискриминации генетической информации (GINA), запрещающий дискриминацию на основе генетической информации в отношении медицинского страхования и занятости. [168] [169] В 2012 году Президентская комиссия США по изучению биоэтических вопросов сообщила, что существующее законодательство о конфиденциальности данных секвенирования ДНК, такое как GINA и Закон о переносимости и подотчетности медицинского страхования, было недостаточным, отметив, что данные секвенирования всего генома являются особенно конфиденциальными, поскольку они могут быть использованы для идентификации не только человека, у которого были созданы данные, но и его родственников. [170] [171]
В большинстве штатов США ДНК, которая «брошена», например, найденная на облизанной марке или конверте, кофейной чашке, сигарете, жевательной резинке, бытовом мусоре или волосе, упавшем на тротуар, может быть законно собрана и секвенирована кем угодно, включая полицию, частных детективов, политических оппонентов или людей, вовлеченных в споры об отцовстве. По состоянию на 2013 год, в одиннадцати штатах действуют законы, которые можно интерпретировать как запрещающие «кражу ДНК». [172]
Этические вопросы также были подняты в связи с растущим использованием скрининга генетических вариаций как у новорожденных, так и у взрослых такими компаниями, как 23andMe . [173] [174] Было заявлено, что скрининг генетических вариаций может быть вредным, увеличивая беспокойство у людей, у которых был обнаружен повышенный риск заболевания. [175] Например, в одном случае, отмеченном в Time , врачи, проводившие скрининг больного ребенка на генетические вариации, решили не сообщать родителям о неродственном варианте, связанном с деменцией, из-за вреда, который это могло бы причинить родителям. [176] Однако исследование 2011 года в The New England Journal of Medicine показало, что люди, проходящие профилирование риска заболевания, не показали повышенного уровня беспокойства. [175] Кроме того, разработка технологий секвенирования следующего поколения, таких как секвенирование на основе нанопор, также вызвала дополнительные этические проблемы. [177]
Технологии секвенирования следующего поколения (NGS) произвели революцию в геномных исследованиях. (вводное предложение статьи)