Геномика — это междисциплинарная область молекулярной биологии, фокусирующаяся на структуре, функции, эволюции, картировании и редактировании геномов . Геном — это полный набор ДНК организма , включая все его гены, а также его иерархическую трехмерную структурную конфигурацию. [1] [2] [3] [4] В отличие от генетики , которая занимается изучением отдельных генов и их роли в наследовании, геномика нацелена на коллективную характеристику и количественную оценку всех генов организма, их взаимосвязей и влияния на организм. [5] Гены могут направлять производство белков с помощью ферментов и молекул-мессенджеров. В свою очередь, белки составляют структуры тела, такие как органы и ткани, а также контролируют химические реакции и переносят сигналы между клетками. Геномика также включает в себя секвенирование и анализ геномов с помощью использования высокопроизводительного секвенирования ДНК и биоинформатики для сборки и анализа функции и структуры целых геномов. [6] [7] Достижения в области геномики вызвали революцию в исследованиях, основанных на открытиях, и системной биологии, способствуя пониманию даже самых сложных биологических систем, таких как мозг. [8]
Область также включает исследования внутригеномных (внутри генома) явлений, таких как эпистаз (влияние одного гена на другой), плейотропия (один ген влияет более чем на один признак), гетерозис (гибридная сила) и другие взаимодействия между локусами и аллелями внутри генома. [9]
От греческого ΓΕΝ [10] gen , «ген» (гамма, эпсилон, ню, эпсилон), что означает «становиться, создавать, создание, рождение», и последующих вариантов: генеалогия, генезис, генетика, genic, геномер, генотип, род и т. д. Хотя слово геном (от немецкого Genom , приписываемого Гансу Винклеру ) использовалось в английском языке еще в 1926 году, [11] термин геномика был придуман Томом Родериком, генетиком из Лаборатории Джексона ( Бар-Харбор, Мэн ), за кружкой пива с Джимом Уомаком, Томом Шоусом и Стивеном О'Брайеном на встрече, состоявшейся в Мэриленде по картированию генома человека в 1986 году. [12] Сначала как название нового журнала , а затем как целой новой научной дисциплины. [13]
После подтверждения Розалинд Франклин спиральной структуры ДНК, публикации Джеймсом Д. Уотсоном и Фрэнсисом Криком структуры ДНК в 1953 году и публикации Фредом Сэнгером аминокислотной последовательности инсулина в 1955 году секвенирование нуклеиновых кислот стало основной целью ранних молекулярных биологов . [14] В 1964 году Роберт В. Холли и его коллеги опубликовали первую когда-либо определенную последовательность нуклеиновых кислот, рибонуклеотидную последовательность аланиновой транспортной РНК . [15] [16] Продолжая эту работу, Маршалл Ниренберг и Филип Ледер раскрыли триплетную природу генетического кода и смогли определить последовательности 54 из 64 кодонов в своих экспериментах. [17] В 1972 году Уолтер Фирс и его команда в Лаборатории молекулярной биологии Гентского университета ( Гент , Бельгия ) первыми определили последовательность гена: ген белка оболочки бактериофага MS2 . [18] Группа Фирса расширила свою работу по белку оболочки MS2, определив полную нуклеотидную последовательность бактериофага MS2-RNA (геном которого кодирует всего четыре гена в 3569 парах оснований [пн]) и вируса обезьян 40 в 1976 и 1978 годах соответственно. [19] [20]
В дополнение к его основополагающей работе по аминокислотной последовательности инсулина, Фредерик Сэнгер и его коллеги сыграли ключевую роль в разработке методов секвенирования ДНК, которые позволили создать комплексные проекты по секвенированию генома. [9] В 1975 году он и Алан Коулсон опубликовали процедуру секвенирования с использованием ДНК-полимеразы с радиоактивно мечеными нуклеотидами, которую он назвал техникой «Плюс и Минус» . [21] [22] Это включало два тесно связанных метода, которые генерировали короткие олигонуклеотиды с определенными 3'-концами. Их можно было фракционировать с помощью электрофореза в полиакриламидном геле (так называемый электрофорез в полиакриламидном геле) и визуализировать с помощью авторадиографии. Процедура могла секвенировать до 80 нуклеотидов за один раз и была большим улучшением, но все еще была очень трудоемкой. Тем не менее, в 1977 году его группа смогла секвенировать большую часть из 5386 нуклеотидов одноцепочечного бактериофага φX174 , завершив первый полностью секвенированный геном на основе ДНК. [23] Усовершенствование метода «Плюс и минус» привело к появлению метода обрыва цепи, или метода Сэнгера (см. ниже), который лег в основу методов секвенирования ДНК, картирования генома, хранения данных и биоинформатического анализа, наиболее широко используемых в последующую четверть века исследований. [24] [25] В том же году Уолтер Гилберт и Аллан Максам из Гарвардского университета независимо друг от друга разработали метод Максама-Гилберта (также известный как химический метод ) секвенирования ДНК, включающий преимущественное расщепление ДНК по известным основаниям, менее эффективный метод. [26] [27] За свою новаторскую работу в области секвенирования нуклеиновых кислот Гилберт и Сэнгер разделили половину Нобелевской премии по химии 1980 года с Полом Бергом ( рекомбинантная ДНК ).
Появление этих технологий привело к быстрому расширению масштабов и скорости завершения проектов по секвенированию генома . Первая полная последовательность генома эукариотической органеллы , человеческой митохондрии (16 568 п.н., около 16,6 кб [килобаз]), была опубликована в 1981 году [28] , а первые геномы хлоропластов последовали в 1986 году [29] [30] В 1992 году была секвенирована первая эукариотическая хромосома , хромосома III пивных дрожжей Saccharomyces cerevisiae (315 кб). [31] Первым свободно живущим организмом, который был секвенирован, был Haemophilus influenzae (1,8 Мб [мегабаза]) в 1995 году. [32] В следующем году консорциум исследователей из лабораторий Северной Америки , Европы и Японии объявил о завершении первой полной последовательности генома эукариота, S. cerevisiae (12,1 Мб), и с тех пор геномы продолжают секвенироваться экспоненциально растущими темпами. [33] По состоянию на октябрь 2011 года [обновлять]полные последовательности доступны для: 2719 вирусов , 1115 архей и бактерий и 36 эукариот , из которых около половины являются грибами . [34] [35]

Большинство микроорганизмов, геномы которых были полностью секвенированы, являются проблемными патогенами , такими как Haemophilus influenzae , что привело к выраженному смещению в их филогенетическом распределении по сравнению с широтой микробного разнообразия. [36] [37] Из других секвенированных видов большинство были выбраны потому, что они были хорошо изученными модельными организмами или обещали стать хорошими моделями. Дрожжи ( Saccharomyces cerevisiae ) долгое время были важным модельным организмом для эукариотической клетки , в то время как плодовая мушка Drosophila melanogaster была очень важным инструментом (особенно в ранней домолекулярной генетике ). Червь Caenorhabditis elegans является часто используемой простой моделью для многоклеточных организмов . Рыбка данио-рерио Brachydanio rerio используется для многих исследований развития на молекулярном уровне, а растение Arabidopsis thaliana является модельным организмом для цветковых растений. Японская рыба-собака ( Takifugu rubripes ) и пятнистая зеленая рыба-собака ( Tetraodon nigroviridis ) интересны своими небольшими и компактными геномами, которые содержат очень мало некодирующей ДНК по сравнению с большинством видов. [38] [39] Млекопитающие собака ( Canis familiaris ), [40] серая крыса ( Rattus norvegicus ), мышь ( Mus musculus ) и шимпанзе ( Pan troglodytes ) являются важными модельными животными в медицинских исследованиях. [27]
Черновой вариант генома человека был завершен в рамках проекта «Геном человека» в начале 2001 года, что вызвало много шума. [41] В рамках этого проекта, завершенного в 2003 году, был секвенирован весь геном одного конкретного человека, и к 2007 году эта последовательность была объявлена «завершенной» (менее одной ошибки на 20 000 оснований и все хромосомы были собраны). [41] За прошедшие с тех пор годы были секвенированы геномы многих других людей, частично под эгидой проекта «1000 геномов» , который объявил о секвенировании 1092 геномов в октябре 2012 года. [42] Завершение этого проекта стало возможным благодаря разработке значительно более эффективных технологий секвенирования и потребовало привлечения значительных ресурсов биоинформатики из большого международного сотрудничества. [43] Продолжающийся анализ данных генома человека имеет глубокие политические и социальные последствия для человеческого общества. [44]

Англоязычный неологизм omics неформально относится к области изучения в биологии, заканчивающейся на -omics , например, геномика, протеомика или метаболомика . Связанный суффикс -ome используется для обозначения объектов изучения таких областей, таких как геном , протеом или метаболом ( липидом ) соответственно. Суффикс -ome , используемый в молекулярной биологии, относится к совокупности некоторого рода; аналогично omics стал в целом относиться к изучению больших, всеобъемлющих наборов биологических данных. Хотя рост использования термина привел некоторых ученых ( Джонатан Эйзен и другие [45] ) к утверждению, что он был переоценен, [46] он отражает изменение ориентации в сторону количественного анализа полного или почти полного ассортимента всех компонентов системы. [47] Например, при изучении симбиозов исследователи, которые когда-то были ограничены изучением одного генного продукта, теперь могут одновременно сравнивать полный набор нескольких типов биологических молекул. [48] [49]
После выбора организма геномные проекты включают три компонента: секвенирование ДНК, сборку этой последовательности для создания представления исходной хромосомы, а также аннотацию и анализ этого представления. [9]
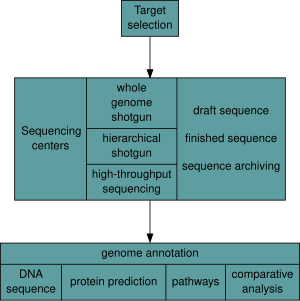
Исторически секвенирование проводилось в центрах секвенирования , централизованных учреждениях (от крупных независимых учреждений, таких как Joint Genome Institute , которые секвенируют десятки терабаз в год, до местных основных учреждений молекулярной биологии), которые содержат исследовательские лаборатории с дорогостоящим оборудованием и необходимой технической поддержкой. Однако, поскольку технология секвенирования продолжает совершенствоваться, новое поколение эффективных настольных секвенаторов с быстрым оборотом стало доступно для средней академической лаборатории. [50] [51] В целом, подходы к секвенированию генома делятся на две широкие категории: дробовик и высокопроизводительное (или следующего поколения ) секвенирование. [9]

Секвенирование методом дробовика — это метод секвенирования, разработанный для анализа последовательностей ДНК длиной более 1000 пар оснований, вплоть до целых хромосом. [52] Он назван по аналогии с быстро расширяющейся квазислучайной схемой выстрела дробовика . Поскольку секвенирование с помощью гель-электрофореза может использоваться только для довольно коротких последовательностей (от 100 до 1000 пар оснований), более длинные последовательности ДНК должны быть разбиты на случайные небольшие сегменты, которые затем секвенируются для получения прочтений . Множественные перекрывающиеся прочтения для целевой ДНК получаются путем выполнения нескольких раундов этой фрагментации и секвенирования. Затем компьютерные программы используют перекрывающиеся концы различных прочтений, чтобы собрать их в непрерывную последовательность. [52] [53] Секвенирование методом дробовика — это процесс случайной выборки, требующий избыточной выборки для обеспечения того, чтобы заданный нуклеотид был представлен в реконструированной последовательности; среднее число прочтений, на которые геном перевыбран, называется покрытием . [54]
На протяжении большей части своей истории технология, лежащая в основе дробового секвенирования, была классическим методом терминации цепи или « методом Сэнгера », который основан на селективном включении дидезоксинуклеотидов , завершающих цепь , ДНК-полимеразой во время репликации ДНК in vitro . [23] [55] В последнее время дробовое секвенирование было вытеснено высокопроизводительными методами секвенирования, особенно для крупномасштабных автоматизированных геномных анализов. Тем не менее, метод Сэнгера по-прежнему широко используется, в основном для проектов меньшего масштаба и для получения особенно длинных непрерывных последовательностей ДНК (>500 нуклеотидов). [56] Методы терминации цепи требуют одноцепочечной матрицы ДНК, праймера ДНК , ДНК-полимеразы , нормальных дезоксинуклеозидтрифосфатов (dNTP) и модифицированных нуклеотидов (dideoxyNTP), которые завершают удлинение цепи ДНК. Эти нуклеотиды, завершающие цепь, не имеют 3'- ОН- группы, необходимой для образования фосфодиэфирной связи между двумя нуклеотидами, что приводит к прекращению ДНК-полимеразой удлинения ДНК при включении ddNTP. ddNTP могут быть радиоактивно или флуоресцентно помечены для обнаружения в секвенаторах ДНК . [9] Обычно эти машины могут секвенировать до 96 образцов ДНК в одной партии (запуске) при выполнении до 48 запусков в день. [57]
Высокий спрос на недорогое секвенирование привел к разработке технологий высокопроизводительного секвенирования, которые распараллеливают процесс секвенирования, производя тысячи или миллионы последовательностей одновременно. [58] [59] Высокопроизводительное секвенирование предназначено для снижения стоимости секвенирования ДНК сверх того, что возможно при использовании стандартных методов терминации красителем. При сверхвысокопроизводительном секвенировании параллельно может выполняться до 500 000 операций секвенирования путем синтеза. [60] [61]

Метод секвенирования красителей Illumina основан на обратимых терминаторах красителей и был разработан в 1996 году в Женевском институте биомедицинских исследований Паскалем Майером и Лораном Фаринелли. [62] В этом методе молекулы ДНК и праймеры сначала прикрепляются к предметному стеклу и амплифицируются полимеразой, так что образуются локальные клональные колонии, изначально названные «колониями ДНК». Для определения последовательности добавляются четыре типа обратимых терминаторных оснований (RT-основы), а невключенные нуклеотиды смываются. В отличие от пиросеквенирования, цепи ДНК удлиняются по одному нуклеотиду за раз, и получение изображения может быть выполнено в отложенный момент, что позволяет захватывать очень большие массивы колоний ДНК с помощью последовательных изображений, полученных с одной камеры. Разделение ферментативной реакции и захвата изображения обеспечивает оптимальную пропускную способность и теоретически неограниченную емкость секвенирования; при оптимальной конфигурации конечная пропускная способность прибора зависит только от скорости преобразования АЦП камеры. Камера делает снимки флуоресцентно меченых нуклеотидов, затем краситель вместе с терминальным 3'-блокатором химически удаляется из ДНК, что позволяет начать следующий цикл. [63]
Альтернативный подход, ионно-полупроводниковое секвенирование, основан на стандартной химии репликации ДНК. Эта технология измеряет высвобождение иона водорода каждый раз, когда включается основание. Микролунку, содержащую шаблонную ДНК, заливают одним нуклеотидом , если нуклеотид комплементарен шаблонной цепи, он будет включен, и ион водорода будет освобожден. Этот выброс активирует ионный датчик ISFET . Если в шаблонной последовательности присутствует гомополимер, несколько нуклеотидов будут включены в один цикл затопления, и обнаруженный электрический сигнал будет пропорционально выше. [64]
Сборка последовательности относится к выравниванию и слиянию фрагментов гораздо более длинной последовательности ДНК для реконструкции исходной последовательности. [9] Это необходимо, поскольку современная технология секвенирования ДНК не может считывать целые геномы как непрерывную последовательность, а скорее считывает небольшие фрагменты от 20 до 1000 оснований, в зависимости от используемой технологии. Технологии секвенирования третьего поколения, такие как PacBio или Oxford Nanopore, обычно генерируют считывания последовательности длиной 10-100 кб; однако они имеют высокий уровень ошибок, приблизительно 1 процент. [65] [66] Обычно короткие фрагменты, называемые считываниями, являются результатом дробового секвенирования геномной ДНК или генных транскриптов ( EST ). [9]
Сборку можно в целом разделить на два подхода: сборка de novo для геномов, которые не похожи ни на один из секвенированных в прошлом, и сравнительная сборка, которая использует существующую последовательность близкородственного организма в качестве эталона во время сборки. [54] По сравнению со сравнительной сборкой сборка de novo является вычислительно сложной ( NP-трудной ), что делает ее менее благоприятной для технологий короткого считывания NGS. В парадигме сборки de novo есть две основные стратегии для сборки: стратегии эйлерова пути и стратегии перекрытия-разметки-консенсуса (OLC). Стратегии OLC в конечном итоге пытаются создать гамильтонов путь через граф перекрытия, что является NP-трудной задачей. Стратегии эйлерова пути вычислительно более поддаются обработке, поскольку они пытаются найти эйлеров путь через граф де Брейна. [54]
Завершенные геномы определяются как имеющие единую непрерывную последовательность без двусмысленностей, представляющую каждый репликон . [67]
Сборка последовательности ДНК сама по себе не имеет большой ценности без дополнительного анализа. [9] Аннотирование генома — это процесс присоединения биологической информации к последовательностям , который состоит из трех основных этапов: [68]
Инструменты автоматического аннотирования пытаются выполнить эти шаги in silico , в отличие от ручного аннотирования (также известного как курирование), которое включает человеческий опыт и потенциальную экспериментальную проверку. [69] В идеале эти подходы сосуществуют и дополняют друг друга в одном и том же конвейере аннотирования (см. также ниже).
Традиционно базовый уровень аннотации заключается в использовании BLAST для поиска сходств, а затем аннотации геномов на основе гомологов. [9] Совсем недавно на платформу аннотации была добавлена дополнительная информация. Дополнительная информация позволяет ручным аннотаторам деконволюционировать несоответствия между генами, которым дана одна и та же аннотация. Некоторые базы данных используют информацию о контексте генома, оценки сходства, экспериментальные данные и интеграцию других ресурсов для предоставления аннотаций генома с помощью своего подхода Subsystems. Другие базы данных (например, Ensembl ) полагаются как на курируемые источники данных, так и на ряд программных инструментов в своем автоматизированном конвейере аннотации генома. [70] Структурная аннотация состоит из идентификации геномных элементов, в первую очередь ORF и их локализации или структуры гена. Функциональная аннотация состоит из присоединения биологической информации к геномным элементам.
Необходимость воспроизводимости и эффективного управления большими объемами данных, связанных с геномными проектами, означает, что вычислительные конвейеры имеют важные приложения в геномике. [71]
Функциональная геномика — это область молекулярной биологии , которая пытается использовать огромное количество данных, полученных в ходе геномных проектов (например, проектов по секвенированию генома ), для описания функций и взаимодействий генов (и белков ). Функциональная геномика фокусируется на динамических аспектах, таких как транскрипция генов , трансляция и взаимодействия белок-белок , в отличие от статических аспектов геномной информации, таких как последовательность или структуры ДНК. Функциональная геномика пытается ответить на вопросы о функции ДНК на уровне генов, транскриптов РНК и белковых продуктов. Ключевой характеристикой исследований функциональной геномики является их подход к этим вопросам на уровне всего генома, обычно включающий высокопроизводительные методы, а не более традиционный подход «ген за геном».
Основная ветвь геномики по-прежнему занимается секвенированием геномов различных организмов, но знание полных геномов создало возможность для области функциональной геномики , в основном занимающейся паттернами экспрессии генов в различных условиях. Наиболее важными инструментами здесь являются микрочипы и биоинформатика .

Структурная геномика стремится описать трехмерную структуру каждого белка, кодируемого данным геномом . [72] [73] Этот подход, основанный на геноме, позволяет использовать высокопроизводительный метод определения структуры путем сочетания экспериментальных и модельных подходов . Принципиальное различие между структурной геномикой и традиционным структурным прогнозированием заключается в том, что структурная геномика пытается определить структуру каждого белка, кодируемого геномом, а не сосредотачивается на одном конкретном белке. При наличии полных геномных последовательностей прогнозирование структуры может быть выполнено быстрее с помощью сочетания экспериментальных и модельных подходов, особенно потому, что наличие большого количества секвенированных геномов и ранее решенных структур белков позволяет ученым моделировать структуру белка на основе структур ранее решенных гомологов. Структурная геномика включает в себя использование большого количества подходов к определению структуры, включая экспериментальные методы с использованием геномных последовательностей или подходов, основанных на моделировании, основанных на гомологии последовательности или структуры с белком известной структуры или на химических и физических принципах для белка без гомологии с какой-либо известной структурой. В отличие от традиционной структурной биологии , определение структуры белка посредством структурной геномики часто (но не всегда) происходит до того, как что-либо известно о функции белка. Это поднимает новые проблемы в структурной биоинформатике , то есть определении функции белка из его трехмерной структуры. [74]
Эпигеномика — это изучение полного набора эпигенетических модификаций генетического материала клетки, известного как эпигеном . [75] Эпигенетические модификации — это обратимые модификации ДНК или гистонов клетки, которые влияют на экспрессию генов, не изменяя последовательность ДНК (Russell 2010, стр. 475). Две из наиболее характерных эпигенетических модификаций — это метилирование ДНК и модификация гистонов . [76] Эпигенетические модификации играют важную роль в экспрессии и регуляции генов и участвуют в многочисленных клеточных процессах, таких как дифференциация/развитие [77] и опухолеобразование . [75] Изучение эпигенетики на глобальном уровне стало возможным лишь недавно благодаря адаптации геномных высокопроизводительных анализов. [78]
Метагеномика — это изучение метагеномов , генетического материала, извлеченного непосредственно из образцов окружающей среды . Широкая область может также называться экологической геномикой, экогеномикой или геномикой сообществ. В то время как традиционная микробиология и секвенирование микробного генома полагаются на культивируемые клонированные культуры , раннее секвенирование генов окружающей среды клонировало определенные гены (часто ген 16S рРНК ) для получения профиля разнообразия в естественном образце. Такая работа показала, что подавляющее большинство микробного биоразнообразия было упущено методами , основанными на культивировании . [79] Недавние исследования используют секвенирование по методу «дробовика» Сэнгера или массивное параллельное пиросеквенирование для получения в значительной степени беспристрастных образцов всех генов от всех членов отобранных сообществ. [80] Благодаря своей способности раскрывать ранее скрытое разнообразие микроскопической жизни, метагеномика предлагает мощную линзу для просмотра микробного мира, которая имеет потенциал для революционного изменения понимания всего живого мира. [81] [82]
Бактериофаги играли и продолжают играть ключевую роль в генетике бактерий и молекулярной биологии . Исторически они использовались для определения структуры генов и регуляции генов. Также первым секвенированным геномом был бактериофаг . Однако исследования бактериофагов не привели к революции в геномике, в которой явно доминирует бактериальная геномика. Лишь совсем недавно изучение геномов бактериофагов стало заметным, что позволило исследователям понять механизмы, лежащие в основе эволюции фагов . Последовательности геномов бактериофагов можно получить путем прямого секвенирования изолированных бактериофагов, но их также можно получить как часть микробных геномов. Анализ бактериальных геномов показал, что значительное количество микробной ДНК состоит из последовательностей профагов и профагоподобных элементов. [83] Подробный анализ базы данных этих последовательностей позволяет получить представление о роли профагов в формировании бактериального генома: в целом, этот метод подтвердил многие известные группы бактериофагов, что делает его полезным инструментом для прогнозирования взаимосвязей профагов из бактериальных геномов. [84] [85]
В настоящее время существует 24 цианобактерии , для которых доступна полная последовательность генома. 15 из этих цианобактерий происходят из морской среды. Это шесть штаммов Prochlorococcus , семь морских штаммов Synechococcus , Trichodesmium erythraeum IMS101 и Crocosphaera watsonii WH8501. Несколько исследований продемонстрировали, как эти последовательности могут быть очень успешно использованы для выведения важных экологических и физиологических характеристик морских цианобактерий. Однако в настоящее время ведется еще много геномных проектов, среди которых есть дополнительные изоляты Prochlorococcus и морских Synechococcus , Acaryochloris и Prochloron , фиксирующие N 2 нитчатые цианобактерии Nodularia spumigena , Lyngbya aestuarii и Lyngbya majuscula , а также бактериофаги, инфицирующие морские цианобактерии. Таким образом, растущий объем геномной информации может быть также использован более общим образом для решения глобальных проблем путем применения сравнительного подхода. Некоторые новые и захватывающие примеры прогресса в этой области включают идентификацию генов для регуляторных РНК, понимание эволюционного происхождения фотосинтеза или оценку вклада горизонтального переноса генов в проанализированные геномы. [86]

Геномика нашла применение во многих областях, включая медицину , биотехнологию , антропологию и другие социальные науки . [44]
Геномные технологии следующего поколения позволяют врачам и биомедицинским исследователям радикально увеличить объем геномных данных, собираемых на больших выборках. [87] В сочетании с новыми информационными подходами, которые интегрируют многие виды данных с геномными данными в исследованиях заболеваний, это позволяет исследователям лучше понять генетические основы реакции на лекарственные препараты и заболевания. [88] [89]
Ранние попытки применить геном в медицине включали усилия команды Стэнфорда во главе с Юаном Эшли , который разработал первые инструменты для медицинской интерпретации генома человека. [90] [91] [92] Исследовательская программа Genomes2People в больнице Brigham and Women's Hospital , Институте Брода и Гарвардской медицинской школе была создана в 2012 году для проведения эмпирических исследований по переводу геномики в здравоохранение. Больница Brigham and Women's Hospital открыла клинику профилактической геномики в августе 2019 года, а через месяц за ней последовала больница общего профиля Массачусетса . [93] [94] Исследовательская программа All of Us направлена на сбор данных о последовательности генома у 1 миллиона участников, чтобы стать важнейшим компонентом исследовательской платформы точной медицины [95] , а инициатива UK Biobank изучила более 500 000 человек с помощью глубоких геномных и фенотипических данных. [96]
Рост геномных знаний позволил использовать все более сложные приложения синтетической биологии . [97] В 2010 году исследователи из Института Дж. Крейга Вентера объявили о создании частично синтетического вида бактерий , Mycoplasma laboratorium , полученного из генома Mycoplasma genitalium . [98]
Популяционная геномика развилась как популярная область исследований, где методы геномного секвенирования используются для проведения крупномасштабных сравнений последовательностей ДНК среди популяций - за пределами генетических маркеров, таких как продукты ПЦР короткого радиуса действия или микросателлиты, традиционно используемые в популяционной генетике . Популяционная геномика изучает эффекты на уровне генома , чтобы улучшить наше понимание микроэволюции , чтобы мы могли узнать филогенетическую историю и демографию популяции. [99] Методы популяционной геномики используются во многих различных областях, включая эволюционную биологию , экологию , биогеографию , биологию сохранения и управление рыболовством . Аналогичным образом ландшафтная геномика развилась из ландшафтной генетики , чтобы использовать геномные методы для выявления связей между моделями экологической и генетической изменчивости.
Специалисты по охране природы могут использовать информацию, собранную с помощью геномного секвенирования, чтобы лучше оценить генетические факторы, имеющие ключевое значение для сохранения видов, такие как генетическое разнообразие популяции или является ли особь гетерозиготной по рецессивному наследственному генетическому заболеванию. [100] Используя геномные данные для оценки эффектов эволюционных процессов и выявления закономерностей в изменчивости в пределах данной популяции, специалисты по охране природы могут разрабатывать планы по оказанию помощи данному виду, не оставляя неизвестными столько же переменных, сколько те, которые не учитываются стандартными генетическими подходами . [101]
{{cite book}}: CS1 maint: DOI неактивен по состоянию на ноябрь 2024 г. ( ссылка )электронная книга ISBN 978-94-007-5561-1 ISSN 1559-0836 электронная- ISSN 1868-0402