
Гомологичная рекомбинация — тип генетической рекомбинации , при котором происходит обмен генетической информацией между двумя похожими или идентичными молекулами двухцепочечных или одноцепочечных нуклеиновых кислот (обычно ДНК, как в клеточных организмах, но может быть и РНК у вирусов ).
Гомологичная рекомбинация широко используется клетками для точного восстановления вредных разрывов ДНК, которые возникают на обеих цепях ДНК, известных как двухцепочечные разрывы (DSB), в процессе, называемом гомологичной рекомбинационной репарацией (HRR). [1]
Гомологичная рекомбинация также производит новые комбинации последовательностей ДНК во время мейоза , процесса, посредством которого эукариоты создают гаметные клетки, такие как сперма и яйцеклетки у животных. Эти новые комбинации ДНК представляют собой генетическую изменчивость у потомства, что, в свою очередь, позволяет популяциям адаптироваться в ходе эволюции . [2]
Гомологичная рекомбинация также используется в горизонтальном переносе генов для обмена генетическим материалом между различными штаммами и видами бактерий и вирусов. Горизонтальный перенос генов является основным механизмом распространения устойчивости к антибиотикам у бактерий.
Хотя гомологичная рекомбинация широко варьируется среди разных организмов и типов клеток, для двухцепочечной ДНК ( dsDNA ) большинство форм включают одни и те же основные этапы. После того, как происходит двухцепочечный разрыв, участки ДНК вокруг 5'-концов разрыва отрезаются в процессе, называемом резекцией . На следующем этапе вторжения в цепь выступающий 3'-конец разорванной молекулы ДНК затем «вторгается» в похожую или идентичную молекулу ДНК, которая не разорвана. После вторжения в цепь дальнейшая последовательность событий может следовать одному из двух основных путей, обсуждаемых ниже (см. Модели); путь DSBR (репарация двухцепочечного разрыва) или путь SDSA (отжиг цепи, зависящий от синтеза). Гомологичная рекомбинация, которая происходит во время репарации ДНК, как правило, приводит к некроссоверным продуктам, по сути восстанавливая поврежденную молекулу ДНК в том виде, в котором она существовала до двухцепочечного разрыва.
Гомологичная рекомбинация сохраняется во всех трех доменах жизни, а также в ДНК- и РНК- вирусах , что позволяет предположить, что это почти универсальный биологический механизм. Открытие генов гомологичной рекомбинации у протистов — разнообразной группы эукариотических микроорганизмов — было интерпретировано как доказательство того, что гомологичная рекомбинация возникла на ранних этапах эволюции эукариот. Поскольку их дисфункция была тесно связана с повышенной восприимчивостью к нескольким типам рака , белки, которые облегчают гомологичную рекомбинацию, являются темами активных исследований. Гомологичная рекомбинация также используется в нацеливании генов — методе внесения генетических изменений в целевые организмы. За разработку этого метода Марио Капеччи , Мартин Эванс и Оливер Смитис были удостоены Нобелевской премии по физиологии и медицине 2007 года ; Капеччи [3] и Смитис [4] независимо друг от друга открыли применение к эмбриональным стволовым клеткам мышей, однако высококонсервативные механизмы, лежащие в основе модели восстановления DSB, включая равномерную гомологичную интеграцию трансформированной ДНК (генная терапия), были впервые продемонстрированы в экспериментах с плазмидами Орр-Уивером, Шостаком и Ротштейном. [5] [6] [7] Исследования индуцированных плазмидой DSB с использованием γ-облучения [8] в 1970-1980-х годах привели к более поздним экспериментам с использованием эндонуклеаз (например, I-SceI) для разрезания хромосом в целях генной инженерии клеток млекопитающих, где негомологичная рекомбинация встречается чаще, чем в дрожжах. [9]

В начале 1900-х годов Уильям Бейтсон и Реджинальд Паннетт обнаружили исключение из одного из принципов наследования, первоначально описанного Грегором Менделем в 1860-х годах. В отличие от представления Менделя о том, что признаки независимо сортируются при передаче от родителя к ребенку — например, что цвет шерсти кошки и длина ее хвоста наследуются независимо друг от друга — Бейтсон и Паннетт показали, что определенные гены, связанные с физическими признаками, могут наследоваться вместе или быть генетически связанными . [10] [11] В 1911 году, заметив, что связанные признаки иногда могут наследоваться по отдельности, Томас Хант Морган предположил, что между связанными генами могут происходить « кроссоверы », [12] когда один из связанных генов физически переходит на другую хромосому . Два десятилетия спустя Барбара МакКлинток и Харриет Крейтон продемонстрировали, что кроссинговер хромосом происходит во время мейоза , [13] [14] процесса деления клеток, в ходе которого образуются сперматозоиды и яйцеклетки . В том же году, когда было сделано открытие МакКлинток, Курт Стерн показал, что кроссинговер, позже названный «рекомбинацией», может также происходить в соматических клетках, таких как лейкоциты и клетки кожи , которые делятся посредством митоза . [13] [15]
В 1947 году микробиолог Джошуа Ледерберг показал, что бактерии, которые, как предполагалось, размножаются только бесполым путем посредством бинарного деления , способны к генетической рекомбинации, которая больше похожа на половое размножение. Эта работа установила E. coli как модельный организм в генетике [16] и помогла Ледербергу выиграть Нобелевскую премию по физиологии и медицине 1958 года . [17] Основываясь на исследованиях грибов , в 1964 году Робин Холлидей предложил модель рекомбинации в мейозе, которая представила ключевые детали того, как может работать этот процесс, включая обмен материалом между хромосомами через соединения Холлидея . [18] В 1983 году Джек Шостак и его коллеги представили модель, теперь известную как путь DSBR, которая учитывала наблюдения, не объясненные моделью Холлидея. [18] [7] В течение следующего десятилетия эксперименты на дрозофиле , почкующихся дрожжах и клетках млекопитающих привели к появлению других моделей гомологичной рекомбинации, называемых путями SDSA, которые не всегда полагаются на соединения Холлидея. [18]
Большая часть более поздней работы по идентификации белков, участвующих в этом процессе, и определению их механизмов была проделана рядом специалистов, включая Джеймса Хабера , Патрика Санга , Стивена Ковальчиковски и других.
Гомологичная рекомбинация (HR) необходима для деления клеток у эукариот, таких как растения, животные, грибы и простейшие. Гомологичная рекомбинация восстанавливает двухцепочечные разрывы в ДНК, вызванные ионизирующим излучением или химическими веществами, повреждающими ДНК. [19] Если их не восстанавливать, эти двухцепочечные разрывы могут вызвать крупномасштабную перестройку хромосом в соматических клетках , [20] что в свою очередь может привести к раку. [21]
Помимо восстановления ДНК, гомологичная рекомбинация также помогает производить генетическое разнообразие , когда клетки делятся в мейозе, чтобы стать специализированными гаметными клетками — сперматозоидами или яйцеклетками у животных, пыльцой или семяпочками у растений и спорами у грибов . Это происходит за счет содействия хромосомному кроссинговеру , при котором области похожей, но не идентичной ДНК обмениваются между гомологичными хромосомами . [22] [23] Это создает новые, возможно, полезные комбинации генов, которые могут дать потомству эволюционное преимущество. [24] Хромосомный кроссинговер часто начинается, когда белок под названием Spo11 делает целевой двухцепочечный разрыв в ДНК. [25] Эти участки неслучайно расположены на хромосомах; обычно в межгенных промоторных областях и преимущественно в доменах , богатых GC [26] Эти сайты двухцепочечных разрывов часто встречаются в рекомбинационных горячих точках , областях хромосом, длина которых составляет около 1000–2000 пар оснований и которые имеют высокие показатели рекомбинации. Отсутствие рекомбинационной горячей точки между двумя генами на одной хромосоме часто означает, что эти гены будут унаследованы будущими поколениями в равной пропорции. Это представляет собой сцепление между двумя генами, большее, чем можно было бы ожидать от генов, которые независимо сортируются во время мейоза. [27]

Двухцепочечные разрывы могут быть восстановлены посредством гомологичной рекомбинации, полимеразного тета-опосредованного соединения концов (TMEJ) или посредством негомологичного соединения концов (NHEJ). [28] NHEJ — это механизм репарации ДНК, который, в отличие от гомологичной рекомбинации, не требует длинной гомологичной последовательности для управления репарацией. Используется ли гомологичная рекомбинация или NHEJ для восстановления двухцепочечных разрывов, во многом определяется фазой клеточного цикла . Гомологичная рекомбинация восстанавливает ДНК до того, как клетка вступит в митоз (фазу М). Она происходит во время и вскоре после репликации ДНК , в фазах S и G 2 клеточного цикла, когда сестринские хроматиды более доступны. [29] По сравнению с гомологичными хромосомами, которые похожи на другую хромосому, но часто имеют разные аллели , сестринские хроматиды являются идеальным шаблоном для гомологичной рекомбинации, поскольку они являются идентичной копией данной хромосомы. Когда гомологичный шаблон недоступен или когда шаблон не может быть доступен из-за дефекта гомологичной рекомбинации, разрыв восстанавливается посредством TMEJ в фазах S и G 2 клеточного цикла. В отличие от гомологичной рекомбинации и TMEJ, NHEJ преобладает в фазе G 1 клеточного цикла, когда клетка растет, но еще не готова к делению. Он встречается реже после фазы G 1 , но сохраняет по крайней мере некоторую активность на протяжении всего клеточного цикла. Механизмы, которые регулируют гомологичную рекомбинацию и NHEJ на протяжении всего клеточного цикла, сильно различаются между видами. [30]
Циклинзависимые киназы (CDK), которые изменяют активность других белков, добавляя к ним фосфатные группы (то есть фосфорилируя их), являются важными регуляторами гомологичной рекомбинации у эукариот. [30] Когда в почкующихся дрожжах начинается репликация ДНК, циклинзависимая киназа Cdc28 начинает гомологичную рекомбинацию, фосфорилируя белок Sae2 . [31] После активации добавлением фосфата Sae2 вызывает чистый разрез вблизи двухцепочечного разрыва ДНК. Неясно, является ли эндонуклеаза, ответственная за этот разрез, самим Sae2 или другим белком, Mre11 . [32] Это позволяет белковому комплексу, включающему Mre11, известному как комплекс MRX , связываться с ДНК и начинать серию управляемых белками реакций, которые обмениваются материалом между двумя молекулами ДНК. [33]
Упаковка эукариотической ДНК в хроматин представляет собой барьер для всех основанных на ДНК процессов, которые требуют привлечения ферментов к их сайтам действия. Чтобы обеспечить гомологичную рекомбинацию (HR) ДНК-репарацию, хроматин должен быть ремоделирован. У эукариот зависимые от АТФ комплексы ремоделирования хроматина и гистон-модифицирующие ферменты являются двумя преобладающими факторами, используемыми для выполнения этого процесса ремоделирования. [34]
Релаксация хроматина происходит быстро в месте повреждения ДНК. [35] На одном из самых ранних этапов стресс-активируемая протеинкиназа, c-Jun N-терминальная киназа (JNK) , фосфорилирует SIRT6 на серине 10 в ответ на двухцепочечные разрывы или другие повреждения ДНК. [36] Эта посттрансляционная модификация облегчает мобилизацию SIRT6 к местам повреждения ДНК и необходима для эффективного привлечения поли (АДФ-рибоза) полимеразы 1 (PARP1) к местам разрыва ДНК и для эффективного восстановления DSB. [36] Белок PARP1 начинает появляться в местах повреждения ДНК менее чем за секунду, с полумаксимальным накоплением в течение 1,6 секунды после возникновения повреждения. [37] Затем ремоделер хроматина Alc1 быстро прикрепляется к продукту действия PARP1, цепи поли-АДФ-рибозы, и Alc1 завершает прибытие к повреждению ДНК в течение 10 секунд с момента возникновения повреждения. [35] Около половины максимальной релаксации хроматина, предположительно из-за действия Alc1, происходит за 10 секунд. [35] Это затем позволяет рекрутировать фермент репарации ДНК MRE11 , чтобы инициировать репарацию ДНК, в течение 13 секунд. [37]
γH2AX, фосфорилированная форма H2AX, также участвует в ранних этапах, ведущих к деконденсации хроматина после двухцепочечных разрывов ДНК. Гистоновый вариант H2AX составляет около 10% гистонов H2A в хроматине человека. [38] γH2AX (H2AX, фосфорилированный по серину 139) можно обнаружить уже через 20 секунд после облучения клеток (с образованием двухцепочечного разрыва ДНК), а половина максимального накопления γH2AX происходит через одну минуту. [38] Степень хроматина с фосфорилированным γH2AX составляет около двух миллионов пар оснований в месте двухцепочечного разрыва ДНК. [38] Сам по себе γH2AX не вызывает деконденсации хроматина, но в течение 30 секунд после облучения можно обнаружить белок RNF8 в ассоциации с γH2AX. [39] RNF8 опосредует обширную деконденсацию хроматина посредством его последующего взаимодействия с CHD4 , [40] компонентом комплекса ремоделирования нуклеосом и деацетилазы NuRD .
После релаксации, вызванной повреждением ДНК, и последующей репарации ДНК, хроматин восстанавливается до состояния уплотнения, близкого к уровню до повреждения, примерно через 20 минут. [35]
У позвоночных места, в которых происходит рекомбинация, определяются местами связывания PRDM9 , белка, который распознает определенный мотив последовательности с помощью своего массива цинковых пальцев. [41] В этих местах другой белок, SPO11, катализирует инициирующие рекомбинацию двухцепочечные разрывы (DSB), подмножество которых восстанавливается путем рекомбинации с гомологичной хромосомой. PRDM9 откладывает метки метилирования гистонов H3K4me3 и H3K36me3 в местах, с которыми он связывается, и эта активность метилтрансферазы необходима для его роли в позиционировании DSB. После их формирования сайты DSB обрабатываются путем резекции, в результате чего образуется одноцепочечная ДНК (ssDNA), которая становится декорированной DMC1. От середины зиготены до ранней пахитены, как часть процесса рекомбинационной репарации, DMC1 диссоциирует от одноцепочечной ДНК, и количество уменьшается до тех пор, пока все разрывы (за исключением тех, что находятся на хромосомах XY) не будут репарированы в поздней пахитене. В этом процессе участвуют несколько других белков, включая ZCWPW1, [42] первый белок, напрямую позиционируемый двойными гистоновыми метками PRDM9. ZCWPW1 важен для гомологичной репарации DSB, а не для позиционирования.
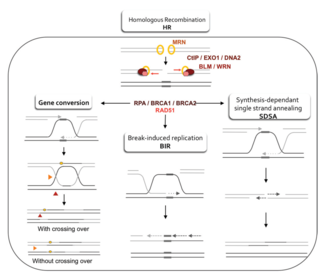
Две основные модели того, как гомологичная рекомбинация восстанавливает двухцепочечные разрывы в ДНК, — это путь репарации двухцепочечных разрывов (DSBR) (иногда называемый моделью двойного соединения Холлидея ) и путь синтез-зависимого отжига цепей (SDSA). [43] Эти два пути похожи на своих первых нескольких этапах. После того, как происходит двухцепочечный разрыв, комплекс MRX ( комплекс MRN у людей) связывается с ДНК по обе стороны от разрыва. Затем происходит резекция, при которой ДНК вокруг 5'-концов разрыва разрезается. Это происходит в два отдельных этапа: сначала комплекс MRX привлекает белок Sae2, и эти два белка обрезают 5'-концы по обе стороны от разрыва, чтобы создать короткие 3'-выступы одноцепочечной ДНК; на втором этапе резекция 5'→3' продолжается геликазой Sgs1 и нуклеазами Exo1 и Dna2 . Как геликаза , Sgs1 «распаковывает» двухцепочечную ДНК, в то время как нуклеазная активность Exo1 и Dna2 позволяет им разрезать одноцепочечную ДНК, продуцируемую Sgs1. [31]
Затем белок RPA , имеющий высокое сродство к одноцепочечной ДНК, связывает 3'-выступы. [44] С помощью нескольких других белков, которые опосредуют этот процесс, белок Rad51 (и Dmc1 в мейозе) затем образует нить нуклеиновой кислоты и белка на одинарной цепи ДНК, покрытой RPA. Затем эта нить нуклеопротеина начинает искать последовательности ДНК, похожие на последовательности 3'-выступа. После обнаружения такой последовательности одноцепочечная нить нуклеопротеина перемещается (вторгается) в подобный или идентичный дуплекс ДНК-реципиента в процессе, называемом вторжением нити . В клетках, которые делятся посредством митоза, дуплекс ДНК-реципиента, как правило, является сестринской хроматидом, которая идентична поврежденной молекуле ДНК и обеспечивает шаблон для восстановления. Однако в мейозе ДНК-реципиент, как правило, происходит из похожей, но не обязательно идентичной гомологичной хромосомы. [43] Петля смещения ( D-петля ) образуется во время вторжения нити между вторгающейся 3'-выступающей нитью и гомологичной хромосомой. После вторжения нити ДНК-полимераза удлиняет конец вторгающейся 3'-нити, синтезируя новую ДНК. Это изменяет D-петлю на крестообразную структуру, известную как соединение Холлидея . После этого на вторгающейся нити (т. е. на одном из исходных 3'-выступов) происходит еще больший синтез ДНК, эффективно восстанавливая нить на гомологичной хромосоме, которая была смещена во время вторжения нити. [43]

После стадий резекции, вторжения в нить и синтеза ДНК пути DSBR и SDSA становятся отдельными. [43] Путь DSBR уникален тем, что второй 3'-выступ (который не участвовал в вторжении в нить) также образует соединение Холлидея с гомологичной хромосомой. Двойные соединения Холлидея затем преобразуются в продукты рекомбинации с помощью никирующих эндонуклеаз , типа эндонуклеазы рестрикции , которая разрезает только одну цепь ДНК. Путь DSBR обычно приводит к кроссинговеру, хотя иногда он может приводить к продуктам, не являющимся кроссинговерными; способность разорванной молекулы ДНК собирать последовательности из разделенных донорских локусов была показана в митотических почкующихся дрожжах с использованием плазмид или эндонуклеазной индукции хромосомных событий. [45] [46] Из-за этой тенденции к хромосомному кроссинговеру путь DSBR является вероятной моделью того, как происходит кроссинговерная гомологичная рекомбинация во время мейоза. [22]
Приведет ли рекомбинация в пути DSBR к хромосомному кроссинговеру, определяется тем, как разрезается или «разрешается» двойной стык Холлидея. Хромосомный кроссинговер произойдет, если один стык Холлидея будет разрезана на пересекающейся нити, а другой стык Холлидея будет разрезана на непересекающейся нити (на рисунке 5, вдоль горизонтальных фиолетовых наконечников стрелок на одном стыке Холлидея и вдоль вертикальных оранжевых наконечников стрелок на другом). В качестве альтернативы, если два стыка Холлидея будут разрезаны на пересекающихся нитях (вдоль горизонтальных фиолетовых наконечников стрелок на обоих стыках Холлидея на рисунке 5), то будут получены хромосомы без кроссинговера. [47]
Гомологичная рекомбинация через путь SDSA происходит в клетках, которые делятся через митоз и мейоз и приводит к некроссоверным продуктам. В этой модели вторгающаяся 3'-цепь удлиняется вдоль дуплекса ДНК-реципиента ДНК-полимеразой и высвобождается, когда соединение Холлидея между молекулами ДНК донора и реципиента скользит в процессе, называемом миграцией ветвей . Затем вновь синтезированный 3'-конец вторгающейся цепи может отжигаться с другим 3'-выступом в поврежденной хромосоме посредством комплементарного спаривания оснований. После отжига цепей иногда может оставаться небольшой лоскут ДНК. Любые такие лоскуты удаляются, и путь SDSA заканчивается повторным закрытием, также известным как лигирование , любых оставшихся одноцепочечных пробелов. [48]
Во время митоза основным путем гомологичной рекомбинации для восстановления двухцепочечных разрывов ДНК, по-видимому, является путь SDSA (а не путь DSBR). [49] Путь SDSA производит некроссоверные рекомбинанты (рисунок 5). Во время мейоза некроссоверные рекомбинанты также встречаются часто, и они, по-видимому, также возникают в основном по пути SDSA. [49] [50] События некроссоверной рекомбинации, происходящие во время мейоза, вероятно, отражают случаи восстановления двухцепочечных повреждений ДНК или других типов повреждений ДНК.

Путь одноцепочечного отжига (SSA) гомологичной рекомбинации восстанавливает двухцепочечные разрывы между двумя повторяющимися последовательностями . Путь SSA уникален тем, что он не требует отдельной похожей или идентичной молекулы ДНК, как пути DSBR или SDSA гомологичной рекомбинации. Вместо этого путь SSA требует только одного дуплекса ДНК и использует повторяющиеся последовательности в качестве идентичных последовательностей, которые необходимы гомологичной рекомбинации для восстановления. Путь относительно прост по своей концепции: после того, как две нити одного и того же дуплекса ДНК разрезаются вокруг места двухцепочечного разрыва, два полученных 3'-выступа затем выравниваются и отжигаются друг с другом, восстанавливая ДНК как непрерывный дуплекс. [48] [51]
По мере того как ДНК вокруг двухцепочечного разрыва разрезается, образующиеся одноцепочечные 3'-выступы покрываются белком RPA , который предотвращает слипание 3'-выступов. [52] Затем белок, называемый Rad52, связывает каждую из последовательностей повторов по обе стороны разрыва и выравнивает их, чтобы позволить двум комплементарным последовательностям повторов отжигаться. [52] После завершения отжига оставшиеся негомологичные створки 3'-выступов отрезаются набором нуклеаз, известных как Rad1/Rad10, которые переносятся к створкам белками Saw1 и Slx4 . [52] [53] Новый синтез ДНК заполняет любые пробелы, а лигирование восстанавливает дуплекс ДНК в виде двух непрерывных цепей. [54] Последовательность ДНК между повторами всегда теряется, как и один из двух повторов. Путь SSA считается мутагенным , поскольку он приводит к таким делециям генетического материала. [48]
Во время репликации ДНК , двухцепочечные разрывы иногда могут встречаться в репликационных вилках , когда ДНК-хеликаза расстегивает матричную цепь. Эти дефекты исправляются в пути репликации, вызванной разрывом (BIR) гомологичной рекомбинации. Точные молекулярные механизмы пути BIR остаются неясными. Три предложенных механизма имеют вторжение цепи в качестве начального шага, но они различаются тем, как они моделируют миграцию D-петли и более поздние фазы рекомбинации. [55]
Путь BIR также может помочь поддерживать длину теломер (участков ДНК на конце эукариотических хромосом) при отсутствии (или в сотрудничестве с) теломеразы . Без рабочих копий фермента теломеразы теломеры обычно укорачиваются с каждым циклом митоза, что в конечном итоге блокирует деление клеток и приводит к старению . В почкующихся дрожжевых клетках, где теломераза была инактивирована посредством мутаций, было замечено, что два типа «выживших» клеток избегают старения дольше, чем ожидалось, удлиняя свои теломеры посредством путей BIR. [55]
Поддержание длины теломер имеет решающее значение для иммортализации клеток , ключевой особенности рака. Большинство видов рака поддерживают теломеры путем повышения регуляции теломеразы. Однако в нескольких типах рака человека BIR-подобный путь помогает поддерживать некоторые опухоли, действуя как альтернативный механизм поддержания теломер. [56] Этот факт побудил ученых исследовать, могут ли такие основанные на рекомбинации механизмы поддержания теломер помешать противораковым препаратам, таким как ингибиторы теломеразы . [57]

Гомологичная рекомбинация является основным процессом репарации ДНК у бактерий. Она также важна для создания генетического разнообразия в бактериальных популяциях, хотя этот процесс существенно отличается от мейотической рекомбинации, которая восстанавливает повреждения ДНК и обеспечивает разнообразие в эукариотических геномах . Гомологичная рекомбинация была наиболее изучена и лучше всего понята для Escherichia coli . [59] Двухцепочечные разрывы ДНК у бактерий восстанавливаются путем гомологичной рекомбинации RecBCD . Считается, что разрывы, которые происходят только на одной из двух цепей ДНК, известные как одноцепочечные разрывы, восстанавливаются путем RecF. [60] Оба пути RecBCD и RecF включают серию реакций, известных как миграция ветвей , в которой отдельные цепи ДНК обмениваются между двумя перекрещенными молекулами дуплексной ДНК, и разрешение , в которой эти две перекрещенные молекулы ДНК разрезаются и восстанавливаются до своего нормального двухцепочечного состояния.
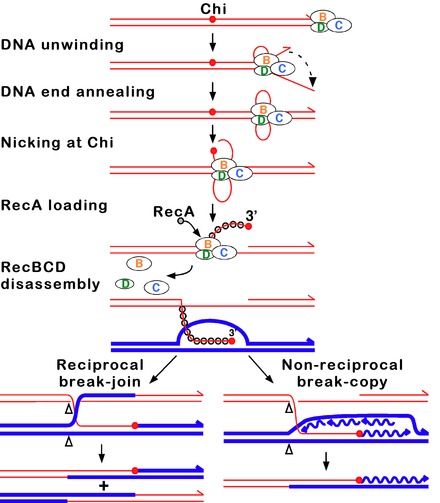

Путь RecBCD является основным путем рекомбинации, используемым во многих бактериях для восстановления двухцепочечных разрывов в ДНК, и эти белки встречаются у широкого спектра бактерий. [63] [64] [65] Эти двухцепочечные разрывы могут быть вызваны ультрафиолетовым светом и другим излучением , а также химическими мутагенами . Двухцепочечные разрывы могут также возникать при репликации ДНК через одноцепочечный надрез или зазор. Такая ситуация вызывает то, что известно как свернувшаяся репликационная вилка , и фиксируется несколькими путями гомологичной рекомбинации, включая путь RecBCD. [66]
В этом пути трехсубъединичный ферментный комплекс , называемый RecBCD, инициирует рекомбинацию, связываясь с тупым или почти тупым концом разрыва в двухцепочечной ДНК. После того, как RecBCD связывает конец ДНК, субъединицы RecB и RecD начинают расстегивать дуплекс ДНК посредством геликазной активности. Субъединица RecB также имеет домен нуклеазы , который разрезает одиночную цепь ДНК, которая появляется в процессе расстегивания. Это расстегивание продолжается до тех пор, пока RecBCD не встретит определенную нуклеотидную последовательность (5'-GCTGGTGG-3'), известную как сайт Chi . [65]
При столкновении с сайтом Chi активность фермента RecBCD резко меняется. [64] [61] [67] Раскручивание ДНК приостанавливается на несколько секунд, а затем возобновляется примерно с половиной начальной скорости. Это, вероятно, потому, что более медленная геликаза RecB раскручивает ДНК после Chi, а не более быстрая геликаза RecD, которая раскручивает ДНК до Chi. [68] [69] Распознавание сайта Chi также изменяет фермент RecBCD таким образом, что он разрезает цепь ДНК с помощью Chi и начинает загружать несколько белков RecA на одноцепочечную ДНК с вновь образованным 3'-концом. Полученная нуклеопротеиновая нить, покрытая RecA, затем ищет похожие последовательности ДНК на гомологичной хромосоме. Процесс поиска вызывает растяжение дуплекса ДНК, что усиливает распознавание гомологии (механизм, называемый конформационной корректурой [70] [71] [72] ). При обнаружении такой последовательности одноцепочечная нуклеопротеиновая нить перемещается в гомологичный дуплекс ДНК реципиента в процессе, называемом вторжением нити . [73] Вторгающийся 3'-выступ приводит к смещению одной из нитей дуплекса ДНК реципиента, образуя D-петлю. Если D-петля разрезана, еще один обмен нитями образует крестообразную структуру, называемую соединением Холлидея . [65] Разрешение соединения Холлидея некоторой комбинацией RuvABC или RecG может производить две рекомбинантные молекулы ДНК с реципрокными генетическими типами, если две взаимодействующие молекулы ДНК различаются генетически. Альтернативно, вторгающийся 3'-конец около Chi может инициировать синтез ДНК и образовывать репликативную вилку. Этот тип разрешения производит только один тип рекомбинанта (нереципрокный).
Бактерии, по-видимому, используют путь RecF гомологичной рекомбинации для восстановления одноцепочечных разрывов в ДНК. Когда путь RecBCD инактивируется мутациями, а дополнительные мутации инактивируют нуклеазы SbcCD и ExoI, путь RecF также может восстанавливать двухцепочечные разрывы ДНК. [74] В пути RecF геликаза RecQ раскручивает ДНК, а нуклеаза RecJ разрушает цепь с 5'-концом, оставляя цепь с 3'-концом нетронутой. Белок RecA связывается с этой цепью и либо поддерживается белками RecF, RecO и RecR, либо стабилизируется ими. Затем нить нуклеопротеина RecA ищет гомологичную ДНК и обменивается местами с идентичной или почти идентичной цепью в гомологичной ДНК.
Хотя белки и конкретные механизмы, задействованные в их начальных фазах, различаются, эти два пути схожи в том, что им обоим требуется одноцепочечная ДНК с 3'-концом и белок RecA для вторжения в нить. Пути также схожи в своих фазах миграции ветвей , в которой соединение Холлидея скользит в одном направлении, и разрешения , в котором соединения Холлидея расщепляются ферментами. [75] [76] Альтернативный, невзаимный тип разрешения также может происходить по любому из путей.
Сразу после внедрения нити соединение Холлидея перемещается вдоль связанной ДНК во время процесса миграции ветвей. Именно в этом движении соединения Холлидея происходит обмен парами оснований между двумя гомологичными дуплексами ДНК. Чтобы катализировать миграцию ветвей, белок RuvA сначала распознает соединение Холлидея и связывается с ним, а затем привлекает белок RuvB для формирования комплекса RuvAB. Два набора белка RuvB, каждый из которых образует кольцевую АТФазу , загружаются на противоположные стороны соединения Холлидея, где они действуют как двойные насосы, обеспечивающие силу для миграции ветвей. Между этими двумя кольцами RuvB два набора белка RuvA собираются в центре соединения Холлидея таким образом, что ДНК в соединении оказывается зажатой между каждым набором RuvA. Нити обоих дуплексов ДНК — дуплексов «донора» и «реципиента» — раскручиваются на поверхности RuvA, поскольку они направляются белком от одного дуплекса к другому. [77] [78]
В фазе разрешения рекомбинации любые соединения Холлидея, образованные процессом вторжения нитей, разрезаются, тем самым восстанавливая две отдельные молекулы ДНК. Это расщепление выполняется комплексом RuvAB, взаимодействующим с RuvC, которые вместе образуют комплекс RuvABC . RuvC — это эндонуклеаза , которая разрезает вырожденную последовательность 5'-(A/T)TT(G/C)-3'. Последовательность часто встречается в ДНК, примерно один раз на каждые 64 нуклеотида. [78] Перед разрезанием RuvC, вероятно, получает доступ к соединению Холлидея, вытесняя один из двух тетрамеров RuvA, покрывающих там ДНК. [77] Рекомбинация приводит к продуктам либо «сплайсинга», либо «заплатки», в зависимости от того, как RuvC расщепляет соединение Холлидея. [78] Продукты сплайсинга являются продуктами кроссинговера, в которых происходит перестройка генетического материала вокруг места рекомбинации. С другой стороны, продукты заплатки являются некроссоверными продуктами, в которых нет такой перестройки, а в продукте рекомбинации есть только «заплатка» гибридной ДНК. [79]
Гомологичная рекомбинация является важным методом интеграции донорской ДНК в геном организма-реципиента при горизонтальном переносе генов , процессе, при котором организм включает чужеродную ДНК из другого организма, не будучи потомком этого организма. Гомологичная рекомбинация требует, чтобы входящая ДНК была очень похожа на геном реципиента, и поэтому горизонтальный перенос генов обычно ограничивается похожими бактериями. [80] Исследования нескольких видов бактерий установили, что существует логлинейное снижение частоты рекомбинации с увеличением разницы в последовательности между ДНК хозяина и реципиента. [81] [82] [83]
При бактериальной конъюгации , когда ДНК передается между бактериями посредством прямого контакта клетка-клетка, гомологичная рекомбинация помогает интегрировать чужеродную ДНК в геном хозяина через путь RecBCD. Фермент RecBCD способствует рекомбинации после того, как ДНК преобразуется из одноцепочечной ДНК — в форме, в которой она изначально попадает в бактерию — в двухцепочечную ДНК во время репликации. Путь RecBCD также необходим для заключительной фазы трансдукции , типа горизонтального переноса генов, при котором ДНК передается от одной бактерии к другой вирусом . Чужеродная бактериальная ДНК иногда неправильно включается в капсидную головку вирусных частиц бактериофага , поскольку ДНК упаковывается в новые бактериофаги во время репликации вируса. Когда эти новые бактериофаги заражают другие бактерии, ДНК из предыдущей бактерии-хозяина вводится в нового бактериального хозяина в виде двухцепочечной ДНК. Затем фермент RecBCD включает эту двухцепочечную ДНК в геном нового бактериального хозяина. [65]
Естественная бактериальная трансформация включает перенос ДНК от донорной бактерии к реципиентной бактерии, где и донор, и реципиент обычно принадлежат к одному виду . Трансформация, в отличие от бактериальной конъюгации и трансдукции, зависит от многочисленных продуктов бактериальных генов, которые специфически взаимодействуют для выполнения этого процесса. [84] Таким образом, трансформация, очевидно, является бактериальной адаптацией к переносу ДНК. Для того чтобы бактерия связала, взяла и интегрировала донорскую ДНК в свою резидентную хромосому путем гомологичной рекомбинации, она должна сначала войти в особое физиологическое состояние, называемое компетентностью . Семейство генов RecA / Rad51 / DMC1 играет центральную роль в гомологичной рекомбинации во время бактериальной трансформации, как и во время эукариотического мейоза и митоза. Например, белок RecA необходим для трансформации у Bacillus subtilis и Streptococcus pneumoniae , [85] и экспрессия гена RecA индуцируется во время развития компетентности для трансформации у этих организмов.
В рамках процесса трансформации белок RecA взаимодействует с входящей одноцепочечной ДНК (ssDNA) для формирования нуклеофиламентов RecA/ssDNA, которые сканируют резидентную хромосому на наличие областей гомологии и переносят входящую одноцепочечную ДНК в соответствующую область, где происходит обмен цепями и гомологичная рекомбинация. [86] Таким образом, процесс гомологичной рекомбинации во время бактериальной трансформации имеет фундаментальное сходство с гомологичной рекомбинацией во время мейоза .
Гомологичная рекомбинация происходит в нескольких группах вирусов. В ДНК-вирусах, таких как вирус герпеса , рекомбинация происходит посредством механизма разрыва и повторного соединения, как у бактерий и эукариот. [87] Также имеются доказательства рекомбинации в некоторых РНК-вирусах , в частности, в вирусах одноцепочечной РНК с положительным смыслом, таких как ретровирусы , пикорнавирусы и коронавирусы . [88] Существуют разногласия по поводу того, происходит ли гомологичная рекомбинация в вирусах одноцепочечной РНК с отрицательным смыслом, таких как грипп . [89]
В РНК-вирусах гомологичная рекомбинация может быть как точной, так и неточной. В точном типе рекомбинации РНК-РНК нет никакой разницы между двумя родительскими последовательностями РНК и результирующей кроссоверной областью РНК. Из-за этого часто бывает трудно определить местоположение событий кроссинговера между двумя рекомбинирующими последовательностями РНК. В неточной гомологичной рекомбинации РНК область кроссинговера имеет некоторое отличие от родительских последовательностей РНК, вызванное добавлением, удалением или другой модификацией нуклеотидов. Уровень точности кроссинговера контролируется контекстом последовательности двух рекомбинирующих нитей РНК: последовательности, богатые аденином и урацилом, снижают точность кроссинговера. [88] [90]
Гомологичная рекомбинация важна для содействия вирусной эволюции . [88] [91] Например, если геномы двух вирусов с различными неблагоприятными мутациями подвергаются рекомбинации, то они могут быть способны регенерировать полностью функциональный геном. В качестве альтернативы, если два похожих вируса заразили одну и ту же клетку-хозяина, гомологичная рекомбинация может позволить этим двум вирусам обменять гены и тем самым развить более мощные вариации самих себя. [91]
Гомологичная рекомбинация — это предполагаемый механизм, посредством которого ДНК вируса герпеса человека-6 интегрируется в теломеры человека. [92]
Когда два или более вируса, каждый из которых содержит летальное геномное повреждение, заражают одну и ту же клетку-хозяина, геномы вирусов часто могут спариваться друг с другом и подвергаться гомологичной рекомбинационной репарации для получения жизнеспособного потомства. Этот процесс, известный как множественная реактивация, был изучен на нескольких бактериофагах , включая фаг T4 . [93] Ферменты, используемые в рекомбинационной репарации в фаге T4, функционально гомологичны ферментам, используемым в бактериальной и эукариотической рекомбинационной репарации. [94] В частности, в отношении гена, необходимого для реакции обмена цепями, ключевого шага в гомологичной рекомбинационной репарации, существует функциональная гомология от вирусов к людям (т. е. uvsX в фаге T4; recA в E. coli и других бактериях, а также rad51 и dmc1 в дрожжах и других эукариотах, включая людей). [95] Множественная реактивация также была продемонстрирована на многочисленных патогенных вирусах. [96]
Коронавирусы способны к генетической рекомбинации , когда в одной и той же инфицированной клетке присутствуют по крайней мере два вирусных генома . Рекомбинация РНК , по-видимому, является основной движущей силой в определении (1) генетической изменчивости внутри вида CoV, (2) способности вида CoV переходить от одного хозяина к другому и (3) нечасто, появления новых CoV. [97] Механизм рекомбинации в CoV, вероятно, включает переключение шаблонов во время репликации генома. [97] Рекомбинация в РНК-вирусах, по-видимому, является адаптацией для преодоления повреждения генома. [98]
Весь мотив связывания рецепторов пандемического SARS-CoV-2, по-видимому, был введен посредством рекомбинации из коронавирусов панголинов . [99] Такое событие рекомбинации могло быть критическим шагом в эволюции способности SARS-CoV-2 заражать людей. [99] События рекомбинации , вероятно, являются ключевыми шагами в эволюционном процессе, который приводит к появлению новых человеческих коронавирусов. [100]
Во время пандемии COVID-19 в 2020 году во многих геномных последовательностях австралийских изолятов SARS-CoV-2 были обнаружены делеции или мутации (29742G>A или 29742G>U; «G19A» или «G19U») в мотиве типа 3′ stem-loop II (s2m) коронавируса , мотиве РНК в 3'-нетранслируемой области вирусного генома, что позволяет предположить, что в s2m SARS-CoV-2 могли произойти события рекомбинации РНК. На основе вычислительного анализа 1319 последовательностей SARS-CoV-2 в Австралии с использованием алгоритма Recco (https://recco.bioinf.mpi-inf.mpg.de/) были предсказаны точки рекомбинации 29742G("G19"), 29744G("G21") и 29751G("G28"). [101]
Вспышка SARS-CoV-2 на круизном лайнере Diamond Princess, скорее всего, произошла либо от одного человека, инфицированного вариантом вируса, идентичным изолятам WIV04 из Уханя, либо одновременно с другим первичным случаем, инфицированным вирусом, содержащим мутацию 11083G > T. Анализ неравновесного сцепления подтвердил, что рекомбинация РНК с мутацией 11083G > T также способствовала увеличению мутаций среди вирусного потомства. Результаты показывают, что мутация 11083G > T SARS-CoV-2 распространилась во время карантина на борту судна и возникла посредством рекомбинации РНК de novo под давлением положительного отбора. Кроме того, у трех пациентов в этом круизе две мутации 29736G > T и 29751G > T («G13» и «G28») были также обнаружены в мотиве Coronavirus 3′ stem-loop II-like (s2m) , поскольку «G28» был предсказан как горячие точки рекомбинации у австралийских мутантов SARS-CoV-2. Хотя s2m считается мотивом РНК, высококонсервативным среди многих видов коронавирусов, этот результат также предполагает, что s2m SARS-CoV-2 скорее является горячей точкой рекомбинации/мутации РНК. [102]
Без надлежащей гомологичной рекомбинации хромосомы часто неправильно выравниваются для первой фазы деления клеток в мейозе . Это приводит к тому, что хромосомы не могут правильно разделиться в процессе, называемом нерасхождением . В свою очередь, нерасхождение может привести к тому, что сперма и яйцеклетки будут иметь слишком мало или слишком много хромосом. Синдром Дауна , который вызывается дополнительной копией хромосомы 21 , является одним из многих отклонений, которые являются результатом такого нарушения гомологичной рекомбинации в мейозе. [78] [103]
Дефициты гомологичной рекомбинации тесно связаны с образованием рака у людей. Например, каждое из заболеваний, связанных с раком , синдром Блума , синдром Вернера и синдром Ротмунда–Томсона, вызвано неисправными копиями генов геликазы RecQ , участвующих в регуляции гомологичной рекомбинации: BLM , WRN и RECQL4 соответственно. [104] В клетках пациентов с синдромом Блума, у которых отсутствует рабочая копия белка BLM, наблюдается повышенная скорость гомологичной рекомбинации. [105] Эксперименты на мышах с дефицитом BLM показали, что мутация приводит к раку из-за потери гетерозиготности, вызванной повышенной гомологичной рекомбинацией. [106] Потеря гетерозиготности относится к потере одной из двух версий — или аллелей — гена. Если один из потерянных аллелей помогает подавлять опухоли, как, например, ген белка ретинобластомы , то потеря гетерозиготности может привести к раку. [107] : 1236
Снижение скорости гомологичной рекомбинации приводит к неэффективной репарации ДНК, [107] : 310 , что также может привести к раку. [108] Это касается BRCA1 и BRCA2 , двух похожих генов-супрессоров опухолей , неисправность которых связана со значительным увеличением риска рака груди и яичников . Клетки, в которых отсутствуют BRCA1 и BRCA2, имеют пониженную скорость гомологичной рекомбинации и повышенную чувствительность к ионизирующему излучению , что позволяет предположить, что снижение гомологичной рекомбинации приводит к повышенной восприимчивости к раку. [108] Поскольку единственная известная функция BRCA2 — это помощь в инициировании гомологичной рекомбинации, исследователи предположили, что более детальное знание роли BRCA2 в гомологичной рекомбинации может стать ключом к пониманию причин рака груди и яичников. [108]
Опухоли с дефицитом гомологичной рекомбинации (включая дефекты BRCA) описываются как HRD-положительные. [109]

Хотя пути могут механически различаться, способность организмов выполнять гомологичную рекомбинацию универсально сохраняется во всех доменах жизни. [110] Основываясь на сходстве их аминокислотных последовательностей, гомологи ряда белков могут быть обнаружены во многих доменах жизни, что указывает на то, что они эволюционировали давно и с тех пор разошлись с общими предковыми белками. [110]
Члены семейства рекомбиназы RecA обнаружены почти во всех организмах: RecA — у бактерий, Rad51 и DMC1 — у эукариот, RadA — у архей , а UvsX — у фага T4 . [111]
Родственные одноцепочечные связывающие белки, которые важны для гомологичной рекомбинации и многих других процессов, также обнаружены во всех доменах жизни. [112]
Rad54, Mre11 , Rad50 и ряд других белков также обнаружены как у архей, так и у эукариот. [110] [111] [113]
Белки семейства белков рекомбиназы RecA, как полагают, произошли от общей предковой рекомбиназы. [110] Семейство рекомбиназы RecA содержит белок RecA из бактерий , белки Rad51 и Dmc1 из эукариот и RadA из архей , а также паралоговые белки рекомбиназы. Исследования, моделирующие эволюционные отношения между белками Rad51, Dmc1 и RadA, указывают на то, что они являются монофилетическими или что они имеют общего молекулярного предка. [110] В этом семействе белков Rad51 и Dmc1 сгруппированы вместе в отдельную кладу от RadA. Одной из причин объединения этих трех белков вместе является то, что все они обладают модифицированным мотивом спираль-поворот-спираль , который помогает белкам связываться с ДНК по направлению к их N-концам . [110] Древнее событие дупликации гена эукариотического RecA и последующая мутация были предложены в качестве вероятного источника современных генов RAD51 и DMC1. [110]
Белки обычно разделяют длинную консервативную область , известную как домен RecA/Rad51. Внутри этого домена белка есть два мотива последовательности , мотив Walker A и мотив Walker B. Мотивы Walker A и B позволяют членам семейства белков RecA/Rad51 участвовать в связывании АТФ и гидролизе АТФ . [110] [114]
Открытие Dmc1 у нескольких видов Giardia , одного из самых ранних простейших, отделившихся как эукариоты, предполагает, что мейотическая гомологичная рекомбинация — и, следовательно, сам мейоз — возникли очень рано в эволюции эукариот. [115] В дополнение к исследованиям Dmc1, исследования белка Spo11 предоставили информацию о происхождении мейотической рекомбинации. [116] Spo11, топоизомераза типа II , может инициировать гомологичную рекомбинацию в мейозе, делая целевые двухцепочечные разрывы в ДНК. [25] Филогенетические деревья, основанные на последовательности генов, похожих на SPO11 у животных, грибов, растений, простейших и архей, привели ученых к мысли, что версия Spo11, которая в настоящее время присутствует у эукариот, возникла у последнего общего предка эукариот и архей. [116]

Многие методы введения последовательностей ДНК в организмы для создания рекомбинантной ДНК и генетически модифицированных организмов используют процесс гомологичной рекомбинации. [117] Также называемый нацеливанием генов , этот метод особенно распространен в генетике дрожжей и мышей . Метод нацеливания генов у нокаутированных мышей использует эмбриональные стволовые клетки мыши для доставки искусственного генетического материала (в основном терапевтического интереса), который подавляет целевой ген мыши по принципу гомологичной рекомбинации. Таким образом, мышь выступает в качестве рабочей модели для понимания эффектов определенного гена млекопитающего. В знак признания их открытия того, как гомологичная рекомбинация может быть использована для введения генетических модификаций у мышей через эмбриональные стволовые клетки, Марио Капеччи , Мартин Эванс и Оливер Смитис были награждены Нобелевской премией по физиологии и медицине 2007 года . [118]
Достижения в технологиях генного таргетирования, которые захватывают механику гомологичной рекомбинации клеток, в настоящее время ведут к разработке новой волны более точных изогенных моделей человеческих заболеваний . Считается, что эти сконструированные модели человеческих клеток точнее отражают генетику человеческих заболеваний, чем их предшественники на основе мышиных моделей. Это во многом объясняется тем, что интересующие мутации вводятся в эндогенные гены, так же, как они происходят у реальных пациентов, и тем, что они основаны на человеческих геномах, а не на крысиных. Кроме того, определенные технологии позволяют вводить определенную мутацию, а не просто выключать ее, как это было со старыми технологиями генного таргетирования.
Белковая инженерия с гомологичной рекомбинацией разрабатывает химерные белки путем обмена фрагментами между двумя родительскими белками. Эти методы используют тот факт, что рекомбинация может вносить высокую степень разнообразия последовательностей , сохраняя при этом способность белка складываться в его третичную структуру или трехмерную форму. [119] Это контрастирует с другими методами белковой инженерии, такими как случайный точечный мутагенез , в котором вероятность сохранения функции белка экспоненциально снижается с увеличением замены аминокислот . [120] Химеры, полученные с помощью методов рекомбинации, способны сохранять свою способность к складыванию, поскольку их замененные родительские фрагменты структурно и эволюционно консервативны. Эти рекомбинируемые «строительные блоки» сохраняют структурно важные взаимодействия, такие как точки физического контакта между различными аминокислотами в структуре белка. Вычислительные методы, такие как SCHEMA и статистический анализ сопряжения, могут использоваться для идентификации структурных субъединиц, подходящих для рекомбинации. [121] [122] [123]
Методы, основанные на гомологичной рекомбинации, использовались для создания новых белков. [121] В исследовании, опубликованном в 2007 году, исследователи смогли создать химеры двух ферментов, участвующих в биосинтезе изопреноидов , разнообразного класса соединений, включая гормоны , зрительные пигменты и определенные феромоны . Химерные белки приобрели способность катализировать важную реакцию в биосинтезе изопреноидов — одном из самых разнообразных путей биосинтеза , обнаруженных в природе, — которая отсутствовала в родительских белках. [124] Белковая инженерия посредством рекомбинации также произвела химерные ферменты с новой функцией в членах группы белков, известных как семейство цитохромов P450 , [125] которое у людей участвует в детоксикации чужеродных соединений, таких как лекарства, пищевые добавки и консерванты. [22]
Клетки рака с гомологичной рекомбинацией (HRP) способны восстанавливать повреждения ДНК, вызванные химиотерапией, такой как цисплатин. Таким образом, рак HRP трудно поддается лечению. Исследования показывают, что гомологичную рекомбинацию можно нацелить с помощью ингибирования c-Abl. [126] [127] Раковые клетки с мутациями BRCA имеют дефицит гомологичной рекомбинации, и препараты для использования этих дефицитов были разработаны и успешно использовались в клинических испытаниях. [128] [129] Олапариб , ингибитор PARP1, уменьшил или остановил рост опухолей рака молочной железы , яичников и простаты, вызванных мутациями в генах BRCA1 или BRCA2 , которые необходимы для HR. Когда BRCA1 или BRCA2 отсутствуют, другие типы механизмов репарации ДНК должны компенсировать дефицит HR, такие как репарация с удалением оснований (BER) для остановившихся репликативных вилок или негомологичное соединение концов (NHEJ) для двухцепочечных разрывов. [128] Ингибируя BER в клетке с дефицитом HR, олапариб применяет концепцию синтетической летальности для специфического воздействия на раковые клетки. Хотя ингибиторы PARP1 представляют собой новый подход к терапии рака, исследователи предупредили, что они могут оказаться недостаточными для лечения метастатического рака на поздней стадии. [128] Раковые клетки могут стать устойчивыми к ингибитору PARP1, если они подвергаются делециям мутаций в BRCA2, что подрывает синтетическую летальность препарата за счет восстановления способности раковых клеток восстанавливать ДНК с помощью HR. [130]